本文将探讨了缺失值插补的不同方法,并比较了它们在复原数据真实分布方面的效果,处理插补是一个不确定性的问题,尤其是在样本量较小或数据复杂性高时的挑战,应选择能够适应数据分布变化并准确插补缺失值的方法。
我们假设存在一个潜在的分布P,从中得出观察值X。此外,还绘制了一个与X相同维数的0/1向量,我们称这个向量为M,实际观测到的数据向量X被M掩码为X。我们观测到联合向量(X,M)的n个独立同分布(i.i.d)副本。如果我们把它写成一个数据矩阵,它可能看起来像这样:
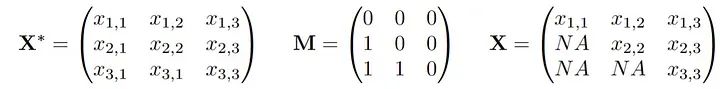
较小的x,m表示“观察到的”,而较大的值则表示随机数量。大家讨论的缺失机制就是对(X*,M)的关系或联合分布的假设:
完全随机缺失(MCAR):一个值丢失的概率就像抛硬币一样,与数据集中的任何变量无关。缺失值只是一件麻烦事。你可以忽略它们,只关注数据集中完全观察到的部分,这样就不会有偏差。在数学中,对于所有m和x:
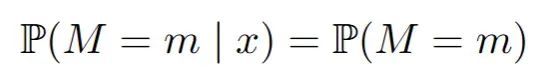
随机缺失(MAR):缺失的概率现在可以依赖于数据集中观察到的变量。一个典型的例子是两个变量,比如收入和年龄,其中年龄总是被观察到,但收入可能会因为年龄的某些值而丢失。这可能听起来很合理,但这里可能会变得复杂。在数学中,对于所有m和x:
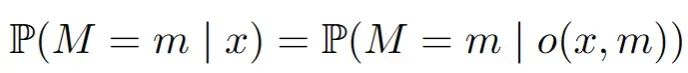
非随机缺失(MNAR):这里一切皆有可能,我们不能笼统地概括。但是最终我们需要学习给定一个模式m '中观测值的缺失值的条件分布,以便在另一个模式m中推算。
实现这一点的著名的方法称为链式方程多重插补(Multiple Imputation by Chained Equations, MICE):首先使用简单的插补方法填充值,例如均值插补。然后对于每一次迭代t,对每一个变量j,根据所有其他已插补的变量进行回归分析(这些变量已被插补)。然后将这些变量的值填入已学习的插补器中,用于所有未观察到的X_j。在R语言中,可以方便地使用mice包来实现。我这种方法在实际应用中效果非常好,MICE中重现某些实例的底层分布的能力非常惊人。我们下面会用一个非常简单的例子(只有一个变量缺失,因此我们可以手动编码),模拟MICE通常会迭代执行的过程,以更好地了解他的工作原理。
数据
我们将看一个非常简单但具有说明性的例子:考虑一个具有两个联合正态变量X_1, X_2的数据集。我们假设两个变量的方差为1,正相关系数为0.5。为了方便说明,我们假设X_1是收入的对数,X_2是年龄。另外我们设置收入X_1存在缺失机制,即当年龄“高”时,X_1往往会缺失。这就是我们设的:
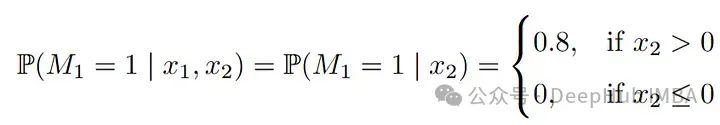
当X_2(年龄)较大(即大于0)时,X_1(收入)缺失的概率为0.8。由于我们假设X_2总是被观察到,这是一个教科书式的MAR示例,它有两种模式,一种是所有变量都被完全观察到(m1),另一种是(m2),其中X_1缺失。尽管这个例子很简单,但如果我们假设年龄越大,收入越高,那么从一种模式转换到另一种模式时,收入和年龄的分布就会发生明显的变化。在模式m2中,收入缺失,观察到的年龄和(未观察到的)收入的值都趋向于更高。
我们先看一下生成数据的代码:
library(MASS)
library(mice)
set.seed(10)
n<-3000
Xstar<-mvrnorm(n=n, mu=c(0,0), Sigma=matrix( c(1,0.7,0.7,1), nrow=2, byrow=T ))
colnames(Xstar) <-paste0("X",1:2)
## Introduce missing mechanisms
M<-matrix(0, ncol=ncol(Xstar), nrow=nrow(Xstar))
M[Xstar[,2] >0, 1]<-sample(c(0,1), size=sum(Xstar[,2] >0), replace=T, prob=c(1-0.8,0.8) )
## This gives rise to the observed dataset by masking X^* with M:
X<-Xstar
X[M==1] <-NA
## Plot the distribution shift
par(mfrow=c(2,1))
plot(Xstar[!is.na(X[,1]),1:2], xlab="", main="", ylab="", cex=0.8, col="darkblue", xlim=c(-4,4), ylim=c(-3,3))
plot(Xstar[is.na(X[,1]),1:2], xlab="", main="", ylab="", cex=0.8, col="darkblue", xlim=c(-4,4), ylim=c(-3,3))
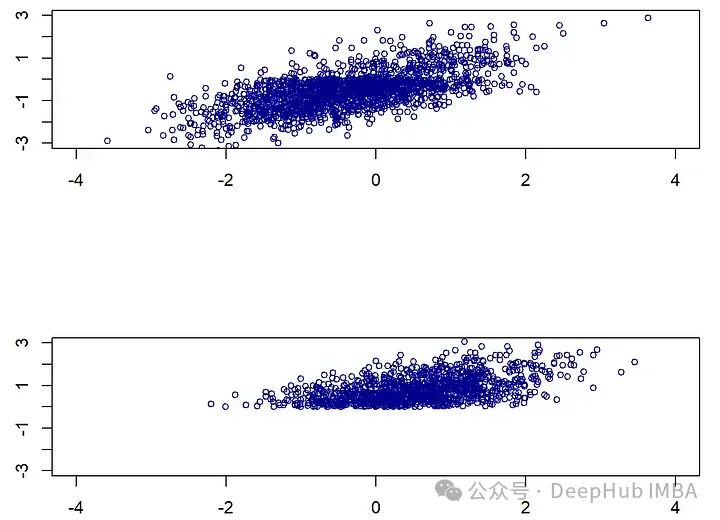
插补是一个分布预测问题
(通用的)插补的目标应该是尽可能地复制底层数据分布。为了说明这一点,考虑第一个例子,其中p=0,这样只有X_1缺失值现在将尝试使用著名的MICE方法来插补这个例子。由于只有X_1缺失,可以手动实现这一点。我们从均值插补开始,简单地计算在观测到的模式中X_1的均值,并将这个均值填补到NA的位置。我们还使用了更为复杂的回归插补:在观测到X_1的模式中,将X_1对X_2进行回归分析,然后对每个缺失的X_1观测值,我们插入回归的预测值。最后,对于高斯插补,我们从X_1对X_2的同样回归开始,但随后通过从高斯分布中抽取来插补每个缺失的X_1值。也就是说我们不是仅插补条件期望(即条件分布的中心),而是从这个分布中抽取。这导致了一种随机插补,起初可能有些违反直觉,但实际上会带来最好的结果:
## (0) Mean Imputation: This would correspond to "mean" in the mice R package ##
# 1. Estimate the mean
meanX<-mean(X[!is.na(X[,1]),1])
## 2. Impute
meanimp<-X
meanimp[is.na(X[,1]),1] <-meanX
## (1) Regression Imputation: This would correspond to "norm.predict" in the mice R package ##
# 1. Estimate Regression
lmodelX1X2<-lm(X1~X2, data=as.data.frame(X[!is.na(X[,1]),]) )
## 2. Impute
impnormpredict<-X
impnormpredict[is.na(X[,1]),1] <-predict(lmodelX1X2, newdata=as.data.frame(X[is.na(X[,1]),]) )
## (2) Gaussian Imputation: This would correspond to "norm.nob" in the mice R package ##
# 1. Estimate Regression
#lmodelX1X2<-lm(X1~X2, X=as.data.frame(X[!is.na(X[,1]),]) )
# (same as before)
## 2. Impute
impnorm<-X
meanx<-predict(lmodelX1X2, newdata=as.data.frame(X[is.na(X[,1]),]) )
var<-var(lmodelX1X2$residuals)
impnorm[is.na(X[,1]),1] <-rnorm(n=length(meanx), mean=meanx, sd=sqrt(var) )
## Plot the different imputations
par(mfrow=c(2,2))
plot(meanimp[!is.na(X[,1]),c("X2","X1")], main=paste("Mean Imputation"), cex=0.8, col="darkblue", cex.main=1.5)
points(meanimp[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
plot(impnormpredict[!is.na(X[,1]),c("X2","X1")], main=paste("Regression Imputation"), cex=0.8, col="darkblue", cex.main=1.5)
points(impnormpredict[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
plot(impnorm[!is.na(X[,1]),c("X2","X1")], main=paste("Gaussian Imputation"), col="darkblue", cex.main=1.5)
points(impnorm[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
#plot(Xstar[,c("X2","X1")], main="Truth", col="darkblue", cex.main=1.5)
plot(Xstar[!is.na(X[,1]),c("X2","X1")], main="Truth", col="darkblue", cex.main=1.5)
points(Xstar[is.na(X[,1]),c("X2","X1")], col="darkgreen", cex=0.8 )
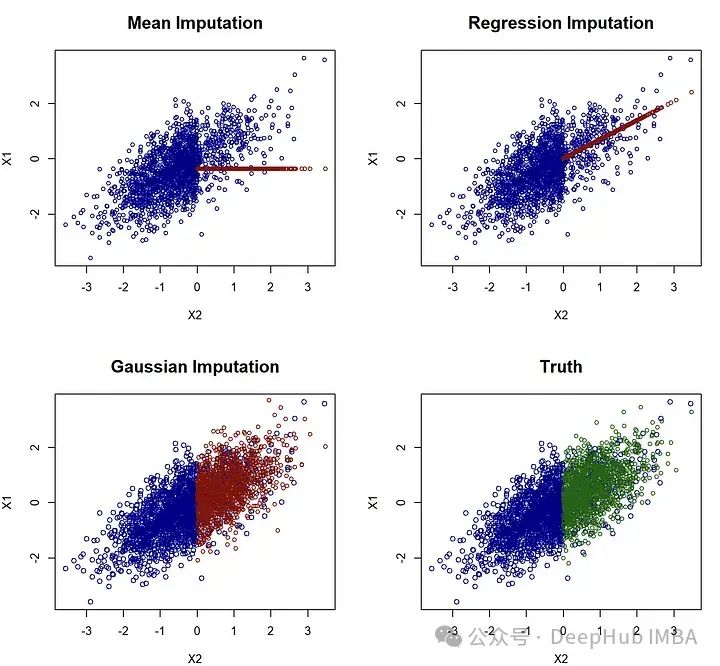
研究这张图可以立即发现,均值和回归插补并不好,因为它们完全未能重现原始数据分布。相比之下,高斯插补看起来相当不错,事实上,我认为很难将其与真实情况区分开来。这可能看起来只是一个技术概念,但这有其后果。想象一下,如果你得到了任何这些插补的数据集,现在你想要找到在将X_2对X_1进行回归时的回归系数(与我们用于插补的操作相反)。在这种情况下,真实值由beta=cov(X_1, X_2)/var(X_1)=0.7给出。
## Regressing X_2 onto X_1
## mean imputation estimate
lm(X2~X1, data=data.frame(meanimp))$coefficients["X1"]
## beta= 0.61
## regression imputation estimate
round(lm(X2~X1, data=data.frame(impnormpredict))$coefficients["X1"],2)
## beta= 0.90
## Gaussian imputation estimate
round(lm(X2~X1, data=data.frame(impnorm))$coefficients["X1"],2)
## beta= 0.71
## Truth imputation estimate
round(lm(X2~X1, data=data.frame(Xstar))$coefficients["X1"],2)
## beta= 0.71
高斯插补的结果非常接近0.7(0.71),更重要的是,它非常接近使用完整(未观测)数据得到的估计!而均值插补低估了beta值,回归插补则高估了beta值。回归插补因为条件均值插补人为地增强了变量之间的关系,这将导致在科学和(数据科学)实践中估计出的效应被过高估计!
回归插补可能看起来过于简单,但是在机器学习和其他领域中非常常用的插补方法正是这样工作的。例如,knn插补和随机森林插补(即missForest)。特别是随机森林插补在几篇基准测试论文中受到赞扬和推荐,且应用非常广泛。missForest是在观测数据上拟合一个随机森林,然后简单地通过条件均值进行插补,使用它的结果将与回归插补非常相似,从而导致变量之间关系的人为强化和估计的偏差!
如何评估插补方法?
上面我们已经说了应将插补视为一个分布预测的问题,那么这个分布预测的问题应该如何评估呢?
设想我们开发了一种新的插补方法,现在想要与已存在的方法如missForest、MICE或GAIN进行基准测试。这种情况下,我们人为地引入缺失值。然后将这个真实数据集与我们的插补结果进行比较。我们假设上面的回归插补是一种新方法,我们想要将其与均值和高斯插补进行比较。
即使在最有声望的会议中,也是通过计算均方根误差(RMSE)来完成的:
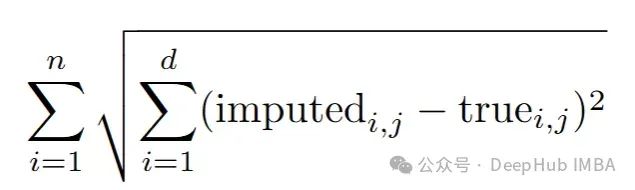
但是使用RMSE来评估我们的插补将偏向于那些插补条件均值的方法,如回归插补、knn插补和missForest。
而就像我们要说明的问题,由于插补本质上是一个分布预测问题,因此除了使用RMSE等统计指标之外,还应评估插补方法是否能够恢复数据的整体分布。这可以通过比较不同插补方法生成的数据分布的统计特性(如均值、方差、偏度等)或使用更复杂的分布相似性度量(如地球移动者距离或Kullback-Leibler散度)来实现。这里使用使用能量距离来衡量完全观测数据的分布与插补“分布”之间的差异
能量距离:能量距离是一种度量两个分布之间差异的统计量,它基于从两个分布中随机抽取样本对的距离。在插补的上下文中,它能有效地评估插补数据分布与原始数据分布之间的相似性。
通过使用能量距离等分布敏感的评估指标,我们可以更准确地评估插补方法是否能够恢复数据的原始统计特性,而不仅仅是平均水平上的接近性。这种方法特别适合于评估那些旨在捕捉数据复杂依赖性和分布特征的高级插补技术。
library(energy)
## Function to calculate the energy distance:
# impX is the imputed data set
# Xstar is the fully observed data set
## Calculating the energy distance using the eqdist.e function of the energy package
energycalc<-function(impX, Xstar){
# Note: eqdist.e calculates the energy statistics for a test, which is actually
# = n^2/(2n)*energydistance(impX,Xstar), but we we are only interested in relative values
round(eqdist.e( rbind(Xstar,impX), c(nrow(Xstar), nrow(impX)) ),2)
}
现在我们将这两个分数应用到这个研究项目中,并测试我们的回归imputation是否比其他两个更好:
par(mfrow=c(2,2))
## Same plots as before, but now with RMSE and energy distance
## added
plot(meanimp[!is.na(X[,1]),c("X2","X1")], main=paste("Mean Imputation", "\nRMSE", RMSEcalc(meanimp, Xstar), "\nEnergy", energycalc(meanimp, Xstar)), cex=0.8, col="darkblue", cex.main=1.5)
points(meanimp[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
plot(impnormpredict[!is.na(X[,1]),c("X2","X1")], main=paste("Regression Imputation","\nRMSE", RMSEcalc(impnormpredict, Xstar), "\nEnergy", energycalc(impnormpredict, Xstar)), cex=0.8, col="darkblue", cex.main=1.5)
points(impnormpredict[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
plot(impnorm[!is.na(X[,1]),c("X2","X1")], main=paste("Gaussian Imputation","\nRMSE", RMSEcalc(impnorm, Xstar), "\nEnergy", energycalc(impnorm, Xstar)), col="darkblue", cex.main=1.5)
points(impnorm[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
plot(Xstar[!is.na(X[,1]),c("X2","X1")], main="Truth", col="darkblue", cex.main=1.5)
points(Xstar[is.na(X[,1]),c("X2","X1")], col="darkgreen", cex=0.8 )
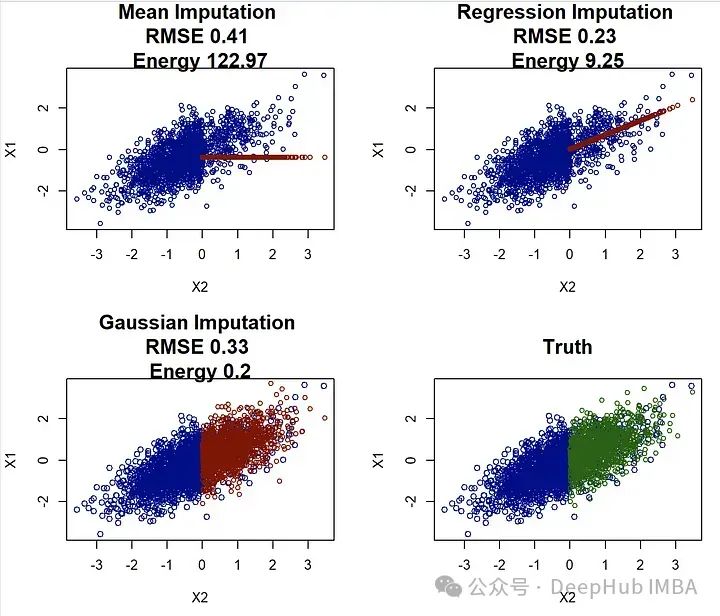
在使用均方根误差(RMSE)评估时,回归插补看起来表现非常好!它超过了均值插补和高斯插补。但是这与我们上面的分析相冲突,选择回归插补可能会导致高度偏见的结果。而(标度化的)能量距离正确地识别出高斯插补是最佳方法,这与视觉和更好的参数估计相一致。
所以,当评估插补方法时(当真实数据可用时),应避免使用像RMSE和MAE这样的度量。而应该被视为并作为分布预测问题来评估,应使用诸如能量距离之类的分布度量。将RMSE作为评估工具的过度使用对这一领域的研究有一些严重的影响。
当底层观察数据不可用时,评估问题变得更加困难。这篇论文[1]中,开发了一个分数,即使在这种情况下也能对插补方法进行排名!
library(mice)
source("Iscore.R")
methods<-c("mean", #mice-mean
"norm.predict", #mice-sample
"norm.nob") # Gaussian Imputation
## We first define functions that allow for imputation of the three methods:
imputationfuncs<-list()
imputationfuncs[["mean"]] <-function(X,m){
# 1. Estimate the mean
meanX<-mean(X[!is.na(X[,1]),1])
## 2. Impute
meanimp<-X
meanimp[is.na(X[,1]),1] <-meanX
res<-list()
for (lin1:m){
res[[l]] <-meanimp
}
return(res)
}
imputationfuncs[["norm.predict"]] <-function(X,m){
# 1. Estimate Regression
lmodelX1X2<-lm(X1~., data=as.data.frame(X[!is.na(X[,1]),]) )
## 2. Impute
impnormpredict<-X
impnormpredict[is.na(X[,1]),1] <-predict(lmodelX1X2, newdata=as.data.frame(X[is.na(X[,1]),]) )
res<-list()
for (lin1:m){
res[[l]] <-impnormpredict
}
return(res)
}
imputationfuncs[["norm.nob"]] <-function(X,m){
# 1. Estimate Regression
lmodelX1X2<-lm(X1~., data=as.data.frame(X[!is.na(X[,1]),]) )
## 2. Impute
impnorm<-X
meanx<-predict(lmodelX1X2, newdata=as.data.frame(X[is.na(X[,1]),]) )
var<-var(lmodelX1X2$residuals)
res<-list()
for (lin1:m){
impnorm[is.na(X[,1]),1] <-rnorm(n=length(meanx), mean=meanx, sd=sqrt(var) )
res[[l]] <-impnorm
}
return(res)
}
scoreslist<-Iscores_new(X,imputations=NULL, imputationfuncs=imputationfuncs, N=30)
scores<-do.call(cbind,lapply(scoreslist, function(x) x$score ))
names(scores)<-methods
scores[order(scores)]
# mean norm.predict norm.nob
# -0.7455304 -0.5702136 -0.4220387
无需看到缺失数据的值,分数也能够识别分布,特别是当数据有两个以上的维度时。
随机缺失比你想象的更奇怪
当阅读关于缺失值插补的文献时,人们容易认为在缺失数据机制为MAR(Missing At Random,随机缺失)的情况下问题已经解决,而所有的缺失问题都来自于是否可以假设为MAR。虽然在使用最大似然等标准程序时这可能是正确的,但如果想要找到一个好的(非参数的)插补方法,情况并非如此。
论文[1]讨论了在MAR下可能发生的复杂分布变化,当从完全观测的模式变化到一个想要插补的模式时,在观测变量中是否可能发生的分布变化呢?回到上面的例子,其中将X_1设为收入,将X_2设为年龄。在第一幅图中就能看到的,分布看起来相当不同。而条件分布 X_1 | X_2 保持不变!这在原则上允许识别正确的插补分布。
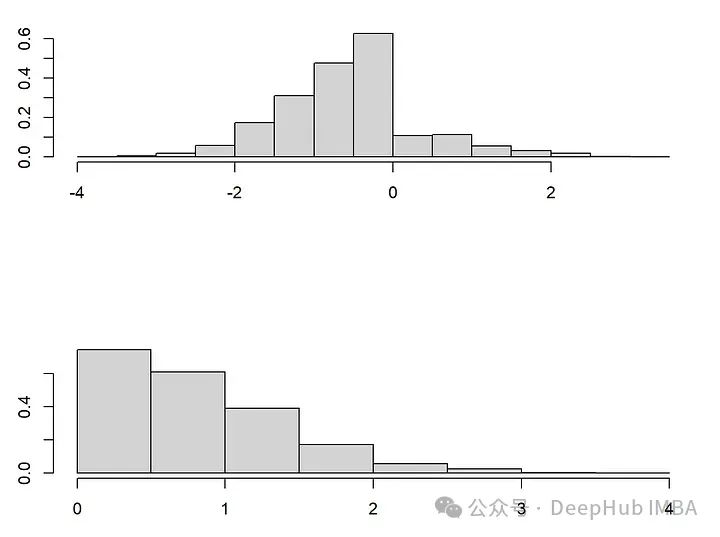
这一发现对于理解和处理MAR下的缺失数据具有重要意义。尽管数据可能看起来在全面观测和部分缺失时有不同的分布,通过关注条件分布的稳定性,可以更精确地插补缺失值。也就是说非参数插补技术能够适应数据的这种内在复杂性,而不仅仅是简单地应用参数方法或假设数据具有均匀分布。在实际应用中,这种深入分析揭示了为何简单的插补方法(如均值插补或条件均值插补)可能不足以捕捉数据的真实结构,尤其是在涉及复杂关联和动态分布时。
library(drf)
## mice-DRF ##
par(mfrow=c(2,2))
#Fit DRF
DRF<-drf(X=X[!is.na(X[,1]),2, drop=F], Y=X[!is.na(X[,1]),1, drop=F], num.trees=100)
impDRF<-X
# Predict weights for unobserved points
wx<-predict(DRF, newdata=X[is.na(X[,1]),2, drop=F] )$weights
impDRF[is.na(X[,1]),1] <-apply(wx,1,function(wxi) sample(X[!is.na(X[,1]),1, drop=F], size=1, replace=T, prob=wxi))
plot(impDRF[!is.na(X[,1]),c("X2","X1")], main=paste("DRF Imputation", "\nRMSE", RMSEcalc(impDRF, Xstar), "\nEnergy", energycalc(impDRF, Xstar)), cex=0.8, col="darkblue", cex.main=1.5)
points(impDRF[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
## mice-cart##
impcart<-X
impcart[is.na(X[,1]),1] <-mice.impute.cart(X[,1], ry=!is.na(X[,1]), X[,2, drop=F], wy=NULL)
plot(impDRF[!is.na(X[,1]),c("X2","X1")], main=paste("cart Imputation", "\nRMSE", RMSEcalc(impcart, Xstar), "\nEnergy", energycalc(impcart, Xstar)), cex=0.8, col="darkblue", cex.main=1.5)
points(impDRF[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
plot(impnorm[!is.na(X[,1]),c("X2","X1")], main=paste("Gaussian Imputation","\nRMSE", RMSEcalc(impnorm, Xstar), "\nEnergy", energycalc(impnorm, Xstar)), col="darkblue", cex.main=1.5)
points(impnorm[is.na(X[,1]),c("X2","X1")], col="darkred", cex=0.8 )
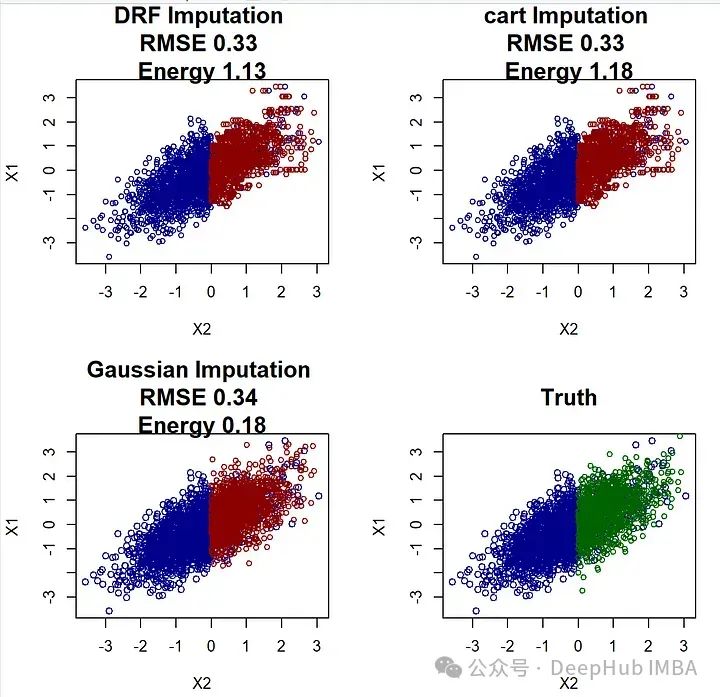
虽然mice-cart和mice-DRF都做得不错,但它们仍然不如高斯插补那么好。这本身并不令人惊讶,因为在这种情况下,高斯插补是理想的插补方法(因为 (X_1, X_2) 确实是高斯分布的)。而X_2中的分布变化可能可能导致mice-cart和mice-DRF在恢复3000个观测值的分布时遇到困难(这些方法通常非常有效)。
论文还讨论了一个类似的,但更极端的例子,涉及两个变量(X_1, X_2)。在这个例子中,分布变化更为显著,基于森林的方法相应地面临挑战:
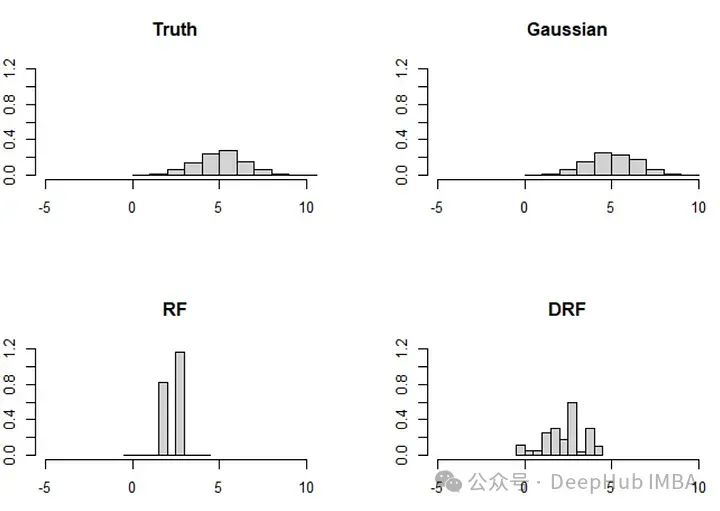
分布变化的影响:当底层数据的分布发生显著变化时,基于模型的插补方法(如基于决策树或随机森林的方法)可能难以准确地恢复数据的真实分布。这是因为这些模型依赖于数据中存在的模式和结构,而当这些模式和结构因为缺失数据而改变时,模型的效果可能会下降。
高斯插补的适用性:高斯插补假设数据遵循高斯分布,这使得它在处理本质上呈高斯分布的数据时非常有效。对于这种类型的数据,高斯插补能够更好地捕捉数据的整体统计特性,包括均值和协方差结构。
方法选择的重要性:选择正确的插补方法对于确保插补结果的质量至关重要。在面对复杂的数据和分布时,理解每种方法的优势和局限性可以帮助研究人员和实践者选择最适合他们数据特征的插补方法。
总结
缺失值确实是一个棘手的问题。,处理缺失值的最佳方式是尽量避免它们的出现,但是这几乎是不可能的,所以即使只考虑随机缺失(MAR),寻找插补方法的工作还远未结束。我们仍然缺乏一种能够(1)进行非参数分布预测和(2)适应在MAR下可能发生的分布变化的方法。有时也感觉人们将问题复杂化了,因为一些MICE方法表现得非常出色,可能已经足以解决许多缺失值问题。
有一些非常先进的机器学习方法,如GAIN及其变体,试图使用神经网络来插补数据。方法可能是正确的,因为它们遵循了正确的思路:插补在观察到的情况下缺失的条件分布。但是使用它们一段时间后,性能并不能让人满意,尤其是与MICE相比。
所以如果我遇到一个缺失值问题,可以首先尝试的是mice-cart或在论文中开发的新方法mice-DRF。它们重现数据的能力非常惊人。但是这些方法需要至少一个中等的样本大小,例如超过200或300。如果在少于200个观察的情况下,可以选择更简单的方法,如高斯插补。如果你想从这些方法中找到最好的,可以尝试使用论文中的评分规则来找到最好的方法。
最后别忘了这些方法都无法有效地处理插补不确定性!因为我们在这篇文章中只讨论了单一插补。正确的多重插补将需要考虑插补方法本身的不确定性,这通常使用贝叶斯方法完成,这可能又是一篇论文了。
最后:因为原论文都是使用R进行编写,我又对R不太熟悉,自己使用进行Python复现有几段出现了问题,所以就直接贴R的代码了,有兴趣的小伙伴请自行使用 Python重写。另外,GPT4的改写效果也不好,而copilot则直接提示无法改写,所以如果你有成功的的GPT4改写的Prompt也可以联系我,感谢。
最后本文引用的论文:
What Is a Good Imputation Under MAR Missingness? [1]
https://hal.science/hal-04521894
作者: Jeffrey Näf