1、.mrxs格式
MIRAX 可以以 JPEG、PNG 或 BMP 格式存储slide。 因为 JPEG 等格式不允许大图像,并且 JPEG 和 PNG 对图像的一部分的随机访问解码提供的支持很差,所以需要多个图像来对slide进行编码。 为避免有许多单独的文件,MIRAX 将这些图像打包成少量的数据文件。 索引文件为每条所需数据提供了数据文件的偏移量。
为了生成第 n + 1个level ,来自第 n 个level的每个图像被 2 下采样,然后连接成一个新图像,每个新图像 由4 个旧图像 (2 x 2)构成。 对每个level重复此过程,不是简单的图像重叠。 因此,在足够高的level上,单个图像可以包含一个或多个非整数宽度的嵌入重叠。
文件被分为三类:index file,data file 和slide position file
更多详细信息可见MIRAX format (openslide.org)
2、.mrxs文件的读取
①slider viewer2.6
1、下载方式见:SlideViewer | 3DHISTECH Ltd.
2、将.mrxs文件存储在相应文件夹下,打开SlideViewer.exe文件,即可找到相应的.mrxs文件
出现MRXS格式
在文件夹中也出现可读文件
3、选定slide后打开,界面如下
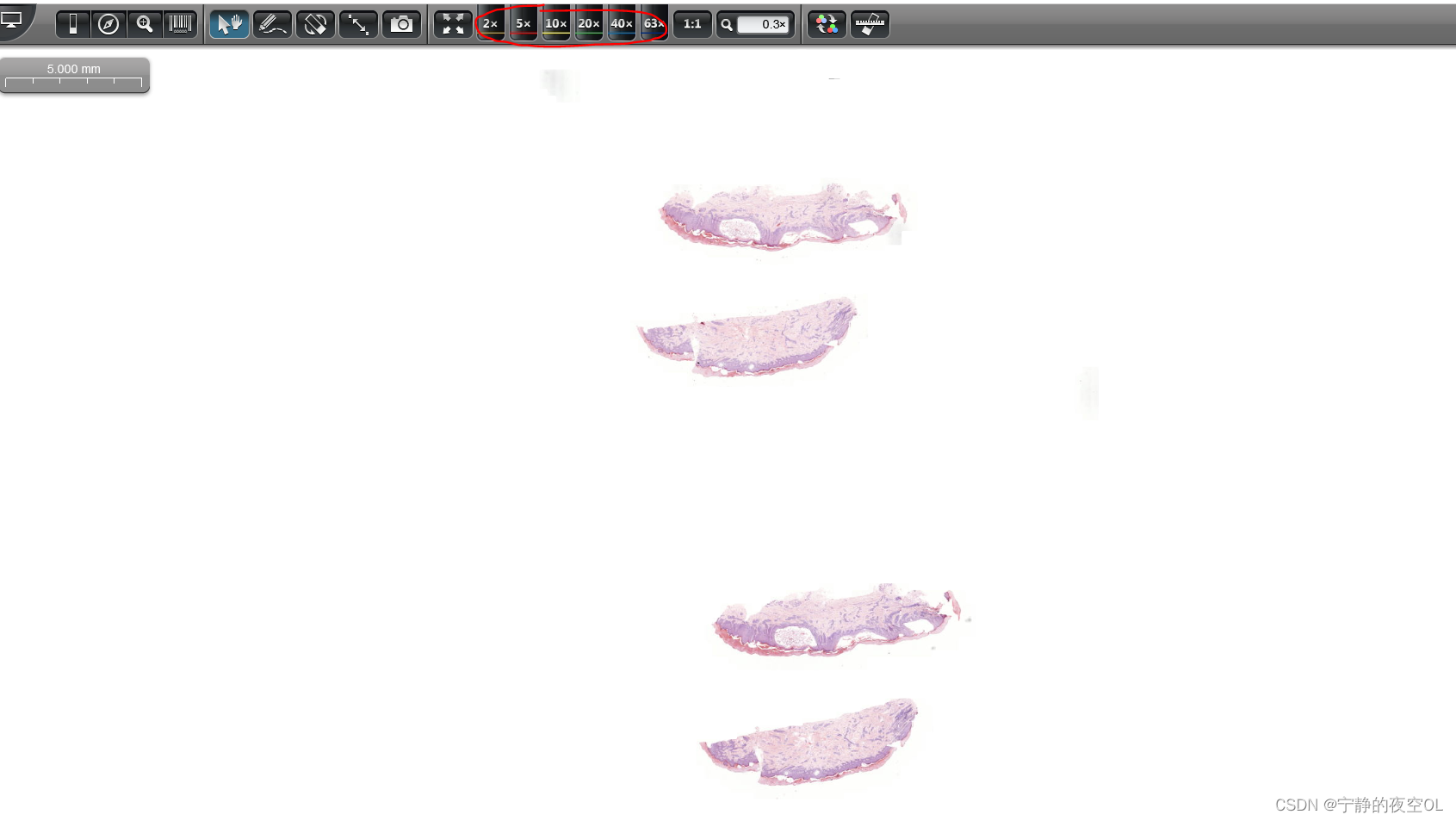
slide viewer界面
②openslide库
1、openslide库的安装
首先,在官网上下载二进制包Downloading OpenSlide
再,解压缩下载好的包,将bin和lib文件添加到系统的环境变量(变量名path2/path3),注意这个bin和lib文件在下载的注册表文件夹里,这个文件夹可以放在任何地方。

最后,进入对应的目录,激活相应的环境,用pip进行安装
conda activate 自己的环境名
pip install openslide
2、报错处理
报错内容:
Python 3.6.10 |Anaconda, Inc.| (default, May 7 2020, 19:46:08) [MSC v.1916 64 bit (AMD64)] on win32
Type "help", "copyright", "credits" or "license" for more information.
>>> import openslide
Traceback (most recent call last):
File "<stdin>", line 1, in <module>
File "d:\ProgramData\Anaconda3\envs\path\lib\site-packages\openslide\__init__.py", line 29, in <module>
from openslide import lowlevel
File "d:\ProgramData\Anaconda3\envs\path\lib\site-packages\openslide\lowlevel.py", line 44, in <module>
_lib = cdll.LoadLibrary('libopenslide-0.dll')
File "d:\ProgramData\Anaconda3\envs\path\lib\ctypes\__init__.py", line 426, in LoadLibrary
return self._dlltype(name)
File "d:\ProgramData\Anaconda3\envs\path\lib\ctypes\__init__.py", line 348, in __init__
self._handle = _dlopen(self._name, mode)
OSError: [WinError 126] 找不到指定的模块
这个错误是因为python搜索的bin库里没有找到相应的dll
处理方法:
找到openslide文件夹下面的lowlevel.py文件,在lowlevel.py下面增加如下代码:
import os
# openslide-bin-path为 openslide 的bin文件夹绝对路径。
os.environ['PATH'] = "openslide-bin-path" + ";" + os.environ['PATH']
# 例如:
os.environ['PATH'] = "D:\\openslide-win64-20171122\\bin" + ";" + os.environ['PATH']
再次运行问题解决。
3、openslide库的应用
(1)import库
import openslide
(2)读取图片
slide = openslide.OpenSlide(r"D:\xxxx\xxxxx.mrxs")
(3)关闭图像
slide.close()
(4)level_count——slide的level数。level从0(最高分辨率)到level_count - 1(最低分辨率)编号。
level_count = slide.level_count
print ('level_count = ',level_count)
代码返回为10,可能应该是slide的层数。
(5)dimensions(width, height)在0级别下,也就是最高分辨率的情况下slide的宽和高(元组)
[m,n] = slide.dimensions #得出高倍下的(宽,高)
print (m,n)
output:
110290 242930
(6)level_dimensions[k]得到(width, height)元组,k下级别k,是指在k水平下的下面举例就知道k的意思,每张全扫描最高级别是0,也就是最高分辨率,这个分辨率在不同的全扫描图片中是不一样的,有的第0层是40倍,第二层是10倍,而有的第0层是20倍,第二层是10倍,k指对应的层数。
level_downsamples 每一个级别K的对应的下采样因子,下采样因子应该对应一个倍率
#级别k,且k必须是整数,下采样因子和k有关
for i in range(level_count):
[m,n] = slide.level_dimensions[i] # 每一个级别对应的长和宽
slide_level_downsamples = slide.level_downsamples[i] # 下采样因子对应一个倍率
print (f"k={i}时的长和宽{m,n}和下采样倍率{slide_level_downsamples}")
slide_downsamples = slide.get_best_level_for_downsample(2.0) # 选定倍率返回下采样级别
print (slide_downsamples)
output:
k=0时的长和宽(110290, 242930)和下采样倍率1.0
k=1时的长和宽(55145, 121465)和下采样倍率2.0
k=2时的长和宽(27572, 60732)和下采样倍率4.0
k=3时的长和宽(13786, 30366)和下采样倍率8.0
k=4时的长和宽(6893, 15183)和下采样倍率16.0
k=5时的长和宽(3446, 7591)和下采样倍率32.0
k=6时的长和宽(1723, 3795)和下采样倍率64.0
k=7时的长和宽(861, 1897)和下采样倍率128.0
k=8时的长和宽(430, 948)和下采样倍率256.0
k=9时的长和宽(215, 474)和下采样倍率512.0
1
(7)read_region(location, level, size) 返回一个RGBA图像,包含指定区域的内容。location指0级别下左上角位置的坐标,元组,level指级别,整数,size是(width, height)是元组
tile = numpy.array(slide.read_region((0,0),6, (1528,3432)))
plt.figure()
plt.imshow(tile)
pylab.show()
#上述代码可以得到左上角坐标(0,0),6级别下,大小是(1528,3432)的图
(8)get_thumbnail(size) 返回一个缩略图的RGB图像,size为(width,height)元组
slide_thumbnail = slide.get_thumbnail((1528,3432))
tile = numpy.array(slide_thumbnail)
# scipy.misc.imsave('/home/xhj/PycharmProjects/openslide-experiment/save/thumbnail.jpg', tile)
plt.imshow(slide_thumbnail)
plt.imshow(tile)
pylab.show()
版权归原作者 宁静的夜空OL 所有, 如有侵权,请联系我们删除。