小罗碎碎念
本期推文主题
这一期推文是病理AI基础模型UNI的详细介绍,原文如下。下期推文会介绍如何使用这个模型,为了你能看懂下期的推文,强烈建议你好好看看今天这期推文。

看完这篇推文以后,你大概就能清楚这个模型对自己的数据有没有效果了。很显然,你看它用什么数据训练的,自然就知道自己的数据能否匹配。当然了,如果你也想做大模型的话,也可以参考这篇文章的思路,不过友情提醒,你要先看看自己能不能搞到32块80GB的A100,以及数块3090。
我是罗小罗同学,下期推文见!!
文献概述
一作&通讯
角色姓名单位(中文)第一作者Richard J. Chen哈佛医学院布里格姆和妇女医院病理科第一作者Tong Ding哈佛医学院工程与应用科学学院第一作者Ming Y. Lu哈佛医学院和麻省理工学院癌症项目通讯作者Faisal Mahmood哈佛医学院布里格姆和妇女医院病理科
这篇文章介绍了一个名为UNI的新型通用自监督模型,它在计算病理学领域通过大规模预训练显著提高了组织图像分析的性能,并在多种临床任务上展现了卓越的泛化能力。
- 研究背景:计算病理学在评估组织图像方面至关重要,需要从全切片图像(Whole-Slide Images, WSIs)中客观地表征组织病理学实体。WSIs的高分辨率和形态特征的变异性带来了显著挑战,使得大规模注释数据变得复杂,限制了高性能应用的发展。
- 研究目的:为了解决这些挑战,研究者们提出了使用预训练图像编码器的方法,通过从自然图像数据集的迁移学习或在公开可用的组织病理学数据集上的自监督学习来实现。然而,这些方法在不同组织类型和规模上的开发和评估还不够广泛。
- 研究方法:文章介绍了UNI,这是一个用于病理学的通用自监督模型,使用超过1亿张图像(超过77 TB数据)从超过10万个诊断H&E染色的WSIs中预训练,涵盖了20种主要组织类型。模型在34个代表性的CPath任务上进行了评估,这些任务在诊断难度上有所不同。
- 研究结果:UNI在多个任务中表现出色,除了超越了之前的最佳模型,还展示了CPath中的新建模能力,例如分辨率无关的组织分类、使用少量样本类原型的切片分类,以及在OncoTree分类系统中对多达108种癌症类型的疾病亚型泛化分类。
- 模型评估:研究者们还评估了UNI在不同数据规模下的预训练效果,以及不同ViT(Vision Transformer)架构大小对模型性能的影响。通过与MoCoV3等其他自监督学习算法的比较,研究者们发现DINOv2算法在大规模数据集上的表现更好。
- 临床应用:文章还讨论了UNI在临床病理学中的潜在应用,包括肿瘤检测、亚型分类、分级和分期,以及分子改变预测等。研究者们强调了UNI在处理罕见疾病和诊断复杂性较高的任务中的优势。
- 数据集和实验设置:研究者们使用了大规模、分层的癌症分类任务来评估UNI的泛化能力,这些任务遵循OncoTree癌症分类系统。他们还评估了不同数据多样性和预训练长度对模型性能的影响。
- 结论:UNI作为一个通用的自监督视觉编码器,展示了在多种机器学习设置中的多功能性,包括ROI级别的分类、分割和图像检索,以及切片级别的弱监督学习。研究者们认为UNI有潜力成为进一步发展解剖病理学中人工智能模型的基础模型。
文章还详细讨论了数据集的构建、模型的架构和预训练协议、评估设置、比较和基线、弱监督切片分类、线性和K-最近邻探针评估、ROI检索、ROI级别的细胞类型分割、少样本ROI分类和原型学习等方法和技术细节。
一、引言
病理学的临床实践中涉及执行大量任务:从肿瘤检测和亚型分类到分级和分期,鉴于存在成千上万的可能诊断,病理学家必须能够解决极其多样化的难题,并且通常是同时进行的1-4。当代计算病理学(CPath)通过实现分子改变的预测5,6、预后评估7-9以及治疗反应的预测10等应用,进一步扩展了这一范围,以及其他多项应用11-14。
由于收集病理学家注释、为单一疾病构建大型组织学收藏以及获取罕见疾病数据的挑战,从零开始训练模型的实际局限性较大。这些因素导致在CPath中依赖于迁移学习技术,该技术已被证明在诸如转移检测15、突变预测16,17、前列腺癌分级18和预后预测9,19,20等任务中是有效的。
自监督(或预训练)模型的迁移学习、泛化和扩展能力取决于训练数据的大小和多样性21-23。在一般的计算机视觉领域,许多基本自监督模型的发展与评估24-27基于ImageNet大型视觉识别挑战28,29和其他大型数据集30-32。这些模型也被称为“基础模型”,因为它们在大量数据上预训练后,能够适应广泛的下游任务33,34。
在CPath中,癌症基因组图谱(TCGA;约29,000张福尔马林固定石蜡包埋和冷冻的H&E全切片图像(WSIs),32种癌症类型)35同样作为许多自监督模型的基础36-46,以及其他组织学数据集47-53,许多先前的工作在为临床病理学任务学习有意义的组织学表征方面取得了重大进展37,38,54-66。然而,当前CPath的预训练模型仍受限于预训练数据的大小和多样性,考虑到TCGA主要包含原发性癌症组织学切片,以及跨多样组织类型泛化性能评估的局限性,许多泛癌分析和流行的临床任务也基于TCGA的注释组织区域(ROIs)和切片6,9,16,17,61,67-74。解决这些限制对于在CPath中进一步发展能广泛适用于实际临床环境的基础模型至关重要。
在这项工作中,作者基于先前的工作,引入了一个通用的、用于病理学的自监督视觉编码器UNI,这是一个在“Mass-100K”上预训练的大型视觉变换器(ViT-Large或ViT-L)75。
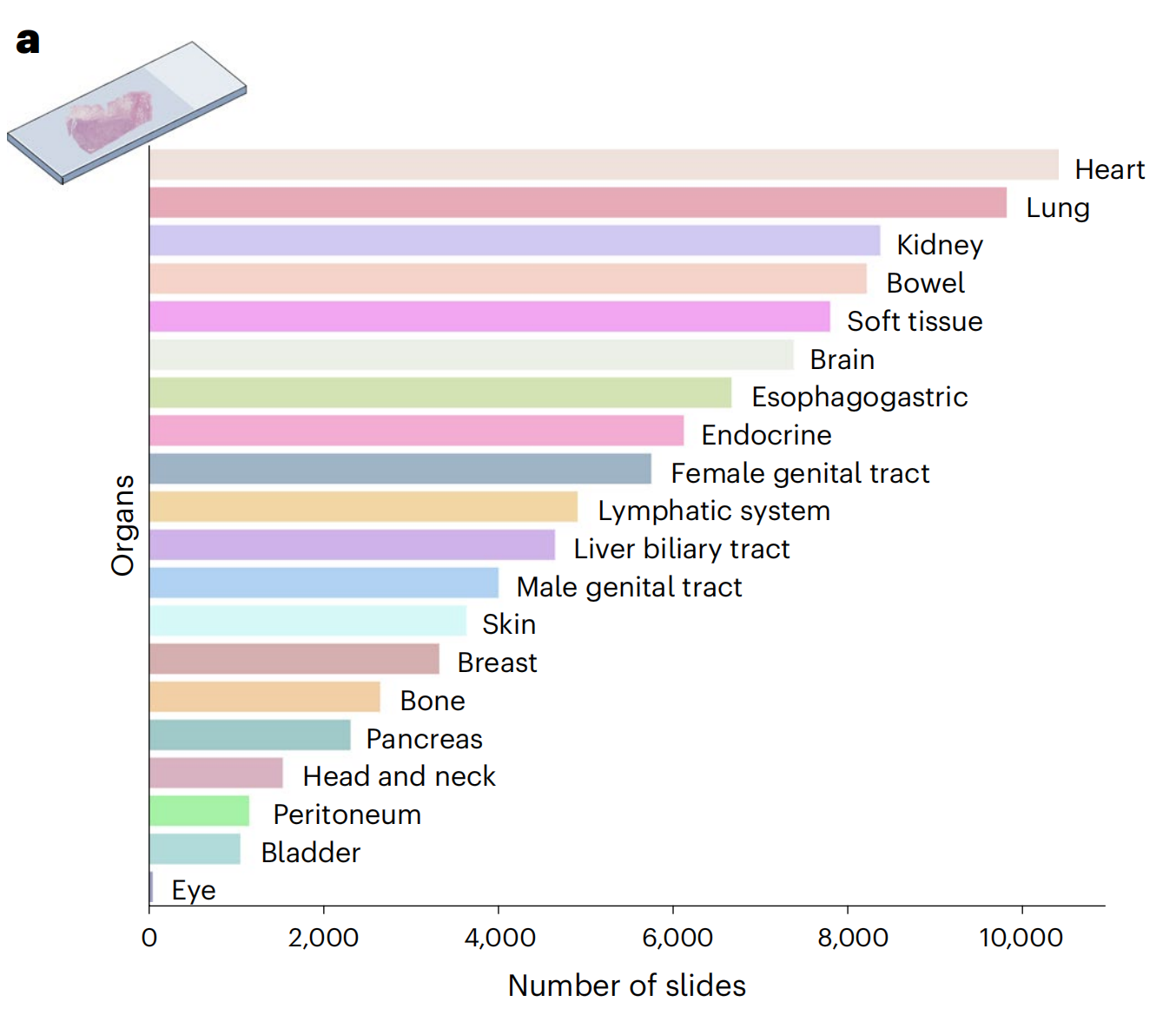
Mass-100K是一个预训练数据集,包含来自马萨诸塞州总医院(MGH)和布里格姆妇女医院(BWH)以及基因型-组织表达(GTEx)联盟76的100,426张诊断性H&E WSIs的超过10亿个组织斑块,涵盖20个主要组织类型,为学习组织病理学生物标志物的客观表征提供了丰富的信息来源(图1a和补充表1-3)。
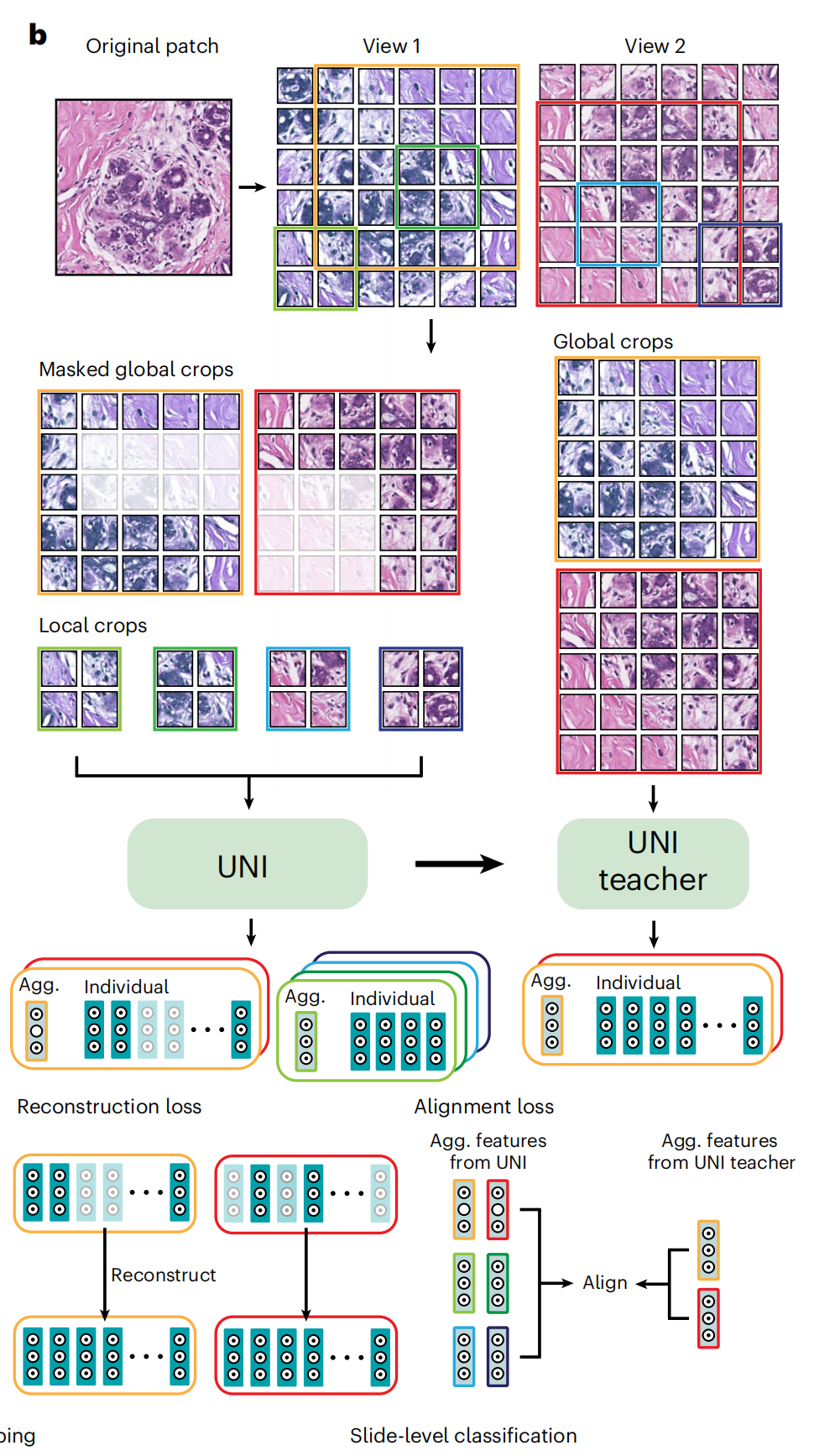
在预训练阶段,作者使用了一种名为DINOv2的自监督学习方法22,该方法已被证明无需进一步使用标注数据进行微调,即可为下游任务提供强大、即用的表征(图1b)。该算法包括一个掩码图像建模目标和一个自我蒸馏目标。这意味着 UNI 通过自监督学习,能够无需人工标注即可学习到图像的特征表示。
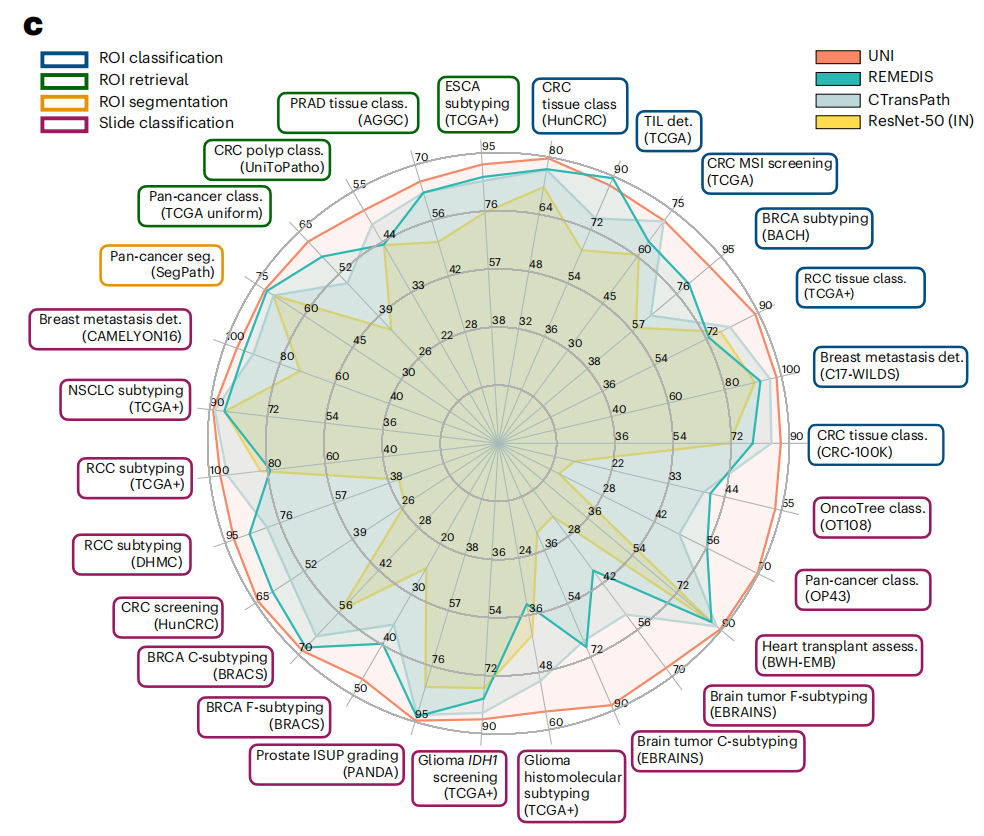
作者在CPath的多样化机器学习设置中展示了UNI的多功能性,包括ROI级分类、分割和图像检索,以及切片级弱监督学习(图1c)。在34个不同的临床病理学任务中,UNI 普遍优于其他预训练编码器,报告的是8个SegPath任务的平均性能。这显示了 UNI 在多种病理学应用中的泛化能力和高效性。
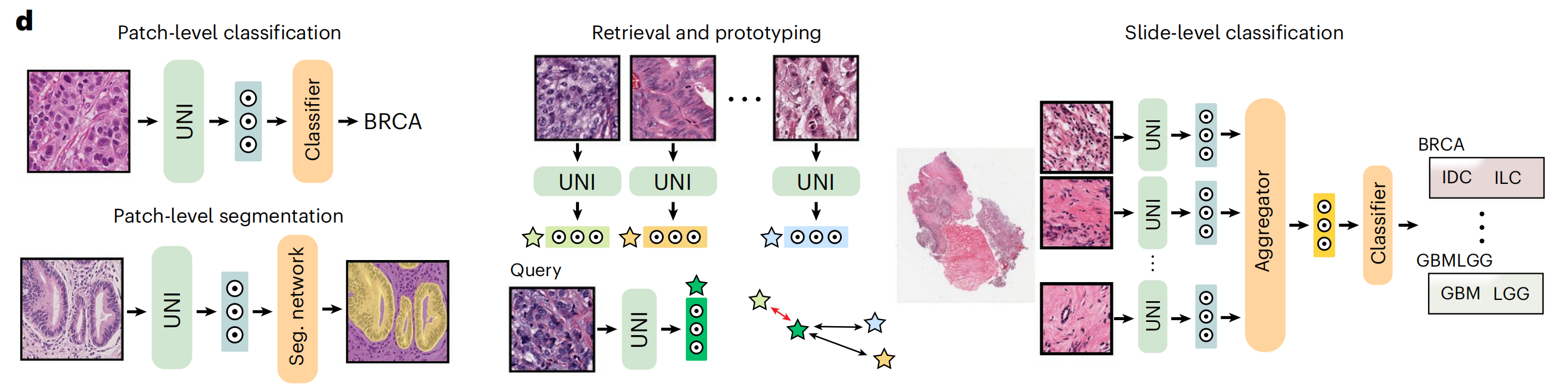
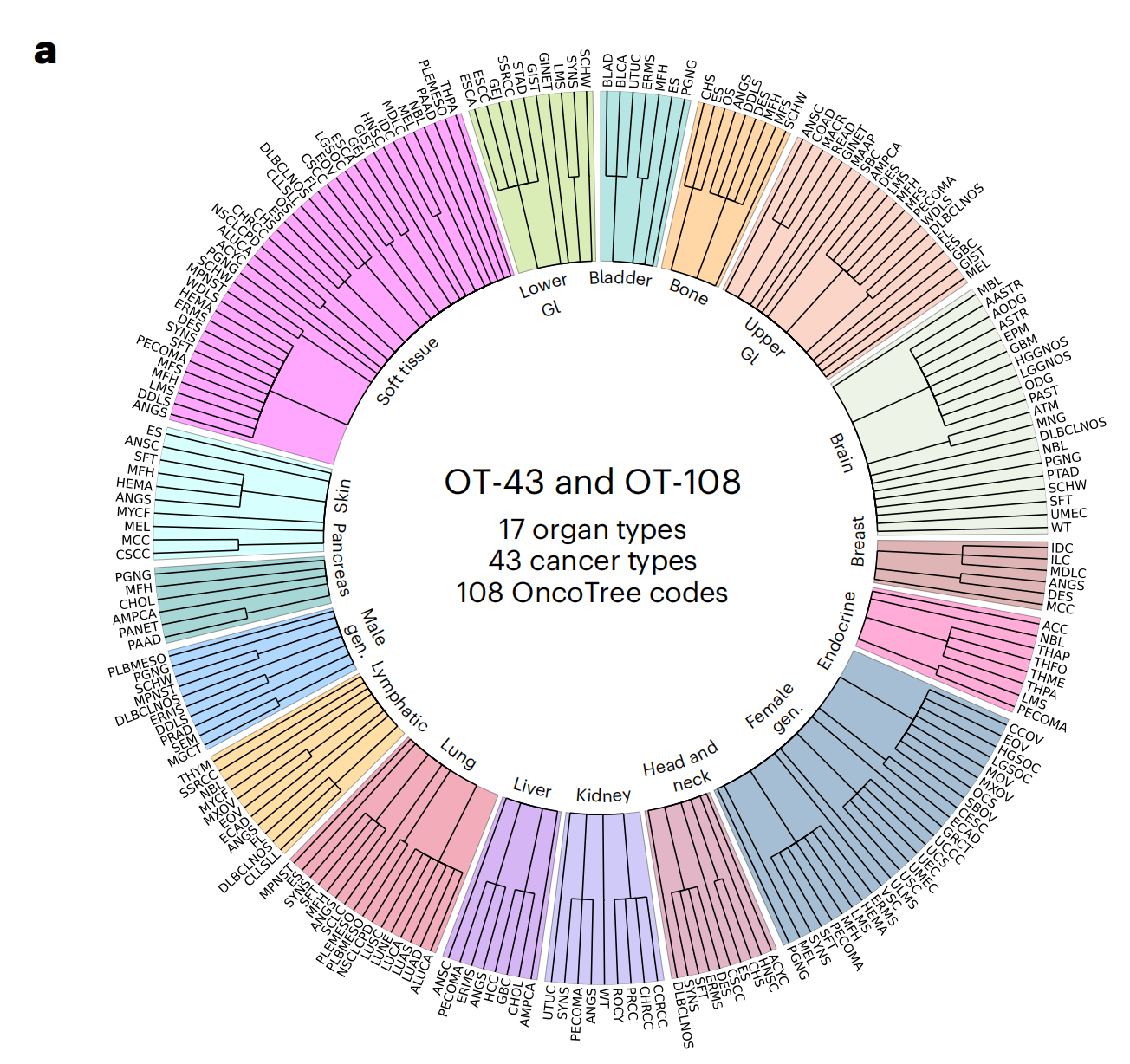
总的来说,作者评估了UNI在解剖病理学和一系列诊断难度临床任务中的应用,如核分割、原发性和转移性癌症检测、癌症分级和亚型分类、生物标志物筛选和分子亚型分类、器官移植评估,以及包括OncoTree癌症分类系统77中108种癌症类型亚型分类在内的多项泛癌分类任务(图1d和2a)。
知识点补充:OncoTree 癌症分类系统
OncoTree 癌症分类系统(OT)是一个专门用于肿瘤分类的系统,它提供了一种标准化的方法来对不同类型的癌症进行分类和编码。这个系统特别适用于精准肿瘤学(precision oncology),目的在于根据肿瘤的具体生物特征为患者提供个性化的治疗方案。
以下是OncoTree系统的几个关键特点:
- 层次化结构:OncoTree采用一个层次化的结构来组织癌症类型,从广泛的癌症类别细分到更具体的亚型。
- 多维度分类:它不仅基于癌症的解剖位置,还可能结合组织学类型、分子特征、遗传变异等多维度信息进行分类。
- 编码系统:每种癌症类型和亚型在OncoTree中都有一个独特的代码,便于在临床和研究中进行准确沟通和数据记录。
- 灵活性和扩展性:随着新癌症类型的发现和对癌症理解的深入,OncoTree可以更新和扩展,以包含新的分类。
- 临床应用:在临床上,OncoTree可以辅助医生做出更准确的诊断,为患者提供针对性的治疗建议。
- 研究应用:在癌症研究中,OncoTree提供了一种统一的分类标准,有助于不同研究之间的比较和数据整合。
在上文提到的研究中,作者利用OncoTree癌症分类系统构建了一个大规模的、分层的癌症分类任务,用以评估UNI模型在不同组织类型和疾病类别中的泛化能力。这项任务包括了43种癌症类型分类(OT-43)和108种癌症亚型分类(OT-108),涵盖了常见的和罕见的癌症类型,为评估模型提供了广泛的疾病实体样本。
评估任务包括了区域感兴趣(ROI)级别的分类、分割、检索和原型制作,以及幻灯片级别的分类任务。这表明 UNI 不仅能够在图像的局部区域进行精确分析,还能够在整个幻灯片的尺度上进行综合评估。
此部分展示了用于幻灯片层面 OT-43 和 OT-108 分类任务的组织类型和癌症类别的分布情况。这包括了各种器官类型和癌症类型,为评估 UNI 模型的泛化能力提供了广泛的疾病类别。
头颈部
头颈部区域涵盖了多种不同类型的癌症,每种癌症都有其特定的组织学类型和生物学行为。
- ACYC: Acinic Cell Carcinoma,即腺泡细胞癌,这是一种发生在唾液腺的恶性肿瘤。
- HNSC: Head and Neck Squamous Cell Carcinoma,头颈部鳞状细胞癌,这是一种常见的头颈部癌症,通常与吸烟和饮酒有关。
- CHS: 可能是指Chondrosarcoma of the Head and Neck,即头颈部软骨肉瘤,这是一种罕见的恶性软骨肿瘤。
- ES: 可能是指Esthesioneuroblastoma,即嗅神经母细胞瘤,这是一种发生在鼻腔和鼻窦的恶性肿瘤。
- ERMS: Embryonal Rhabdomyosarcoma,即胚胎性横纹肌肉瘤,这是一种发生在儿童和青少年的恶性肿瘤,通常影响肌肉组织。
- SFT: Solitary Fibrous Tumor,即孤立性纤维瘤,这是一种罕见的间叶组织肿瘤,可以在身体多个部位出现,包括头颈部。
- SYNS: Synovial Sarcoma,即滑膜肉瘤,这是一种发生在滑膜组织的恶性肿瘤,尽管通常与关节相关,但也可以在头颈部发现。
- DLBCLNOS: Diffuse Large B-Cell Lymphoma Not Otherwise Specified,即非特指型弥漫性大B细胞淋巴瘤,这是一种常见的非霍奇金淋巴瘤,可以影响身体的多个部位,包括头颈部。
除了优于先前的最先进模型,如CTransPath37和REMEDIS38外,作者还展示了诸如无视分辨率组织分类和基于提示的切片分类的少量镜头分类原型等能力(图2d),凸显了UNI作为解剖病理学中进一步发展人工智能(AI)模型的基础模型的潜力。
二、结果
2-1:计算病理学中的预训练规模法则
基础模型的一个关键特征在于,当在更大数据集上训练时,它们能够在各种任务上提供改进的下游性能。
尽管像CAMELYON16(2016年淋巴结转移癌挑战赛(参考文献78))和TCGA非小细胞肺癌子集(TCGA-NSCLC)79这样的数据集常用于使用弱监督的多实例学习(MIL)算法15,37,40,80来基准测试预训练编码器,但它们仅来源于单个器官的组织切片,并且通常用于预测二元疾病状态81,这与实际解剖病理学实践中遇到的更广泛的疾病实体不符。
相反,作者通过构建一个遵循OncoTree癌症分类系统77的大规模、分层和罕见癌症分类任务来评估UNI在多样组织类型和疾病类别中的泛化能力。使用BWH内部的切片,作者定义了一个包含来自43种癌症类型的5,564张WSIs的数据集,进一步细分为108个OncoTree代码,每个OncoTree代码至少有20张WSIs。
在108个癌症类型中,有90个被定义为罕见癌症,这是根据RARECARE项目82和国家癌症研究所的监测、流行病学和最终结果(NCI-SEER)计划所定义的。该数据集构成了两个任务的基准,这两个任务的诊断难度不同:43类OncoTree癌症类型分类(OT-43)和108类OncoTree代码分类(OT-108)(图2a和补充表4)。
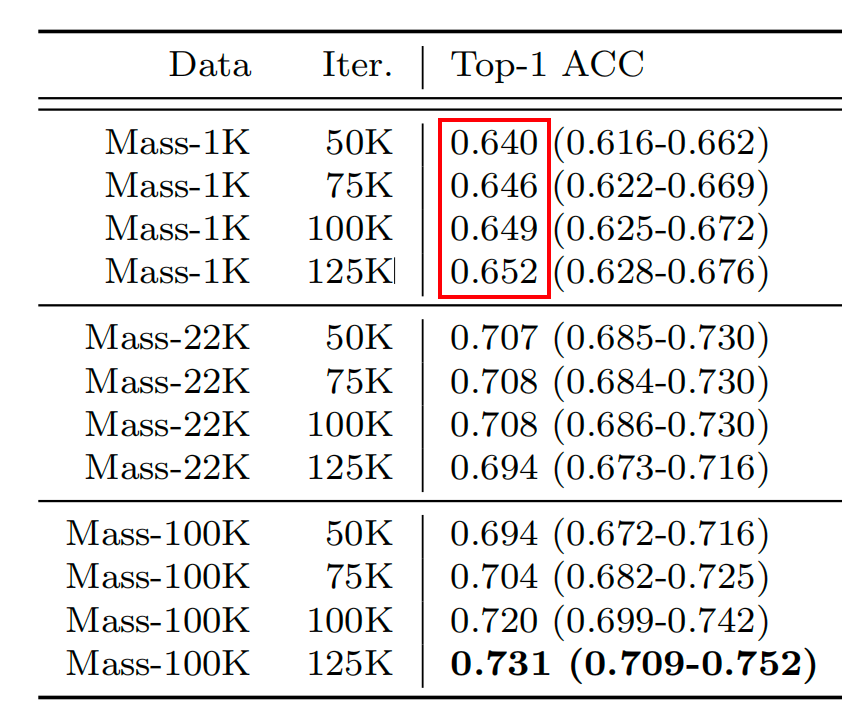
这些大型多类分类任务的目标不一定是为了临床实用性,而是为了评估基础模型的能力以及与其他模型相比特征表征的丰富性。为了评估规模趋势,作者还对UNI进行了不同数据规模的预训练,将Mass-100K子集化为Mass-22K(1600万张图像,21,444张WSIs)和Mass-1K(100万张图像,1,404张WSIs)。作者还通过使用两种不同的ViT架构大小:ViT-Base(或ViT-B)和ViT-Large(或ViT-L)来评估模型规模。
最后,作者还评估了自监督学习算法选择的影响,并与MoCoV3(参考文献24)进行了比较。对于弱监督的切片分类,作者遵循传统的范式,首先使用预训练的编码器从WSI中的组织斑块中预提取斑块级特征,然后训练基于注意力的MIL(ABMIL)算法83。
为了反映这些任务的标签复杂性挑战,作者报告了top-K准确率(K=1,3,5)以及加权F1分数和受试者工作特征曲线下面积(AUROC)性能。关于OT-43和OT-108任务的额外细节、实验设置、实施细节和性能分别提供在方法、补充表1-11和补充表12-18中。
总体而言,作者展示了UNI中自监督模型在模型和数据规模方面的能力,OT-43和OT-108上UNI的规模趋势如图2c,e所示。
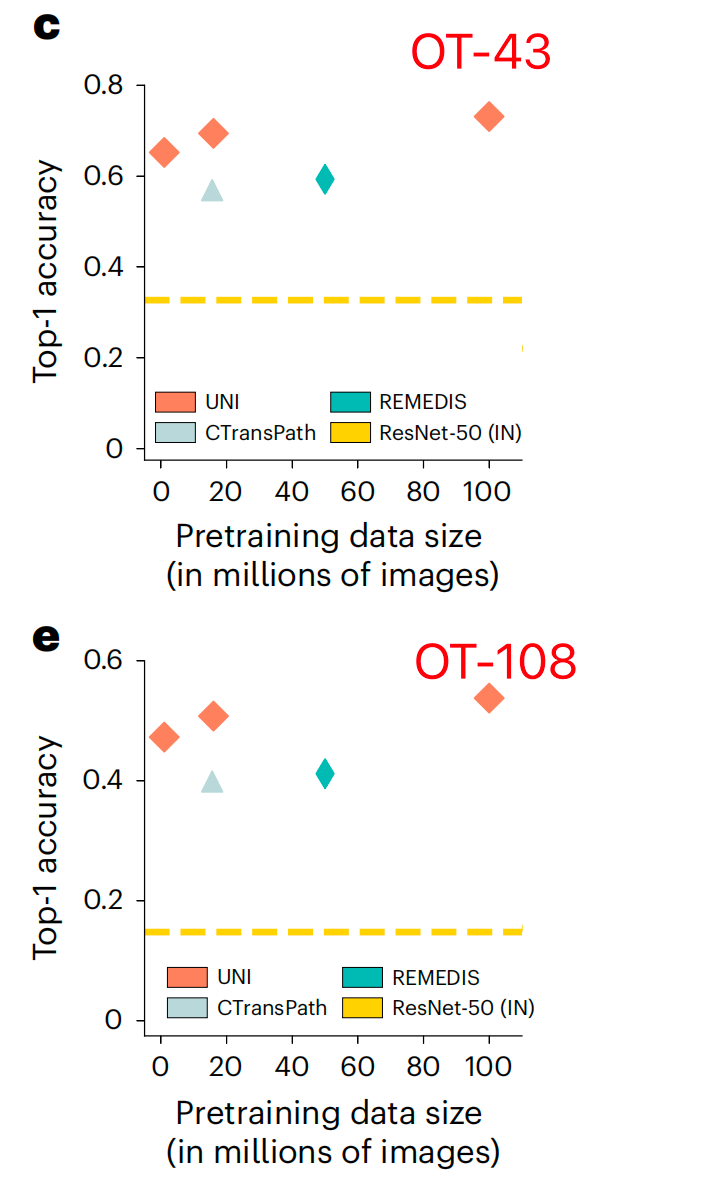
在OT-43和OT-108上,作者观察到当使用ViT-L将UNI从Mass-1K扩展到Mass-22K时,top-1准确率提高了+4.2%(P<0.001,双尾配对置换检验),OT-108上类似的提高了+3.5%(P<0.001)。从Mass-22K到Mass-100K,性能进一步提高:OT-43和OT-108分别提高了+3.7%和+3.0%(P<0.001)。使用ViT-B也观察到了类似的趋势,从Mass-22K到Mass-100K时性能趋于平稳(补充表13和16)。
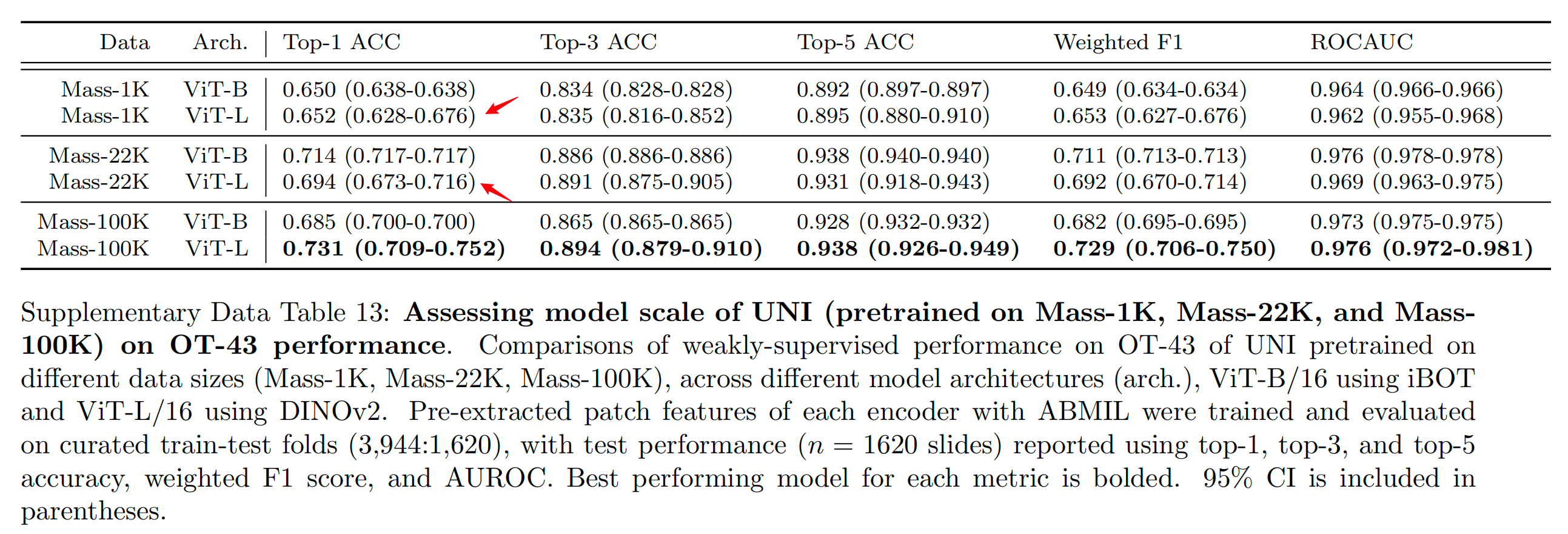
补充表14和17显示了数据多样性和预训练长度的影响,从50,000到125,000训练迭代,两个任务上都呈现单调改善。
总体而言,这些规模趋势与在许多应用于自然图像的ViT模型21,31,75中观察到的发现一致,即随着预训练数据集的增长,较大ViT变体的性能得到提高。探索其他自监督学习算法时,作者还使用ViT-L和ResNet-50骨干在Mass-1K上训练了MoCoV3(参考文献24),其表现不如DINOv2(补充表18)。
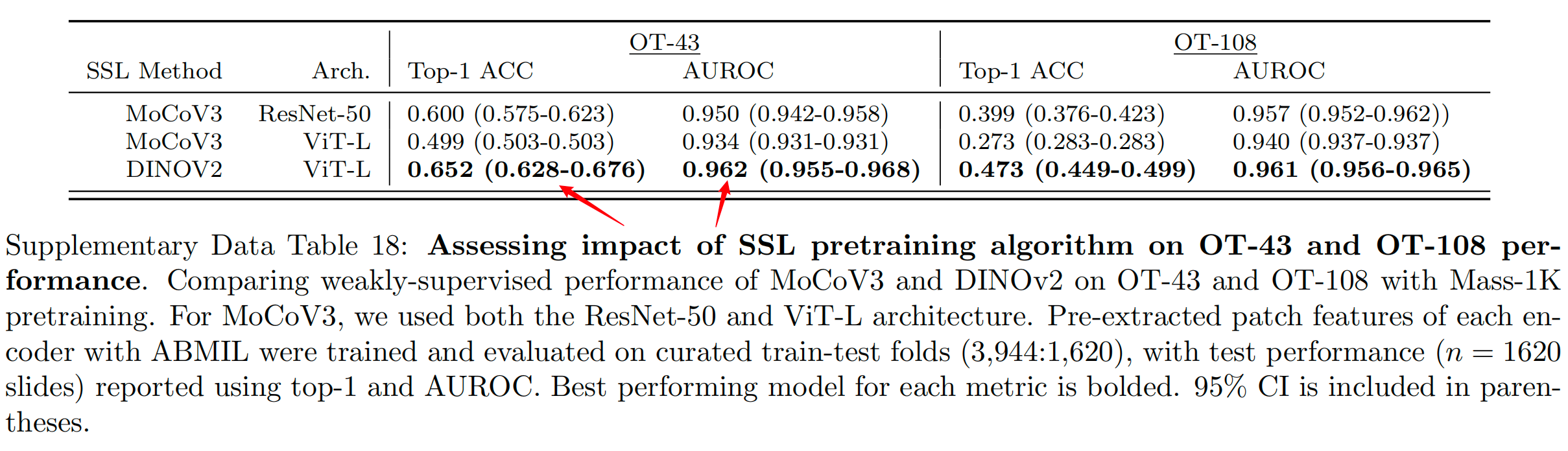
为了随着模型和数据规模的增加而提升性能,算法及其超参数的选择在开发CPath基础模型时也很重要。
作者将使用ViT-L在Mass-100K上预训练的UNI与CPath中公开可用的预训练编码器进行了比较,比较的任务是OT-43和OT-108:
- 在ImageNet-1K上预训练的ResNet-50(参考文献84);
- 在TCGA和PAIP(病理学人工智能平台)85上预训练的CTransPath37;
- 在TCGA上预训练的REMEDIS38。
作者观察到UNI在所有基准测试中都以显著的优势超越了所有基线模型。在OT-43任务中,UNI实现了93.8%的top-5准确率和0.976的AUROC,比表现第二好的模型(REMEDIS)在这些指标上分别高出+6.3%和+0.022(均P<0.001)(图2b和补充表12)。
Supplementary Data Table 12 提供了基于 BWH(Brigham and Women’s Hospital)内部数据的 43 类癌症类型弱监督分类(OT-43)任务的评估结果。
在这个任务中,使用了 ABMIL(Attention-based Deep Multiple Instance Learning)算法来训练和评估预提取的图像编码器特征。数据集被划分为训练集和测试集,比例为 3,944:1,620。
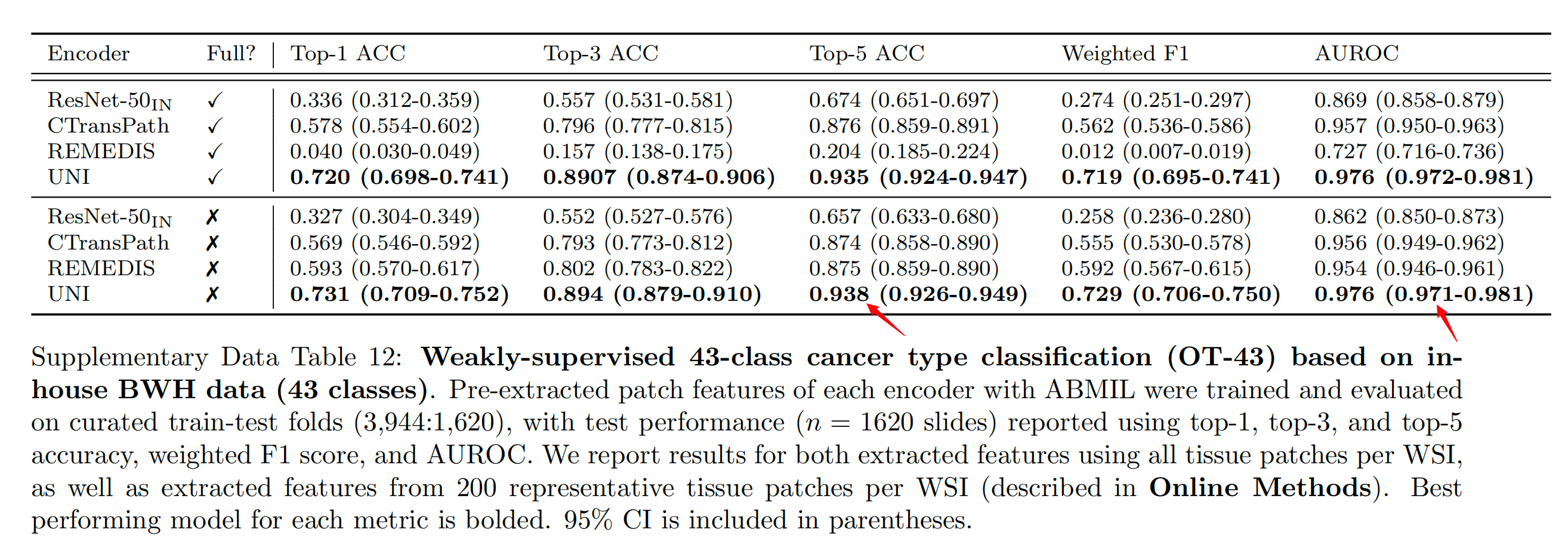
评估使用的指标包括:
- Top-1 ACC(准确率):模型预测的最有可能的类别与实际类别一致的比例。
- Top-3 ACC:模型预测的前三个最可能的类别中包含实际类别的比例。
- Top-5 ACC:模型预测的前五个最可能的类别中包含实际类别的比例。
- Weighted F1:考虑到类别不平衡的 F1 分数,是精确度和召回率的调和平均数。
- AUROC(Area Under the Receiver Operating Characteristic Curve):接收者操作特征曲线下的面积,衡量模型在所有可能的分类阈值上的性能。
表中报告了两种情况的评估结果:
- 使用每个 WSI 的所有组织补丁(tissue patches)提取的特征。
- 从每个 WSI 中提取的 200 个代表性组织补丁中提取的特征。
结果中加粗的模型在相应指标上表现最佳。同时,表中还包括了 95% 置信区间(CI),这有助于理解模型性能的统计显著性。
在OT-108任务中,作者观察到与REMEDIS相比,性能提升的幅度相似,分别为+10.8%和+0.020(P<0.001)(图2c和补充表15)。
总体而言,作者发现UNI能够在OT-43和OT-108中分类罕见癌症,并且在所有预训练编码器中实现了性能的大幅提升。
2-2:弱监督切片分类
此外,作者探讨了UNI在15个不同切片级分类任务中的能力,这些任务包括:
- 乳腺癌转移检测(CAMELYON16)78
- 前列腺癌的国际泌尿病理学会(ISUP)分级(PANDA)18
- 心脏移植评估(BWH内部切片)86
- 脑肿瘤亚型分类(EBRAINS;代表RARECARE项目定义的30种罕见癌症)
与OT-43和OT-108评估类似,作者使用ABMIL83比较了UNI预提取特征与其他预训练编码器的表现。
鉴于CTransPath和REMEDIS几乎使用了所有TCGA切片进行训练,这些模型在TCGA任务上的报告性能可能因数据泄露而受到污染,从而不公平地膨胀。关于切片任务、实验设置和性能的额外细节分别提供在方法、补充表19-21和补充表22-35中。
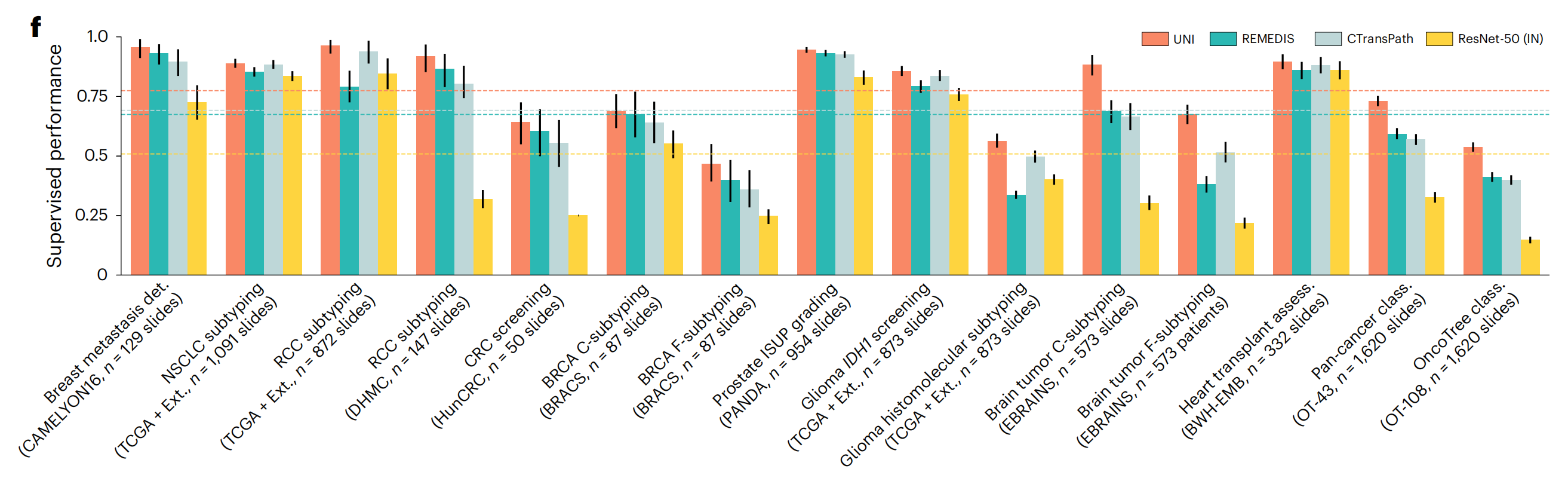
在所有15个切片级任务中,UNI始终优于其他预训练编码器(平均性能分别比ResNet-50提高+26.4%,比CTransPath提高+8.3%,比REMEDIS提高+10.0%),在分类罕见癌症类型或具有较高诊断复杂性的任务上观察到更大的改进(图2f)。
在前列腺ISUP分级(PANDA)任务中,UNI实现了0.946的加权Cohen’s κ值,比表现第二好的模型(REMEDIS)高出+0.014(P<0.05)(补充表29)。
Supplementary Data Table 29 展示了基于 PANDA 数据集的弱监督 ISUP(国际泌尿病理学会)分级任务的评估结果。ISUP 分级是一种对前列腺癌进行分级的方法,这项任务将前列腺癌的 WSIs 分为 6 个类别。
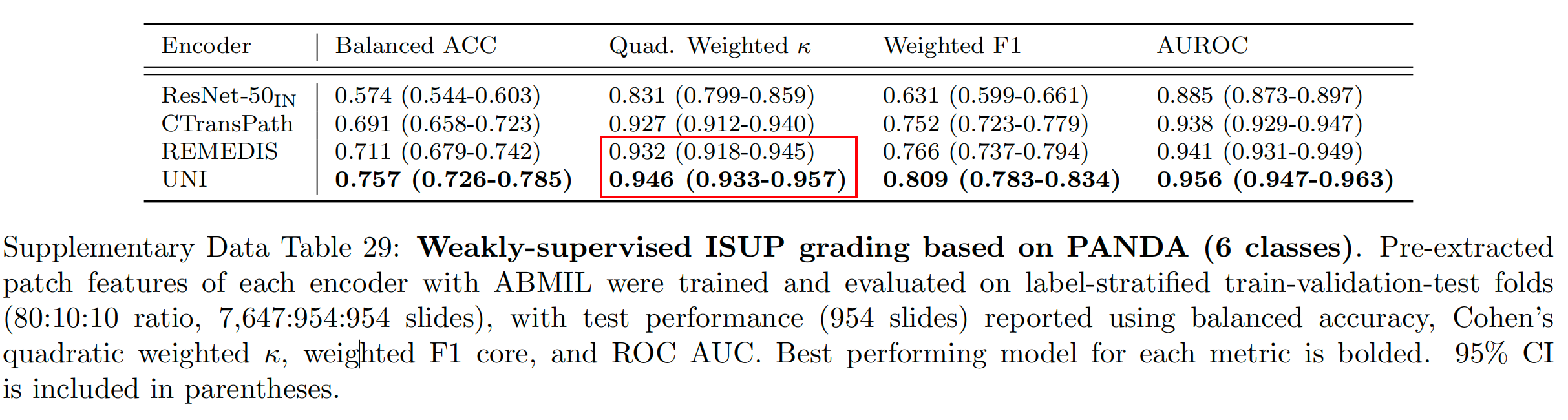
以下是表格中的关键点分析:
- 数据集划分:数据被划分为训练集、验证集和测试集,比例为 80:10:10。具体来说,有 7,647 张幻灯片用于训练,954 张用于验证,954 张用于测试。
- 评估模型:使用了不同的编码器(如 ResNet-50IN, CTransPath, REMEDIS, UNI)来提取图像特征,然后通过 ABMIL(Attention-based Multiple Instance Learning)算法进行训练和评估。
- 评估指标:- 平衡准确率(Balanced Accuracy):考虑到类别不平衡,通过计算所有类别的召回率的未加权平均值来得到。- Cohen’s Quadratic Weighted Kappa(Cohen’s Quadratic Weighted ):这是一种统计度量,用于衡量分类任务中标签的一致性,考虑到了类别的权重。- 加权 F1 分数(Weighted F1 Score):F1 分数是精确度和召回率的调和平均数,这里的加权 F1 分数考虑了每个类别的样本数量。- 接收者操作特征曲线下面积(AUROC):衡量模型在所有可能的分类阈值上的整体性能。
- 结果报告:测试性能在 954 张测试幻灯片上进行了报告,使用了上述指标。
- 最佳性能模型:每个指标的最佳性能模型被加粗显示,这表示在该指标上模型表现最为出色。
- 置信区间:表格中包括了 95% 置信区间(CI),这有助于理解模型性能的统计显著性,并提供了对结果可靠性的估计。
- 模型比较:通过比较不同模型在同一指标下的表现,可以评估它们在 ISUP 分级任务上的优劣。例如,如果 UNI 模型在所有指标上都是加粗的,那么它将是这个任务中表现最好的模型。
总的来说,这个表格提供了一个详细的性能比较,展示了不同模型在前列腺癌分级任务上的效果,以及它们在不同评估指标下的表现。
在涉及罕见疾病类别的分层分类任务(如胶质瘤生物标志物预测(使用TCGA87,88和EBRAINS89的2类IDH1突变预测和5类组织分子亚型)和脑肿瘤亚型分类(使用EBRAINS的12类粗粒度和30类细粒度脑肿瘤亚型))上,UNI优于表现第二好的模型(CTransPath或REMEDIS),分别提高+2.0%(P=0.076)、+6.4%(P=0.001)、+19.6%(P<0.001)和+16.1%(P<0.001)(补充表31-34)。与OT-43和OT-108类似,作者发现UNI在仅涉及罕见癌症类型的脑肿瘤亚型分类任务上的影响最大。
在比较现有排行榜时,作者发现带有UNI特征的ABMIL优于许多复杂的MIL架构。
在乳腺癌转移检测(CAMELYON16)任务中,带有UNI的ABMIL优于所有该任务的最新MIL方法(补充表36),并且是在原始挑战78中唯一一个无需时间限制就超过人类病理学家表现(AUROC为0.966)的MIL结果之一。
在前列腺ISUP分级(PANDA)和细胞介导的异体移植排斥(BWH-EMB)等详细比较的任务中,带有UNI的ABMIL优于WholeSIGHT90和CRANE86等方法(补充表37和38)。
尽管许多这些比较并不完全等同,因为使用了ResNet-50与ImageNet转移(ResNet-50IN)特征,但作者注意到他们提出的MIL架构通常是为了解决这些挑战性任务而专门开发和动机的。作者的比较突出了拥有更好的预训练编码器与MIL架构的优势。
数据污染是训练于大型公共数据集的基础模型的一个关注点91–95。
尽管在自监督训练期间标签可能没有明确泄露到模型中,但预训练于评估测试集的模型可能在其他CPath研究中表现出乐观偏差的性能96。
此外,作者将UNI与CTransPath和REMEDIS在非小细胞肺癌(NSCLC)亚型分类、肾细胞癌(RCC)亚型分类、胶质瘤IDH1突变预测和胶质瘤组织分子亚型分类任务的TCGA测试集上进行了比较,观察到在域内与域外性能比较时的性能下降。
在NSCLC亚型分类任务上,REMEDIS在TCGA评估中优于UNI(97.3%对94.7%),但在CPTAC(临床蛋白质组肿瘤分析联盟)评估中表现不佳(79.0%对96.3%)(补充表23)。
在胶质瘤IDH1突变预测任务上,CTransPath和REMEDIS在TCGA评估中优于UNI(89.1%和81.9%对80.8%),但在EBRAINS评估中表现不佳(83.6%和79.2%对85.6%)(补充表31和32)。
作者强调,数据污染仅存在于模型的使用方式中,而不在于模型本身,这些模型已被证明在独立于TCGA的环境中具有良好的迁移性38,59,97。鉴于许多CPath研究使用TCGA来研究多种癌症类型,UNI在开发公共组织学数据集和基准测试中的灵活性优于CTransPath和REMEDIS。
综上所述,UNI模型在多种计算病理学任务中展现出卓越的性能,其预训练的特征提取能力优于现有的预训练编码器。在各种切片级分类任务中,UNI不仅优于其他预训练模型,而且在涉及罕见癌症类型或具有较高诊断复杂性的任务上表现尤为突出。
此外,UNI在基准测试中的表现也表明了其对数据污染问题的鲁棒性,这使得它在开发基于公共组织学数据的病理学人工智能模型方面具有更大的灵活性和适用性。
2-3:少数样本切片分类的标签效率
作者还评估了UNI在所有切片级任务中的少数样本多实例学习(MIL)性能。
少数样本学习是一种评估方案,研究模型在新任务(C类)上的泛化能力,给定有限的示例数量(每类K个训练样本,也称为支持或示例)。
对于所有预训练的编码器,作者使用K ∈ {1, 2, 4, 8, 16, 32}个训练示例对每类进行训练,其中K限制为32,因为罕见疾病类别中的支持大小较小。考虑到每个类的K示例选择会影响性能,作者重复了五轮实验,每次随机采样C × K个训练示例。关于少数样本MIL实验和性能的额外细节提供在方法和扩展数据图1中。
UNI在所有任务上通常优于其他预训练编码器,并且在标签效率方面表现出色,特别是在分类罕见疾病方面(图2g-j和扩展数据图1)。
当比较UNI与其他编码器(使用中位数性能)的4样本性能时,表现第二好的编码器需要每类8倍于UNI的训练示例数才能达到相同的4样本性能。在前列腺ISUP分级(PANDA)任务上,UNI在所有少数样本设置下都表现出两倍的标签效率(图2j)。
在挑战性罕见癌症亚型分类任务,如细粒度脑肿瘤亚型(EBRAINS)上,UNI的4样本性能明显优于其他编码器,只有在32样本设置下才能与REMEDIS的性能相匹配(图2i)。总的来说,作者对切片分类任务的全面评估表明,UNI具有作为基础模型,用于筛查罕见和代表性不足疾病的组织病理学工作流程的潜力。
2-4:监督ROI分类中的线性分类器
除了切片级任务外,作者还评估了UNI在11个ROI级任务上的性能,这些任务包括:
- 结直肠组织和息肉分类(CRC-100K-NONORM98、HunCRC99、UniToPatho100)
- 前列腺腺癌组织分类(Automated Gleason Grading Challenge 2022 (AGGC)101)
- 泛癌肿瘤-免疫淋巴细胞检测(TCGA-TILS67)
- 32类泛癌组织分类(TCGA Uniform Tumor68)
为了评估和比较,作者在每个编码器的预提取特征上执行逻辑回归和K最近邻(KNN),这通常被称为线性探测和KNN探测,分别测量判别性能和预提取特征的表示质量23。
作者使用平衡准确度评估所有任务,其中PRAD组织分类使用加权F1分数101进行评估。关于ROI任务、实验设置和性能的额外细节提供在方法和补充表39-60中。
在所有11个ROI级任务中,UNI在所有任务上都几乎优于所有基线,在逻辑回归探测中对ResNet-50、CTransPath和REMEDIS的平均性能分别提高了+18.8%、+7.58%和+5.75%(图3a)。

在KNN探测中,UNI同样优于ResNet-50、CTransPath和REMEDIS,平均性能分别提高了+15.6%、+8.6%和+9.4%。作者发现在挑战性任务上,如PRAD组织分类(在加权F1分数上,分别提高了+0.131、+0.020和+0.027,P<0.001)和食管癌亚型分类(分别提高了+25.3%、+10.1%和+5.5%,P<0.001),相对于其他三个预训练编码器,UNI的增益更大。
图3b显示了前列腺癌分级中UNI的预测,其中使用预提取的UNI特征训练的简单线性分类器与病理学家注释达到了高度一致(扩展数据图2)。
在32类泛癌组织分类任务(其中19类是罕见癌症)上,UNI实现了最高的平衡准确度和AUROC,分别为65.7%和0.975,优于表现第二好的模型(REMEDIS)+4.7%和+0.017(P<0.001)。
在肿瘤-免疫淋巴细胞检测任务中,与ChampKit基准中报告最佳模型的AUROC为0.974和假阴性率(FNR)为0.246相比,UNI的AUROC为0.978,FNR为0.193(未进行染色标准化)(补充表61)。
在乳腺癌转移检测(CAMELYON17-WILDS排行榜)中,与目前最佳模型相比,其在外域验证和测试集上的准确度分别为95.2%和96.5%,UNI达到了97.4%和98.3%(补充表62)。
作者注意到,许多这些比较都是在自然图像上进行端到端微调的,而不是从病理学中进行。尽管与UNI的实验不同,但这些比较突出了UNI的多样性,因为使用线性分类器的开箱即用评估与使用端到端微调的最新技术具有竞争力。
2-5:ROI检索
除了使用UNI的表示来构建特定任务的分类器之外,表示还可以用于图像检索。
检索类似于KNN,作者评估查询图像能否很好地检索到同一类的其他图像,因为视觉上相似的图像在表示空间中应该比视觉上不同的图像更接近。
与KNN评估不同,作者考虑检索的准确性,即Acc@K对于K ∈ {1, 3, 5},其中如果一个正确标记的图像在检索到的前K个图像中,则检索成功;MVAcc@5使用前5个检索到的图像的多数投票。作者在六个ROI级任务上评估了组织学图像检索(至少有5个类别)。
关于ROI检索实验和性能的额外细节提供在方法、扩展数据图3和补充表63-68中。
UNI在所有任务上都优于其他编码器,展示了在各种设置下卓越的检索性能。
在PRAD组织分类(AGGC)任务上,UNI在Acc@1和MVAcc@5上分别比表现第二好的编码器(REMEDIS)高出+4%和+3.3%(P<0.001)(图2c)。
在结直肠癌(CRC)组织分类(CRC-100K)任务上,顶级编码器之间的差距相对较小(与REMEDIS相比,Acc@1高出+3.1%,P<0.001,MVAcc@5高出+0.01%,P=0.188),这可能是因为不同的组织类型具有非常独特的形态,正如线性探测中的高分类性能所展示的那样。
在更具挑战性的32类泛癌组织分类任务上,包含许多罕见癌症类型,UNI比表现第二好的编码器(REMEDIS)在Acc@1上高出+4.6%,在MVAcc@5上高出+4.1%(P<0.001)。
2-6:高分辨率图像的鲁棒性
尽管视觉识别模型通常在调整后的224×224像素(224²像素)图像上进行评估,但图像缩放会改变每像素微米数(mpp),并可能改变对细胞异型等形态特征的解释。
作者研究了UNI在不同分辨率下的特征质量如何受到影响,这些分辨率用于:
- 乳腺癌浸润性癌(BRCA)亚型分类(乳腺组织学图像的挑战赛,BACH)(从224²像素在2.88 mpp到1,344²像素在0.48 mpp)
- 结直肠息肉分类(UniToPatho)(从224²像素在3.60 mpp到1,792²像素在0.45 mpp)
并使用线性探测和KNN探测。
关于多分辨率实验和性能的额外细节提供在方法、扩展数据图4和补充表45、46、51和52中。
在这两个任务上,作者展示了UNI对不同图像分辨率以及高分辨率ROI任务中引入的图像缩放偏差的鲁棒性。当调整用于评估的图像分辨率时,作者观察到其他编码器在性能上的下降更为严重,例如在BRCA亚型分类中,CTransPath和REMEDIS的KNN性能分别下降了-18.8%和-32.5%(从224²像素到1,344²像素),而UNI的下降仅为-6.3%。
在结直肠息肉分类中,尽管其他编码器的性能没有显著下降(从224²像素到1,792²像素),UNI通过KNN探测提高了+5.1%。图2e和扩展数据图5、6展示了UNI在评估高分辨率图像时如何突出显示更细粒度的视觉特征。

在结直肠息肉分类中,将图像缩放到224²像素会模糊重要的细粒度细节,这些细节在高分辨率下被UNI检测到。这些观察结果表明,UNI可以编码与大多数图像分辨率无关的语义上有意义的表示,这在已知在不同放大倍数下达到最佳效果的CPath任务中可能非常有价值。
2-7:ROI细胞类型分割
作者评估了UNI在最大的公开ROI级分割数据集SegPath102上的性能,这是一个用于分割肿瘤组织中八种主要细胞类型的数据集:
- 上皮细胞
- 平滑肌细胞
- 红细胞
- 内皮细胞
- 白细胞
- 淋巴细胞
- 浆细胞
- 髓细胞
所有预训练的编码器都使用Mask2Former103进行端到端微调,这是一种通常用于评估预训练编码器开箱即用性能的灵活框架22,104。
由于SegPath数据集将细胞类型划分为单独的密集预测任务(总共八个任务),每个编码器都针对每个细胞类型进行单独微调,使用Dice分数作为主要评估指标。关于分割任务和性能的额外细节提供在方法和补充表69中。
尽管层次视觉骨干网络(如Swin变换器(CTransPath)和卷积神经网络(CNNs;ResNet-50和REMEDIS))在分割任务上比视觉变换器(UNI)具有众所周知的优势,但作者观察到UNI在SegPath中大多数细胞类型上仍然优于所有比较。
在单独的上皮、平滑肌和红细胞类型分割任务上,UNI分别实现了Dice分数0.827、0.690和0.803,分别优于表现第二好的编码器(REMEDIS)+0.003(P=0.164)、+0.016(P<0.001)和+0.008(P=0.001)。
在SegPath中的所有八种细胞类型上,UNI实现了平均Dice分数0.721,优于ResNet-50(0.696)、CTransPath(0.695)和REMEDIS(0.716)。扩展数据图7展示了UNI和其他编码器对所有细胞类型的分割可视化,所有比较在匹配地面实况分割方面都表现良好。
总的来说,作者发现UNI可以在分割任务上超越最先进的CNN和层次视觉模型,将其在非传统设置中的多功能性扩展到新的领域。
2-8:基于类原型的提示驱动切片分类
尽管通过MIL的弱监督学习已经改变了切片级分类,使得不再需要ROI注释81,但获取和组织切片收藏仍然可能成为解决罕见和代表性不足疾病的临床任务的障碍。观察到UNI强大的检索性能和少数样本能力,作者重新审视了使用类原型的少数样本切片分类问题。
类似于文本提示55,作者使用SimpleShot中的类原型也作为“提示”,用于对顶部K个检索到的补丁进行多数投票(顶部K池化),作者称之为多实例SimpleShot(MI-SimpleShot)(图4b)。
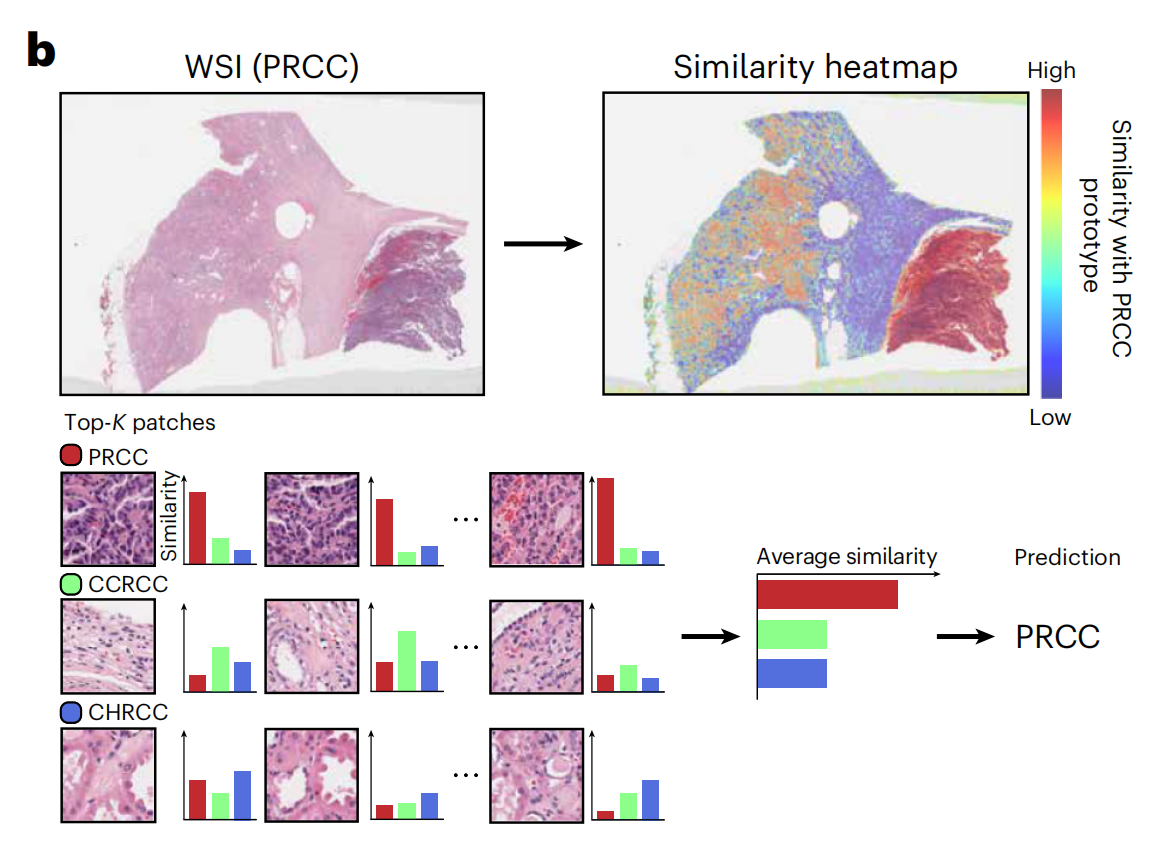
作者在与训练ABMIL模型的少数样本切片分类相同的折叠上评估MI-SimpleShot,使用来自泛癌组织分类任务68的注释ROI(来自训练切片)创建原型。作者还比较了MI-SimpleShot使用其他预训练编码器的情况,以及UNI的MIL基线。
作者还开发了相似性热图,显示了与地面实况标签的类原型相关的所有补丁与幻灯片的标准化欧几里得距离,并用蓝色标出与幻灯片标签匹配的组织区域。关于MI-SimpleShot实验和性能的额外细节提供在方法、扩展数据图9和10以及补充表70和71中。
仅使用每类少数注释的ROI示例作为原型,作者展示了应用UNI与MI-SimpleShot作为简单但高效系统进行切片级疾病亚型和检测的潜力。
在NSCLC和RCC亚型分类(在TCGA上训练并在外部队列上测试)上,使用1、2和4个训练幻灯片每类创建原型时,MI-SimpleShot使用顶部5池化优于ABMIL,并且当使用更多幻灯片时,性能与ABMIL相似(图4f、g)。
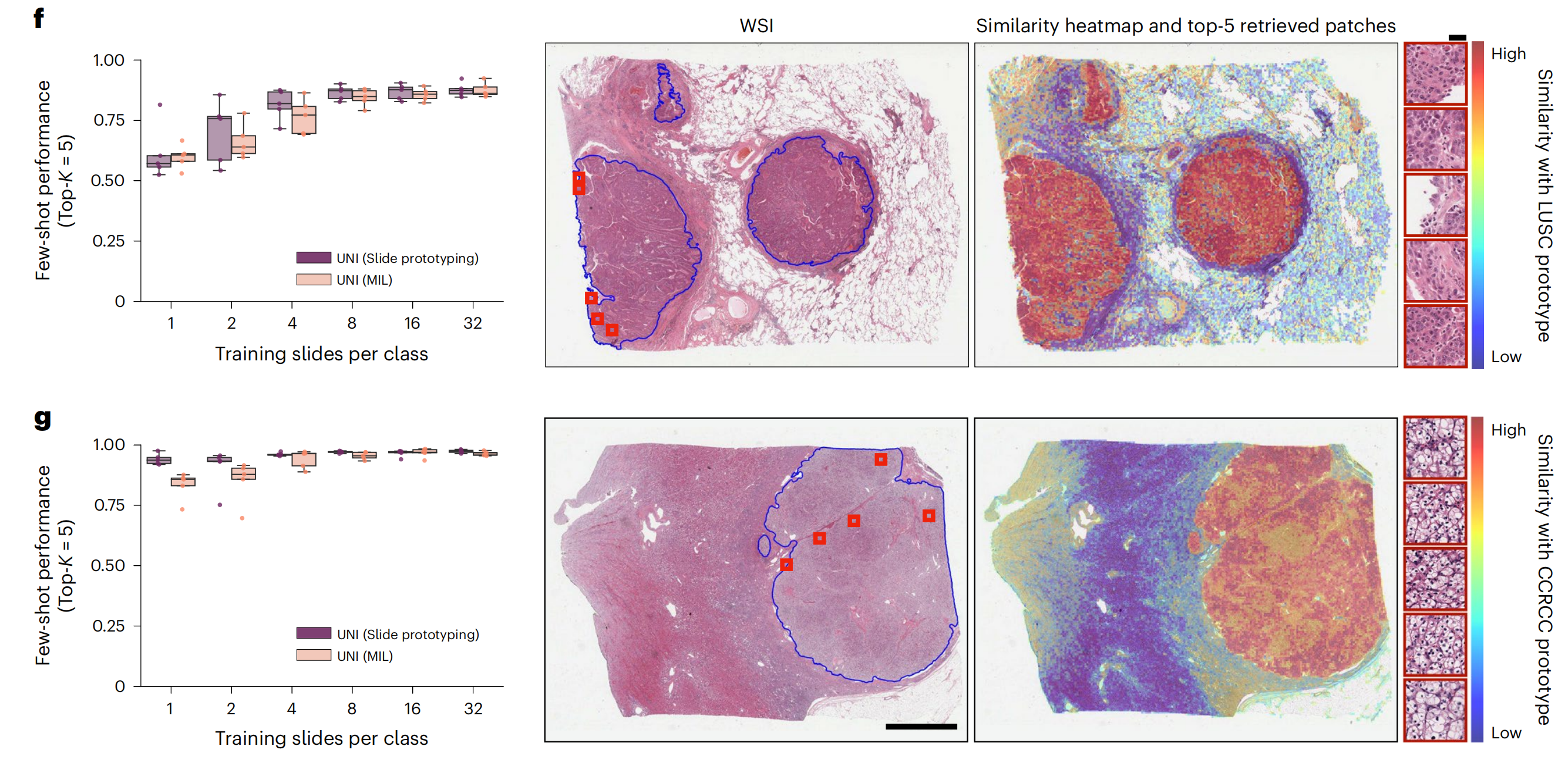
通过相似性热图,作者还观察到UNI检索到的补丁(对应于幻灯片标签)与病理学家注释有强烈的一致性,如图4f、g右侧的LUSC和透明细胞肾细胞癌(CCRCC)幻灯片所示。
作者认为MI-SimpleShot的有效性可以归因于不需要可训练参数(ABMIL模型在少数样本设置中仍可能过度和欠拟合)以及UNI特征在ROI检索中的强表示质量。尽管其他预训练编码器也可以用于MI-SimpleShot中学习原型,但UNI可能对H&E染色变异性更不敏感。这在RCC亚型分类的一样本性能的高标准差中得到了体现(在ABMIL的扩展数据图1和MI-SimpleShot的扩展数据图9中),因为MI-SimpleShot中仅使用了一个地点来学习一个类原型。这也得到了SimpleShot评估中乳腺癌转移检测(CAMELYON17-WILDS)的强调,因为CTransPath和REMEDIS在两个外域医院测试队列之间的性能差异比UNI大得多(准确度差异分别为12.3%和12.8%与5.1%),暗示了H&E染色强度可能影响检索性能的潜在效应(补充表42)。
总的来说,通过MI-SimpleShot对UNI的评估展示了具有强大检索能力的以视觉为中心的基础模型如何可能促进解剖病理学的应用。
三、讨论
在这项研究中,作者展示了UNI的多样性,这是一个通用的、自监督的模型,它在CPath中使用迄今为止最大的组织切片收藏(用于自监督学习)进行预训练。
作者整理了Mass-100K,一个预训练数据集,包含来自100,426张WSIs的超过10亿个组织斑块,涵盖20个主要器官类型,包括正常组织、癌性组织和其他病理学。使用DINOv2自监督学习方法(已证明可以扩展到大型数据集)22,作者开发并验证了一个ViT-L(在Mass-100K上预训练),它始终优于其他组织病理学图像编码器。具体取决于任务,尽管CTransPath和REMEDIS可能达到类似的性能,但作者的发现表明,这些编码器在检索能力、标签效率和在域外评估中可能对H&E染色强度存在潜在偏差。
作为一个可能促进CPath中多样化临床应用的视觉中心的基础模型,开发UNI时出现了一些挑战,例如模型和数据规模如何影响迁移性能。
尽管许多实证研究探讨了这些组成部分以实现自然图像的良好泛化,但许多解决方案可能无法移植,因为病理学和自然图像之间存在差异。例如,尽管MoCoV3在ImageNet上的性能低于DINOv2,但在Mass-1K上开发ViT-L的相同训练配置显示出在OT-108上存在显著的性能差距。
在作者研究之后,作者注意到最近有几项研究在更大的组织切片数据集和收藏上进行了训练107-109。与先前的和最近的工作不同,作者的研究独特地提供了对CPath中自监督模型缩放定律和迁移学习能力的见解。尽管模型和数据规模是构建视觉中心自监督学习的重要组成部分,但作者发现自监督学习(SSL)算法的选择最具影响力。
MoCoV3(在Mass-1K上的ViT-L)不仅低于其DINOv2对应物,而且低于CTransPath和REMEDIS。增加模型规模(从ViT-B到ViT-L)和数据规模(从Mass-1K到Mass-100K)确实反映了性能的提升,但值得注意的是,UNI在OT-43和OT-108上的删减性能相对接近,并持续优于CTransPath和REMEDIS,这表明具有竞争力的预训练编码器仍可以开发出更小的模型和更少的数据。随着UNI展示了许多临床应用,作者相信对上述因素的测试将指导CPath从业者在使用私人内部切片收藏开发自己的基础模型。
就UNI可以应用的广泛临床任务而言,与其它编码器相比,作者发现UNI在分类罕见和代表性不足的疾病方面表现突出,例如在OT-108基准中的90种罕见癌症类型,EBRAINS数字肿瘤图谱中的30种罕见脑肿瘤诊断,以及来自TCGA的32种癌症亚型分类中的19种。
在这些任务以及其他任务上,UNI表现出一致且显著的性能提升,超过表现第二好的编码器(REMEDIS或CTransPath)。作者推测UNI的性能归因于预提取特征的强表示质量,这在少数样本ROI和切片分类中使用类原型时可见。在弱监督范式中,罕见癌症类型在当前切片数据集中很少见且代表性不足,MI-SimpleShot使用UNI显示,注释四个幻灯片每类可以超过特定任务的MIL算法。总的来说,作者相信UNI和其他正在开发中的以视觉为中心的基础模型能够以创新的方式促进临床应用,这些应用通常需要比现在多几个数量级的数据。
与公共排行榜相比,作者认为UNI也代表了CPath中从特定任务模型开发向通用AI模型的转变。
除了在本研究中评估的34个临床任务之外,UNI的开箱即用性能与出版的其他作品的结果竞争,超过了经常端到端训练或精心设计的训练配方实现解决这些特定公共挑战的领先模型。
总的来说,作者的发现突出了拥有更好的预训练编码器与开发针对特定临床问题的特定任务模型的力量,作者希望这将改变CPath的研究方向,转向开发能够针对病理学中多样化的临床应用具有更大性能和灵活性的通用AI模型。遵循计算机视觉中自监督模型的传统命名法22,75,标签如“基础模型”可能会产生误导性的期望。
作者的研究存在一些局限性。
基于ViT-L架构,UNI缺乏解决CPath中密集预测任务的视觉特定偏差,作者注意到在SegPath中的细胞类型分割性能提升并不像观察到的其他任务那样显著。
作者设想随着更好的配方出现,ViT架构的适应性将会有所改善。作者的研究也没有评估DINOv2中的最佳表现ViT-Giant架构,这是一个更大的模型,可能在CPath中表现良好,但需要更多的计算资源进行预训练。尽管作者的研究组织了CPath中评估预训练模型最大的临床任务集合(据作者所知),其他临床任务,如细胞病理学或血液病理学,在作者的分析中没有得到体现。
由于作者的评估范围广泛,某些任务的小型(或缺失)验证集,超参数被固定,这遵循了CPath中的其他工作25,37,40,112,113。进一步的超参数调整和其他训练配方可能会进一步提高结果;然而,作者的评估协议旨在对预训练编码器骨架的表示质量进行排名。
在开发UNI的过程中,尽管Mass-100K被有意开发为与大多数公共组织学收藏不显著重叠,但如果同一模型被用于许多应用,特别是在其对不同人群产生不均衡影响的情况下,数据污染和图像获取偏差等问题仍需要进一步研究。114。UNI是CPath的一个单模态模型,意味着多模态能力,如跨模态检索和视觉问答等,仍然超出了其范围,作者在同时进行的工作115,116中探索了这些能力。
最后,UNI也仅是CPath的一个ROI级模型,大多数临床任务在病理学中是在幻灯片或患者水平进行的。未来的工作将专注于使用UNI作为自监督模型的构建块,并在解剖病理学中开发通用的幻灯片级AI。
四、计算硬件和软件
作者在本研究中使用了Python(v3.8.13)和PyTorch(v2.0.0,CUDA 11.7)(https://pytorch.org)进行所有实验和分析(除非另有说明),这些实验和分析可以使用开源库进行复制,如下所述。
通过DINOv2训练UNI时,作者修改了由Hugging Face(https://huggingface.co)维护的开源timm库(v0.9.2)中的视觉变换器实现作为编码器主干,并使用原始的DINOv2自监督学习算法(https://github.com/facebookresearch/dinov2)进行预训练,该算法使用4×8 80 GB NVIDIA A100 GPU节点(图形处理单元)进行多GPU、多节点训练,使用分布式数据并行(DDP)。
所有下游实验的其他计算都是在单个24 GB NVIDIA 3090 GPU上进行的。所有WSI处理都得到了OpenSlide(v4.3.1)、openslide-python(v1.2.0)和CLAM(https://github.com/mahmoodlab/CLAM)的支持。
作者使用Scikit-learn134(v1.2.1)的K-最近邻实现,以及LGSSL代码库(https://github.com/mbanani/lgssl)提供的逻辑回归实现和SimpleShot实现。
本研究中基准的其他视觉预训练编码器
- 带有ImageNet转移的ResNet-50(https://github.com/mahmoodlab/CLAM)
- CTransPath(https://github.com/Xiyue-Wang/TransPath)
- REMEDIS(https://github.com/google-research/medical-ai-research-foundations)
注意:REMEDIS需要满足数据使用协议,可以在PhysioNet网站(https://physionet.org/content/medical-ai-research-foundation)172,173上访问和提交。
对于多头注意力可视化,作者使用了HIPT代码库(https://github.com/mahmoodlab/HIPT)提供的可视化工具。
对于训练弱监督ABMIL模型,作者改编了CLAM代码库(https://github.com/mahmoodlab/CLAM)中的训练支架代码。
对于训练语义分割,作者使用基于detectron2(参考文献174)(v0.6)的原始Mask2Former实现,为了兼容性,需要以下较旧的包:Python(v3.8)和PyTorch(v1.9.0,CUDA 11.1)。
对于向UNI添加ViT-Adapter,作者将detectron2中的原始实现(https://github.com/czczup/ViT-Adapter)改编,使用Mask2Former进行训练。
作者使用Pillow(v9.3.0)和OpenCV-python进行基本图像处理任务。Matplotlib(v3.7.1)和Seaborn(v0.12.2)用于创建图表和图形。
版权归原作者 罗小罗同学 所有, 如有侵权,请联系我们删除。