无监督聚类方法的评价指标必须依赖于数据和聚类结果的内在属性,例如聚类的紧凑性和分离性,与外部知识的一致性,以及同一算法不同运行结果的稳定性。
本文将全面概述Scikit-Learn库中用于的聚类技术以及各种评估方法。
本文将分为2个部分,1、常见算法比较 2、聚类技术的各种评估方法
本文作为第一部分将介绍和比较各种聚类算法:
- K-Means
- Affinity Propagation
- Agglomerative Clustering
- Mean Shift Clustering
- Bisecting K-Means
- DBSCAN
- OPTICS
- BIRCH
首先我们生成一些数据,后面将使用这些数据作为聚类技术的输入。
importpandasaspd
importnumpyasnp
importseabornassns
importmatplotlib.pyplotasplt
#Set the number of samples and features
n_samples=1000
n_features=4
#Create an empty array to store the data
data=np.empty((n_samples, n_features))
#Generate random data for each feature
foriinrange(n_features):
data[:, i] =np.random.normal(size=n_samples)
#Create 5 clusters with different densities and centroids
cluster1=data[:200, :] +np.random.normal(size=(200, n_features), scale=0.5)
cluster2=data[200:400, :] +np.random.normal(size=(200, n_features), scale=1) +np.array([5,5,5,5])
cluster3=data[400:600, :] +np.random.normal(size=(200, n_features), scale=1.5) +np.array([-5,-5,-5,-5])
cluster4=data[600:800, :] +np.random.normal(size=(200, n_features), scale=2) +np.array([5,-5,5,-5])
cluster5=data[800:, :] +np.random.normal(size=(200, n_features), scale=2.5) +np.array([-5,5,-5,5])
#Combine the clusters into one dataset
X=np.concatenate((cluster1, cluster2, cluster3, cluster4, cluster5))
# Plot the data
plt.scatter(X[:, 0], X[:, 1])
plt.show()
结果如下:

我们将用特征值和簇ID创建一个DF。稍后在模型性能时将使用这些数据。
df=pd.DataFrame(X, columns=["feature_1", "feature_2", "feature_3", "feature_4"])
cluster_id=np.concatenate((np.zeros(200), np.ones(200), np.full(200, 2), np.full(200, 3), np.full(200, 4)))
df["cluster_id"] =cluster_id
df
现在我们将构建和可视化8个不同的聚类模型:
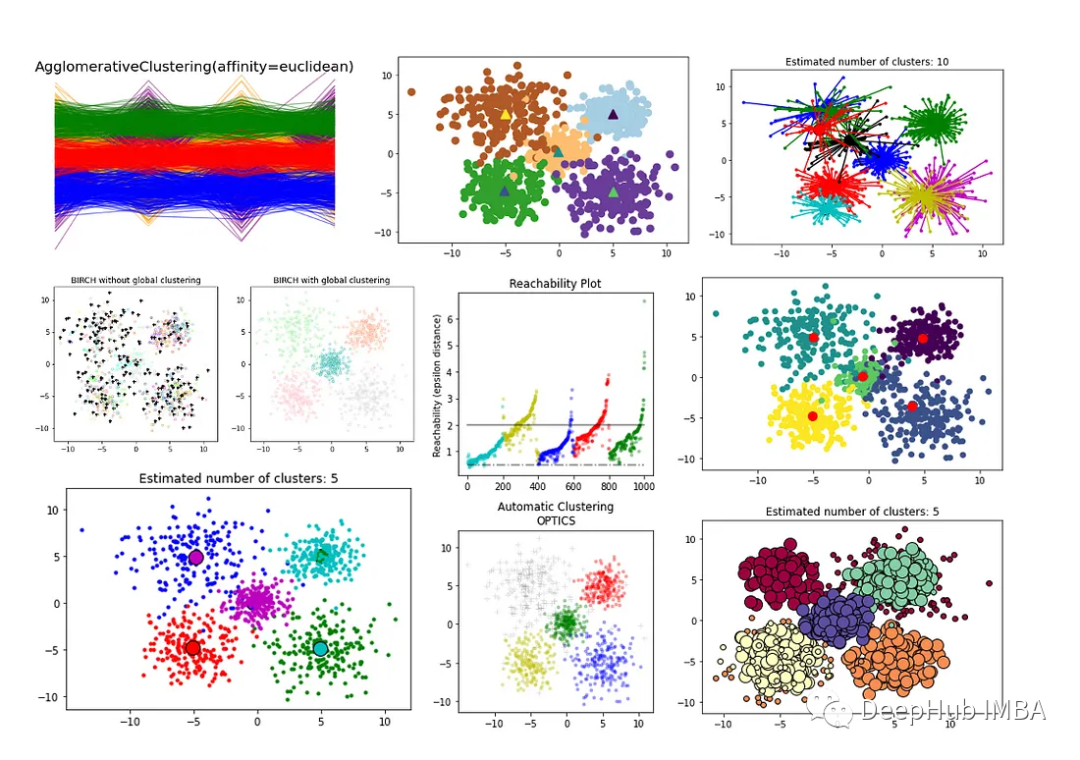
1、K-Means
K-Means聚类算法是一种常用的聚类算法,它将数据点分为K个簇,每个簇的中心点是其所有成员的平均值。K-Means算法的核心是迭代寻找最优的簇心位置,直到达到收敛状态。
K-Means算法的优点是简单易懂,计算速度较快,适用于大规模数据集。但是它也存在一些缺点,例如对于非球形簇的处理能力较差,容易受到初始簇心的选择影响,需要预先指定簇的数量K等。此外,当数据点之间存在噪声或者离群点时,K-Means算法可能会将它们分配到错误的簇中。
#K-Means
fromsklearn.clusterimportKMeans
#Define function:
kmeans=KMeans(n_clusters=5)
#Fit the model:
km=kmeans.fit(X)
km_labels=km.labels_
#Print results:
#print(kmeans.labels_)
#Visualise results:
plt.scatter(X[:, 0], X[:, 1],
c=kmeans.labels_,
s=70, cmap='Paired')
plt.scatter(kmeans.cluster_centers_[:, 0],
kmeans.cluster_centers_[:, 1],
marker='^', s=100, linewidth=2,
c=[0, 1, 2, 3, 4])
2、Affinity Propagation
Affinity Propagation是一种基于图论的聚类算法,旨在识别数据中的"exemplars"(代表点)和"clusters"(簇)。与K-Means等传统聚类算法不同,Affinity Propagation不需要事先指定聚类数目,也不需要随机初始化簇心,而是通过计算数据点之间的相似性得出最终的聚类结果。
Affinity Propagation算法的优点是不需要预先指定聚类数目,且能够处理非凸形状的簇。但是该算法的计算复杂度较高,需要大量的存储空间和计算资源,并且对于噪声点和离群点的处理能力较弱。
fromsklearn.clusterimportAffinityPropagation
#Fit the model:
af=AffinityPropagation(preference=-563, random_state=0).fit(X)
cluster_centers_indices=af.cluster_centers_indices_
af_labels=af.labels_
n_clusters_=len(cluster_centers_indices)
#Print number of clusters:
print(n_clusters_)
importmatplotlib.pyplotasplt
fromitertoolsimportcycle
plt.close("all")
plt.figure(1)
plt.clf()
colors=cycle("bgrcmykbgrcmykbgrcmykbgrcmyk")
fork, colinzip(range(n_clusters_), colors):
class_members=af_labels==k
cluster_center=X[cluster_centers_indices[k]]
plt.plot(X[class_members, 0], X[class_members, 1], col+".")
plt.plot(
cluster_center[0],
cluster_center[1],
"o",
markerfacecolor=col,
markeredgecolor="k",
markersize=14,
)
forxinX[class_members]:
plt.plot([cluster_center[0], x[0]], [cluster_center[1], x[1]], col)
plt.title("Estimated number of clusters: %d"%n_clusters_)
plt.show()

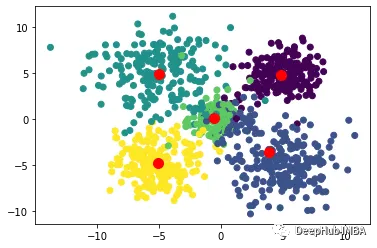
3、Agglomerative Clustering
凝聚层次聚类(Agglomerative Clustering)是一种自底向上的聚类算法,它将每个数据点视为一个初始簇,并将它们逐步合并成更大的簇,直到达到停止条件为止。在该算法中,每个数据点最初被视为一个单独的簇,然后逐步合并簇,直到所有数据点被合并为一个大簇。
Agglomerative Clustering算法的优点是适用于不同形状和大小的簇,且不需要事先指定聚类数目。此外,该算法也可以输出聚类层次结构,便于分析和可视化。缺点是计算复杂度较高,尤其是在处理大规模数据集时,需要消耗大量的计算资源和存储空间。此外,该算法对初始簇的选择也比较敏感,可能会导致不同的聚类结果。
fromsklearn.clusterimportAgglomerativeClustering
#Fit the model:
clustering=AgglomerativeClustering(n_clusters=5).fit(X)
AC_labels=clustering.labels_
n_clusters=clustering.n_clusters_
print("number of estimated clusters : %d"%clustering.n_clusters_)
# Plot clustering results
colors= ['purple', 'orange', 'green', 'blue', 'red']
forindex, metricinenumerate([#"cosine",
"euclidean",
#"cityblock"
]):
model=AgglomerativeClustering(
n_clusters=5, linkage="ward", affinity=metric
)
model.fit(X)
plt.figure()
plt.axes([0, 0, 1, 1])
forl, cinzip(np.arange(model.n_clusters), colors):
plt.plot(X[model.labels_==l].T, c=c, alpha=0.5)
plt.axis("tight")
plt.axis("off")
plt.suptitle("AgglomerativeClustering(affinity=%s)"%metric, size=20)
plt.show()

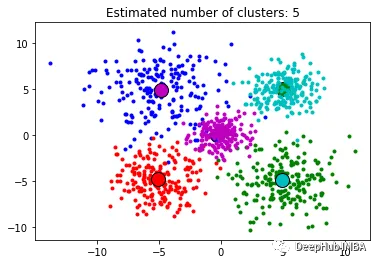
4、Mean Shift Clustering
Mean Shift Clustering是一种基于密度的非参数聚类算法,其基本思想是通过寻找数据点密度最大的位置(称为"局部最大值"或"高峰"),来识别数据中的簇。算法的核心是通过对每个数据点进行局部密度估计,并将密度估计的结果用于计算数据点移动的方向和距离。算法的核心是通过对每个数据点进行局部密度估计,并将密度估计的结果用于计算数据点移动的方向和距离。
Mean Shift Clustering算法的优点是不需要指定簇的数目,且对于形状复杂的簇也有很好的效果。算法还能够有效地处理噪声数据。他的缺点也是计算复杂度较高,尤其是在处理大规模数据集时,需要消耗大量的计算资源和存储空间,该算法还对初始参数的选择比较敏感,需要进行参数调整和优化。
fromsklearn.clusterimportMeanShift, estimate_bandwidth
# The following bandwidth can be automatically detected using
bandwidth=estimate_bandwidth(X, quantile=0.2, n_samples=100)
#Fit the model:
ms=MeanShift(bandwidth=bandwidth)
ms.fit(X)
MS_labels=ms.labels_
cluster_centers=ms.cluster_centers_
labels_unique=np.unique(labels)
n_clusters_=len(labels_unique)
print("number of estimated clusters : %d"%n_clusters_)
fromitertoolsimportcycle
plt.figure(1)
plt.clf()
colors=cycle("bgrcmykbgrcmykbgrcmykbgrcmyk")
fork, colinzip(range(n_clusters_), colors):
my_members=labels==k
cluster_center=cluster_centers[k]
plt.plot(X[my_members, 0], X[my_members, 1], col+".")
plt.plot(
cluster_center[0],
cluster_center[1],
"o",
markerfacecolor=col,
markeredgecolor="k",
markersize=14,
)
plt.title("Estimated number of clusters: %d"%n_clusters_)
plt.show()
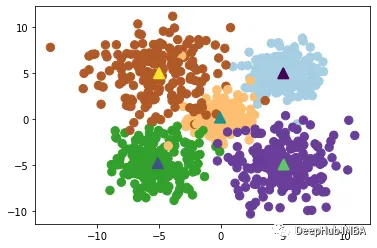
5、Bisecting K-Means
Bisecting K-Means是一种基于K-Means算法的层次聚类算法,其基本思想是将所有数据点划分为一个簇,然后将该簇分成两个子簇,并对每个子簇分别应用K-Means算法,重复执行这个过程,直到达到预定的聚类数目为止。
算法首先将所有数据点视为一个初始簇,然后对该簇应用K-Means算法,将该簇分成两个子簇,并计算每个子簇的误差平方和(SSE)。然后,选择误差平方和最大的子簇,并将其再次分成两个子簇,重复执行这个过程,直到达到预定的聚类数目为止。
Bisecting K-Means算法的优点是具有较高的准确性和稳定性,能够有效地处理大规模数据集,并且不需要指定初始聚类数目。该算法还能够输出聚类层次结构,便于分析和可视化。缺点是计算复杂度较高,尤其是在处理大规模数据集时,需要消耗大量的计算资源和存储空间。此外该算法对初始簇的选择也比较敏感,可能会导致不同的聚类结果。
fromsklearn.clusterimportBisectingKMeans
#Build and fit model:
bisect_means=BisectingKMeans(n_clusters=5).fit(X)
BKM_labels=bisect_means.labels_
#Print model attributes:
#print('Labels: ', bisect_means.labels_)
print('Number of clusters: ', bisect_means.n_clusters)
#Define varaibles to be included in scatterdot:
y=bisect_means.labels_
#print(y)
centers=bisect_means.cluster_centers_
# Visualize the results using a scatter plot
plt.scatter(X[:, 0], X[:, 1], c=y)
plt.scatter(centers[:, 0], centers[:, 1], c='r', s=100)
plt.show()
6、DBSCAN
DBSCAN (Density-Based Spatial Clustering of Applications with Noise)是一种基于密度的聚类算法,其可以有效地发现任意形状的簇,并能够处理噪声数据。DBSCAN算法的核心思想是:对于一个给定的数据点,如果它的密度达到一定的阈值,则它属于一个簇中;否则,它被视为噪声点。
DBSCAN算法的优点是能够自动识别簇的数目,并且对于任意形状的簇都有较好的效果。并且还能够有效地处理噪声数据,不需要预先指定簇的数目。缺点是对于密度差异较大的数据集,可能会导致聚类效果不佳,需要进行参数调整和优化。另外该算法对于高维数据集的效果也不如其他算法
fromsklearn.clusterimportDBSCAN
db=DBSCAN(eps=3, min_samples=10).fit(X)
DBSCAN_labels=db.labels_
# Number of clusters in labels, ignoring noise if present.
n_clusters_=len(set(labels)) - (1if-1inlabelselse0)
n_noise_=list(labels).count(-1)
print("Estimated number of clusters: %d"%n_clusters_)
print("Estimated number of noise points: %d"%n_noise_)
unique_labels=set(labels)
core_samples_mask=np.zeros_like(labels, dtype=bool)
core_samples_mask[db.core_sample_indices_] =True
colors= [plt.cm.Spectral(each) foreachinnp.linspace(0, 1, len(unique_labels))]
fork, colinzip(unique_labels, colors):
ifk==-1:
# Black used for noise.
col= [0, 0, 0, 1]
class_member_mask=labels==k
xy=X[class_member_mask&core_samples_mask]
plt.plot(
xy[:, 0],
xy[:, 1],
"o",
markerfacecolor=tuple(col),
markeredgecolor="k",
markersize=14,
)
xy=X[class_member_mask&~core_samples_mask]
plt.plot(
xy[:, -1],
xy[:, 1],
"o",
markerfacecolor=tuple(col),
markeredgecolor="k",
markersize=6,
)
plt.title(f"Estimated number of clusters: {n_clusters_}")
plt.show()

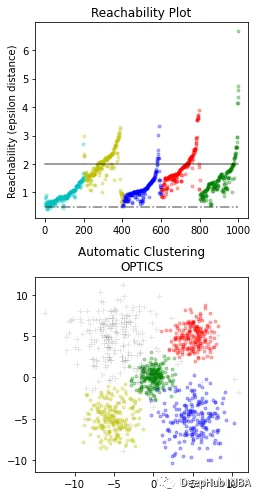
7、OPTICS
OPTICS(Ordering Points To Identify the Clustering Structure)是一种基于密度的聚类算法,其能够自动确定簇的数量,同时也可以发现任意形状的簇,并能够处理噪声数据。OPTICS算法的核心思想是:对于一个给定的数据点,通过计算它到其它点的距离,确定其在密度上的可达性,从而构建一个基于密度的距离图。然后,通过扫描该距离图,自动确定簇的数量,并对每个簇进行划分。
OPTICS算法的优点是能够自动确定簇的数量,并能够处理任意形状的簇,并能够有效地处理噪声数据。该算法还能够输出聚类层次结构,便于分析和可视化。缺点是计算复杂度较高,尤其是在处理大规模数据集时,需要消耗大量的计算资源和存储空间。另外就是该算法对于密度差异较大的数据集,可能会导致聚类效果不佳。
fromsklearn.clusterimportOPTICS
importmatplotlib.gridspecasgridspec
#Build OPTICS model:
clust=OPTICS(min_samples=3, min_cluster_size=100, metric='euclidean')
# Run the fit
clust.fit(X)
space=np.arange(len(X))
reachability=clust.reachability_[clust.ordering_]
OPTICS_labels=clust.labels_[clust.ordering_]
labels=clust.labels_[clust.ordering_]
plt.figure(figsize=(10, 7))
G=gridspec.GridSpec(2, 3)
ax1=plt.subplot(G[0, 0])
ax2=plt.subplot(G[1, 0])
# Reachability plot
colors= ["g.", "r.", "b.", "y.", "c."]
forklass, colorinzip(range(0, 5), colors):
Xk=space[labels==klass]
Rk=reachability[labels==klass]
ax1.plot(Xk, Rk, color, alpha=0.3)
ax1.plot(space[labels==-1], reachability[labels==-1], "k.", alpha=0.3)
ax1.set_ylabel("Reachability (epsilon distance)")
ax1.set_title("Reachability Plot")
# OPTICS
colors= ["g.", "r.", "b.", "y.", "c."]
forklass, colorinzip(range(0, 5), colors):
Xk=X[clust.labels_==klass]
ax2.plot(Xk[:, 0], Xk[:, 1], color, alpha=0.3)
ax2.plot(X[clust.labels_==-1, 0], X[clust.labels_==-1, 1], "k+", alpha=0.1)
ax2.set_title("Automatic Clustering\nOPTICS")
plt.tight_layout()
plt.show()
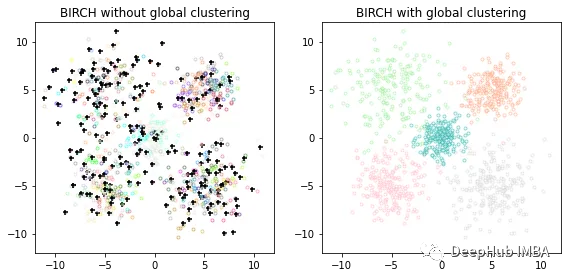
8、BIRCH
BIRCH(Balanced Iterative Reducing and Clustering using Hierarchies)是一种基于层次聚类的聚类算法,其可以快速地处理大规模数据集,并且对于任意形状的簇都有较好的效果。BIRCH算法的核心思想是:通过对数据集进行分级聚类,逐步减小数据规模,最终得到簇结构。BIRCH算法采用一种类似于B树的结构,称为CF树,它可以快速地插入和删除子簇,并且可以自动平衡,从而确保簇的质量和效率。
BIRCH算法的优点是能够快速处理大规模数据集,并且对于任意形状的簇都有较好的效果。该算法对于噪声数据和离群点也有较好的容错性。缺点是对于密度差异较大的数据集,可能会导致聚类效果不佳,对于高维数据集的效果也不如其他算法。
importmatplotlib.colorsascolors
fromsklearn.clusterimportBirch, MiniBatchKMeans
fromtimeimporttime
fromitertoolsimportcycle
# Use all colors that matplotlib provides by default.
colors_=cycle(colors.cnames.keys())
fig=plt.figure(figsize=(12, 4))
fig.subplots_adjust(left=0.04, right=0.98, bottom=0.1, top=0.9)
# Compute clustering with BIRCH with and without the final clustering step
# and plot.
birch_models= [
Birch(threshold=1.7, n_clusters=None),
Birch(threshold=1.7, n_clusters=5),
]
final_step= ["without global clustering", "with global clustering"]
forind, (birch_model, info) inenumerate(zip(birch_models, final_step)):
t=time()
birch_model.fit(X)
print("BIRCH %s as the final step took %0.2f seconds"% (info, (time() -t)))
# Plot result
labels=birch_model.labels_
centroids=birch_model.subcluster_centers_
n_clusters=np.unique(labels).size
print("n_clusters : %d"%n_clusters)
ax=fig.add_subplot(1, 3, ind+1)
forthis_centroid, k, colinzip(centroids, range(n_clusters), colors_):
mask=labels==k
ax.scatter(X[mask, 0], X[mask, 1], c="w", edgecolor=col, marker=".", alpha=0.5)
ifbirch_model.n_clustersisNone:
ax.scatter(this_centroid[0], this_centroid[1], marker="+", c="k", s=25)
ax.set_ylim([-12, 12])
ax.set_xlim([-12, 12])
ax.set_autoscaley_on(False)
ax.set_title("BIRCH %s"%info)
plt.show()
总结
上面就是我们常见的8个聚类算法,我们对他们进行了简单的说明和比较,并且用sklearn演示了如何使用,在下一篇文章中我们将介绍聚类模型评价方法。