Unet
- 参考文献:U-Net: Convolutional Networks for Biomedical Image Segmentation
- 作者:Olaf Ronneberger, Philipp Fischer, and Thomas Brox
什么是Unet模型
Unet是一个优秀的语义分割模型,其主要执行过程与其它语义分割模型类似。与CNN不同的之处在于CNN是图像级的分类,而unet是像素级的分类,其输出的是每个像素点的类别。
Unet损失函数
主要部分:softmax激活函数 + 带权重的交叉熵损失函数 + 权重计算函数
softmax激活函数
softmax激活函数将每个像素点的输入特征与权重做非线性叠加。每个像素点经过softmax的处理后产生的输出值个数等于标签里的类别数。softmax将每个像素点的输出值变换成值为正且和为1的概率分布,从而得到每个像素点中每个类的置信度。
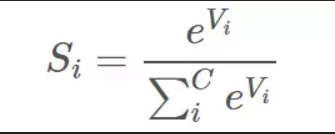
交叉熵损失函数
交叉熵损失函数:用来衡量两个概率分布差异的测量函数
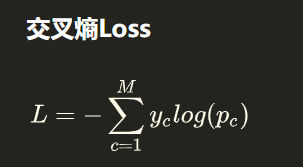
在上式中,yc表示样本的真实分布,其值要么为0,要么为1,Pc表示样本的预测分布。
本论文使用的是带边界权重的交叉熵损失函数:
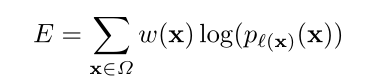
p是经softmax处理后的输出值;
l :Ω → {1, . . . , K},是每个像素的真实标签;
pl(x)(x):点x在对应的label给出的那个类别的输出的激活值。
w : Ω → R 是在训练过程中添加给每个像素的权重 。
权重计算函数
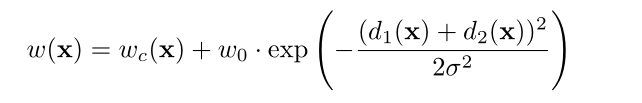
w(x)这个公式主要参考正态分布的式子。
wc(x)是预先计算每个地面真值分割,用来补偿训练数据集中每个类别的像素的不同频率;
d1是距离最近细胞边界的距离
d2是距离第二近细胞边界的距离
当d1、d2都等于0时,后面部分有最大值,且d1、d2越小,后面部分越大,即整个权重越大。当d1、d2越小,意味着越靠近细胞边界,细胞边界所占的权重越大,可以迫使网络学习。
权重的作用:权重可以调整图像中某个区域的重要程度,在计算损失的过程中,给两个细胞重合的边缘部分增加了损失的权重,以此让网络更加注重这类重合的边缘信息。
总结:首先使用softmax操作得到每个类的置信度,然后使用交叉熵来衡量预测和标号之间的差距。
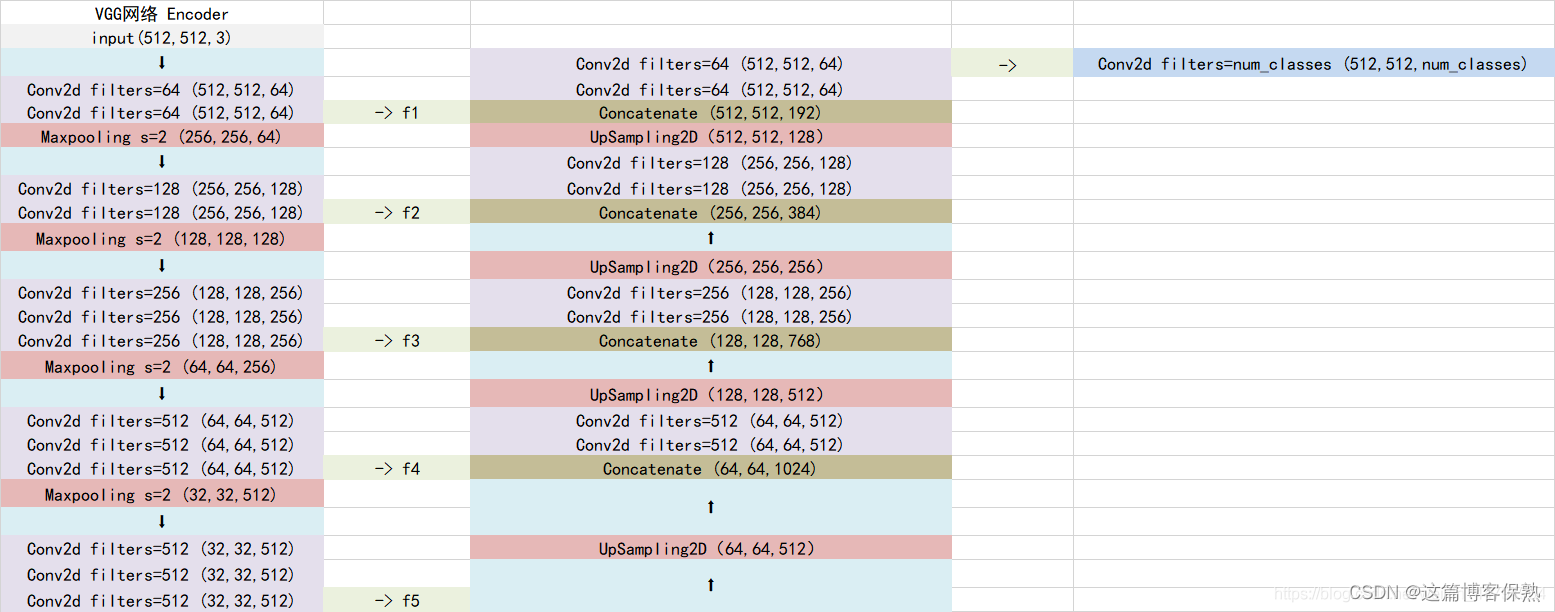
Unet主要结构
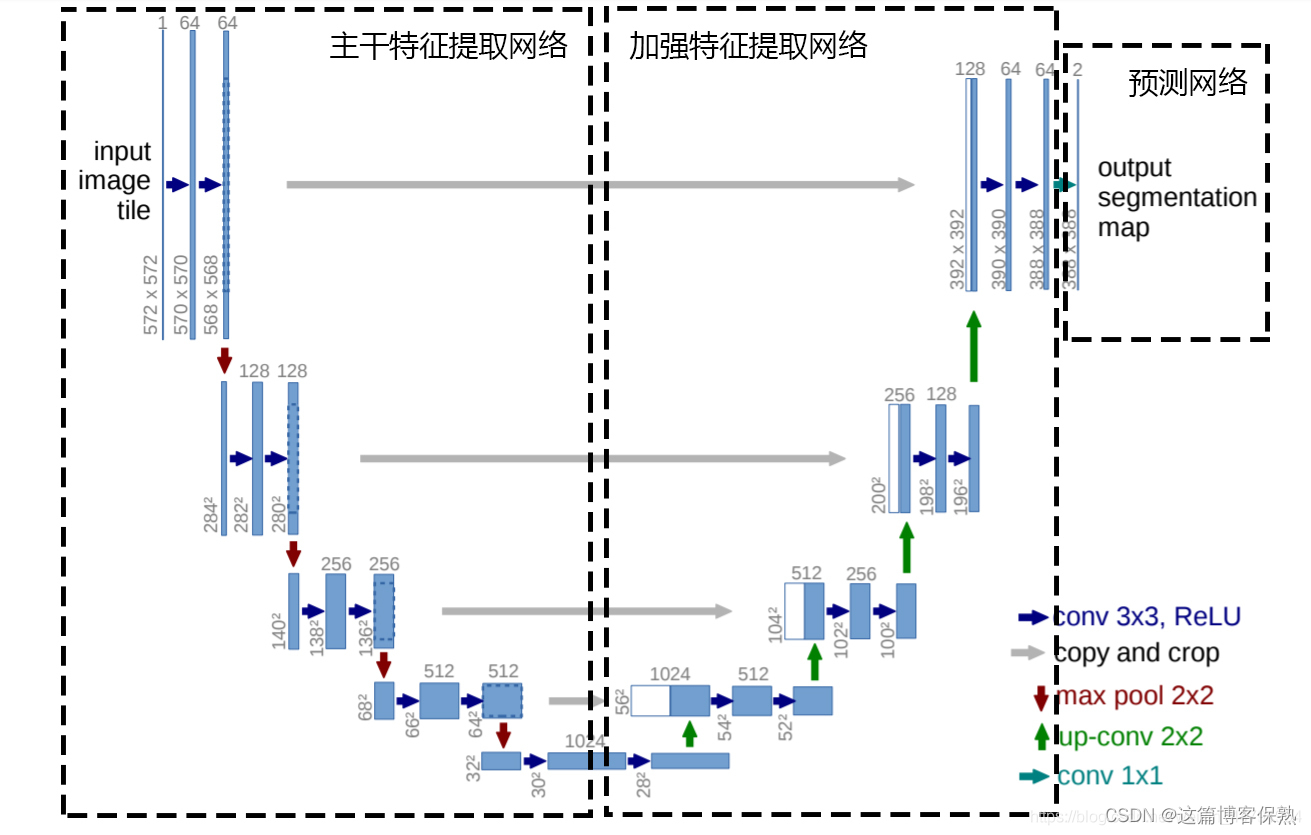
Unet可以分为三个部分,如上图所示:
第一部分是主干特征提取部分,我们可以利用主干部分获得一个又一个的特征层,Unet的主干特征提取部分与VGG相似,为卷积和最大池化的堆叠。利用主干特征提取部分我们可以获得五个初步有效特征层,在第二步中,我们会利用这五个有效特征层可以进行特征融合。
第二部分是加强特征提取部分,我们可以利用主干部分获取到的五个初步有效特征层进行上采样,并且进行特征融合,获得一个最终的,融合了所有特征的有效特征层。
第三部分是预测部分,我们会利用最终获得的最后一个有效特征层对每一个特征点进行分类,相当于对每一个像素点进行分类。
主干特征提取网络
Unet的主干特征提取部分由卷积层+最大池化层组成,整体结构与VGG类似。
 当输入的图像大小为512x512x3的时候,具体执行方式如下:
当输入的图像大小为512x512x3的时候,具体执行方式如下:
1、conv1:进行两次[3,3]的64通道的卷积,获得一个[512,512,64]的初步有效特征层,再进行2X2最大池化,获得一个[256,256,64]的特征层。
2、conv2:进行两次[3,3]的128通道的卷积,获得一个[256,256,128]的初步有效特征层,再进行2X2最大池化,获得一个[128,128,128]的特征层。
3、conv3:进行三次[3,3]的256通道的卷积,获得一个[128,128,256]的初步有效特征层,再进行2X2最大池化,获得一个[64,64,256]的特征层。
4、conv4:进行三次[3,3]的512通道的卷积,获得一个[64,64,512]的初步有效特征层,再进行2X2最大池化,获得一个[32,32,512]的特征层。
5、conv5:进行三次[3,3]的512通道的卷积,获得一个[32,32,512]的初步有效特征层。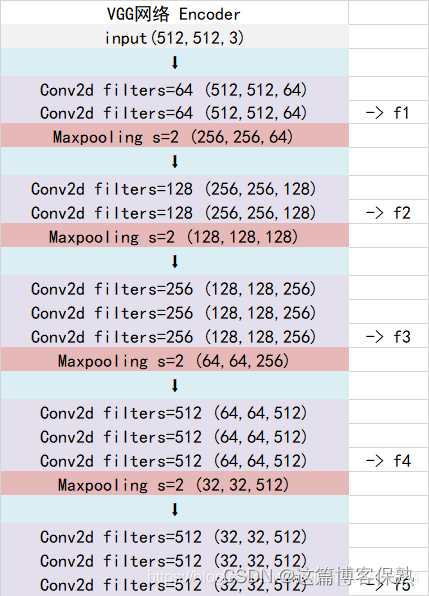
为什么是572x572,不是512x512?
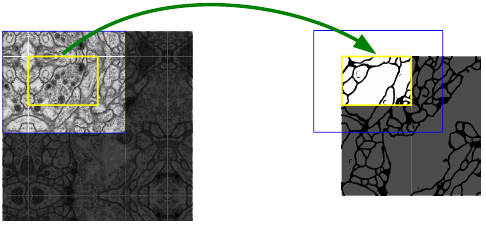
因为图像边界的图像块没有周围像素,卷积会使图像边缘处的信息丢失,因此要对周围像素采用了镜像扩充。
加强特征提取结构
利用第一步我们可以获得五个初步的有效特征层,在加强特征提取网络这里,我们会利用这五个初步的有效特征层进行特征融合,特征融合的方式就是对特征层进行上采样并且进行堆叠。
为了方便网络的构建与更好的通用性,我们的Unet和上图的Unet结构有些许不同,在上采样时直接进行两倍上采样再进行特征融合,最终获得的特征层和输入图片的高宽相同。
利用特征获得预测结果
利用特征获得预测结果的过程为:
利用一个1x1卷积核进行通道调整,将最终特征层的通道数调整成num_classes。
代码复现
数据集:ISBI
模型训练:
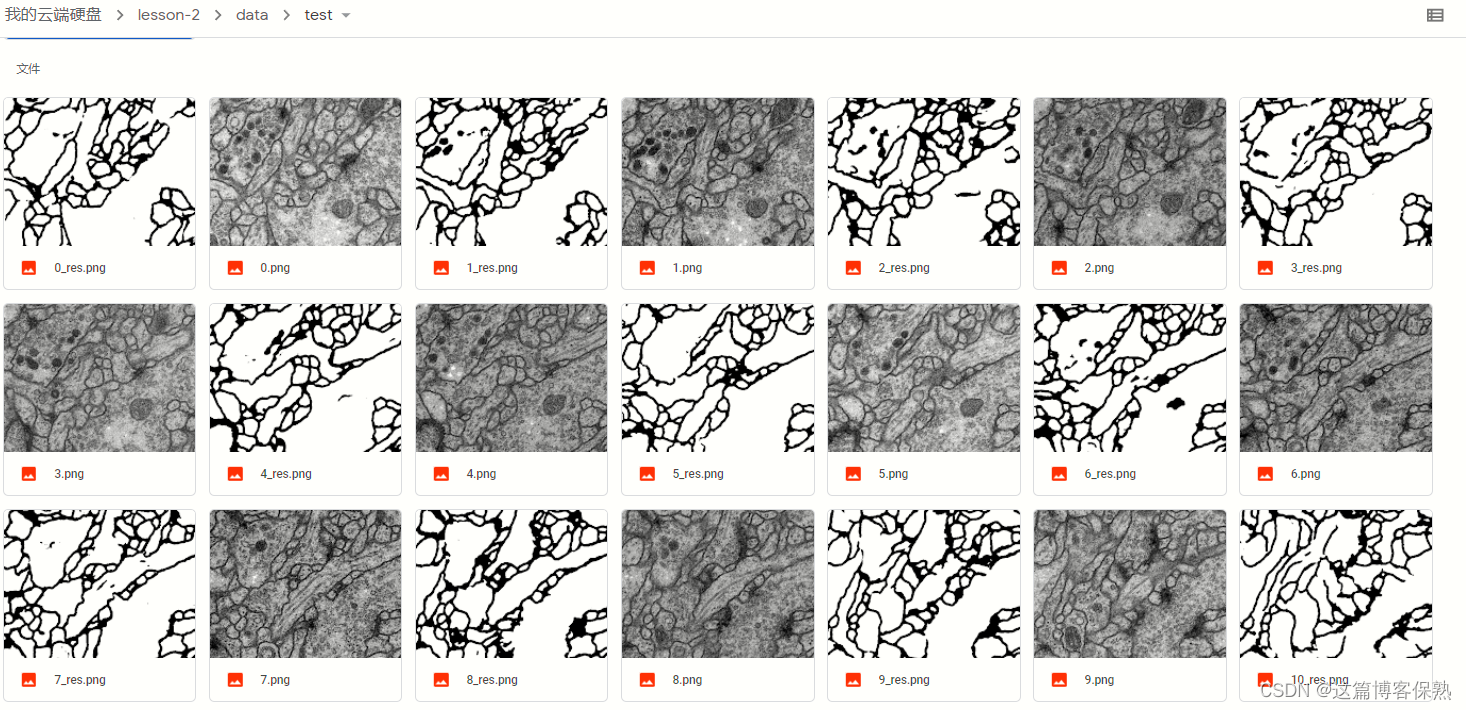
测试集:
版权归原作者 这篇博客保熟 所有, 如有侵权,请联系我们删除。
