小罗碎碎念
最近在整理24年发表在Nature Medicine上的病理AI文章,简单列了一个表。
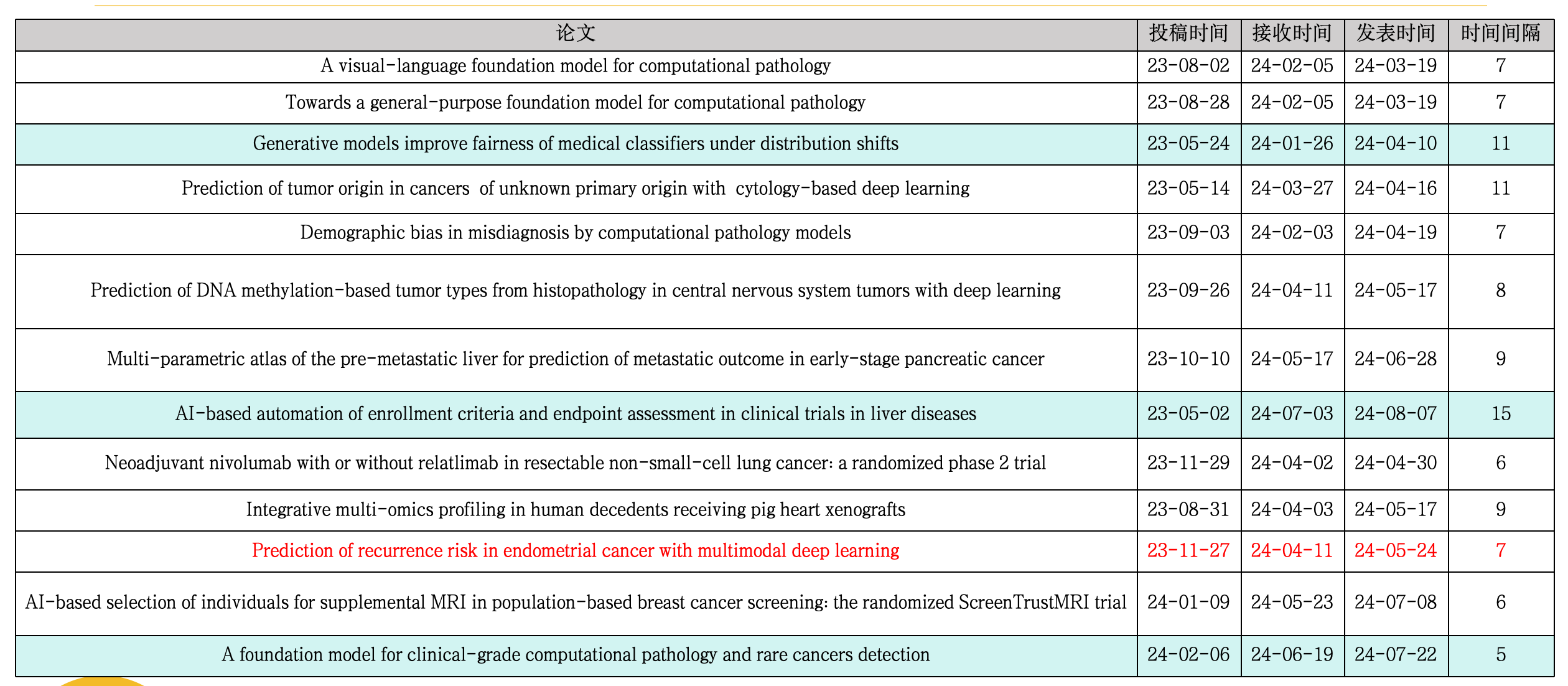
接下来我将按照先后顺序,系统的把这13篇文献分析完。其中底色做了填充的,代表商业公司在本论文中占据了一作或通讯。
本期推文介绍的模型是CONCH(CONtrastive learning from Captions for Histopathology)——一个专门为计算病理学开发的视觉语言基础模型。

作者类型作者姓名单位名称(中文)第一作者Ming Y. Lu哈佛医学院布里格姆和妇女医院病理科第一作者Bowen Chen哈佛医学院布里格姆和妇女医院病理科第一作者Drew F. K. Williamson哈佛医学院布里格姆和妇女医院病理科通讯作者Faisal Mahmood哈佛医学院布里格姆和妇女医院病理科
CONCH在14个不同的基准测试中进行了评估,这些测试涉及病理学图像和/或文本的多种下游任务,包括组织学图像分类、分割、描述生成以及文本到图像和图像到文本的检索。CONCH在这些任务上都取得了最先进的性能,并且可以在无监督微调的情况下直接改善其他模型的性能。
文章强调了数字病理学的快速发展以及深度学习在病理学任务中的应用,但也指出了模型训练中存在的标签稀缺问题,以及大多数模型仅利用图像数据的限制。CONCH模型通过结合图像和文本数据,模拟实际病理学家的工作流程,从而克服了这些限制。
研究结果表明,CONCH在多种病理学任务上都表现出色,包括零样本分类、检索和分割。此外,CONCH还展示了在罕见疾病分类和少量样本学习中的潜力。文章还讨论了CONCH在实际应用中的潜在价值,包括在病理学实践中的语言使用、教育和研究中的应用,以及在临床病理学中的潜在影响。最后,文章指出了当前视觉-语言预训练模型的局限性,并对未来的研究方向提出了建议。
一、CONCH的数据处理和模型训练流程
1-1:数据清洗流程
- 数据源:公开的医学文献和数据库、内部病理图像和报告以及公开数据集
- 对象检测器(Object detector):使用YOLOv5模型来识别和提取图像中的对象。这个过程是为了从文献中自动提取病理图像。
- 标题分割器(Caption splitter):使用一个经过预训练的生成式变换器(GPT)模型来将描述多个图像的标题分割成单独的子标题。这个步骤是为了处理包含多个子图像的图像面板。
- 匹配器(Matcher):使用一个在清理过的教育数据集上训练的CLIP模型来将分割后的子图像与子标题进行匹配。这个过程通过计算图像嵌入和文本嵌入在对齐潜在空间中的余弦相似性得分来完成。
- 数据集过滤:从原始的179万图像-文本对数据集中,首先排除了非人类样本,创建了一个117万人类样本的数据集。然后,进一步通过训练一个分类器来识别H&E(苏木精和伊红)染色,从而过滤出457,373对H&E样本,以及713,595对IHC + 特殊染色样本。
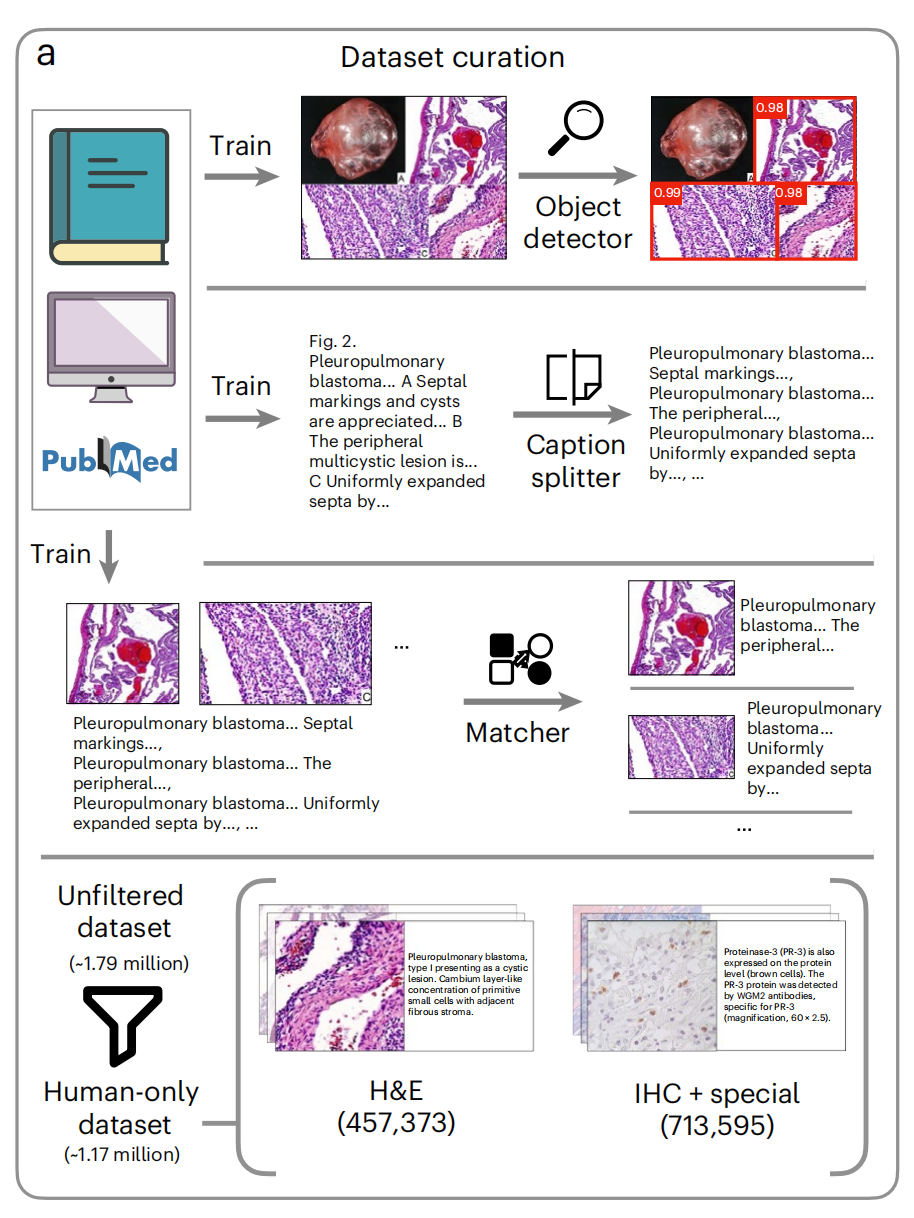
整体上,这个流程自动化地处理和过滤了大量病理图像和相关文本数据,为训练视觉-语言模型准备了一个清洗过的数据集。
1-2:数据集主题分布
这张图展示了数据集的特征描述,具体包括不同人体系统的图像数量和标题的字数分布。
- 人体系统图像数量:图中列出了多个人体系统,并为每个系统提供了图像数量。例如,胃肠道轨迹有最多的图像(121,209张),而眼睛的图像最少(11,569张)。
- 标题字数分布:右下角的直方图展示了标题字数的分布。图中有两个数据集的分布:PMC-Path(紫色)和EDU(蓝色)。可以看到,大多数标题的字数集中在较短的范围,随着字数增加,标题数量迅速下降。
- 数据集比较:从直方图可以观察到PMC-Path数据集的标题通常比EDU数据集的标题要长,这可能反映了两个数据源内容的差异。
- 图标表示:每个人体系统前都有一个简单的图标,帮助快速识别对应的系统。
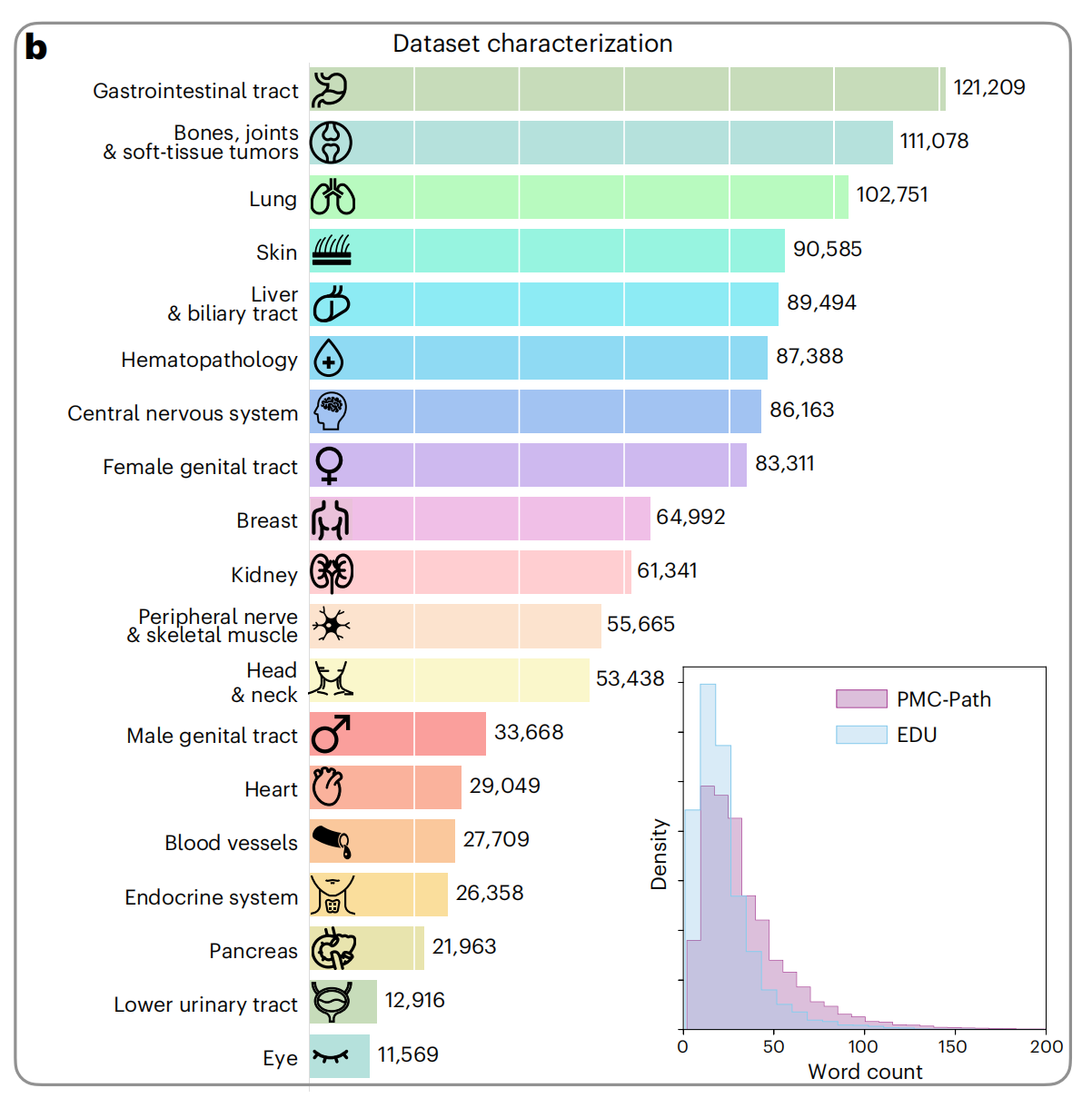
整体上,这个图表提供了数据集的概览,显示了不同人体系统的图像分布和标题的一般特征,对理解数据集的组成和特性很有帮助。
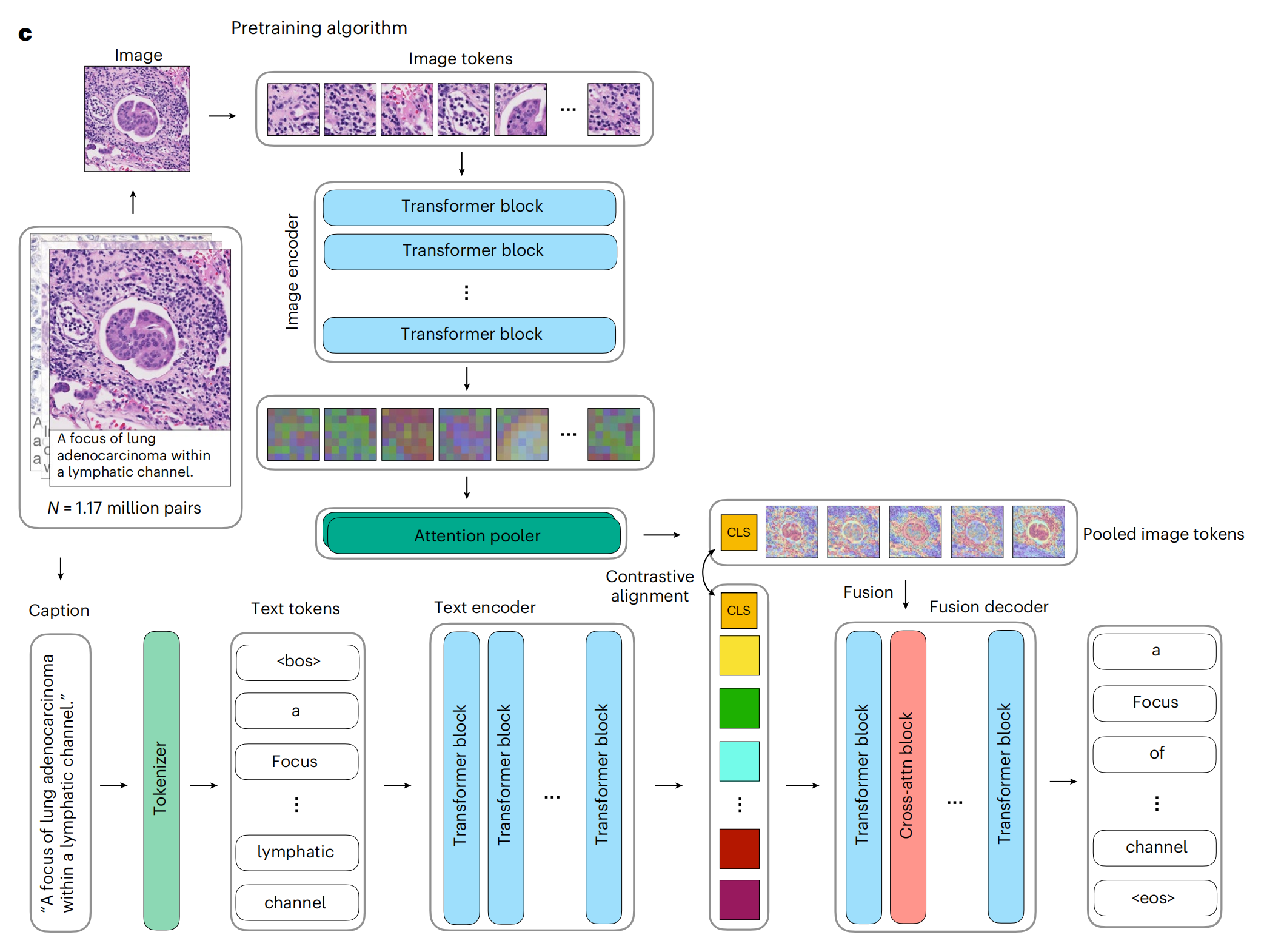
1-3:视觉-语言预训练设置
这张图展示了一个用于病理图像分析的预训练算法架构。
具体细节如下:
- 图像编码器(Image encoder):- 接收输入为病理图像。- 图像被分割成多个图像块(Image tokens),这些块随后被送入Transformer模块进行处理。- Transformer模块由多个Transformer块(Transformer block)组成,用于提取图像特征。
- 注意力池化器(Attention pooler):- 处理完所有图像块后,通过注意力池化器(Attention pooler)聚合信息,生成汇总的图像令牌(Pooled image tokens)。
- 文本编码器(Text encoder):- 接收与图像相对应的描述性文本(Caption)。- 文本被标记化(Tokenized),生成文本令牌(Text tokens)。- 这些文本令牌同样通过Transformer块进行处理,以提取文本特征。
- 对比性对齐(Contrastive alignment):- 目的是在图像和文本的特征空间中对齐它们,使得相关的图像和文本在特征空间中彼此靠近。- 通过最大化图像和文本嵌入的余弦相似性来实现。
- 融合解码器(Fusion decoder):- 结合图像和文本的特征,用于生成描述或执行其他多模态任务。- 包含交叉注意力(Cross-attention)机制,允许模型在生成过程中同时考虑图像和文本信息。
- 预训练数据:- 使用了117万图像-文本对进行预训练,这些数据对用于训练模型以理解图像内容和相关文本描述之间的关系。
整体上,这个架构通过结合图像和文本信息,利用Transformer架构和对比学习,来训练一个能够理解病理图像和对应文本描述的模型。这对于后续的细粒度分析、图像检索、自动报告生成等任务至关重要。
1-4:性能比较
这张雷达图展示了不同模型在多种下游任务上的性能表现。这些任务包括分类(Classification)、检索(Retrieval)和分割(Segmentation)。
图中比较了四种模型:CONCH(青色)、PLIP(黄色)、BiomedCLIP(蓝色)和OpenAICLIP(紫色)。
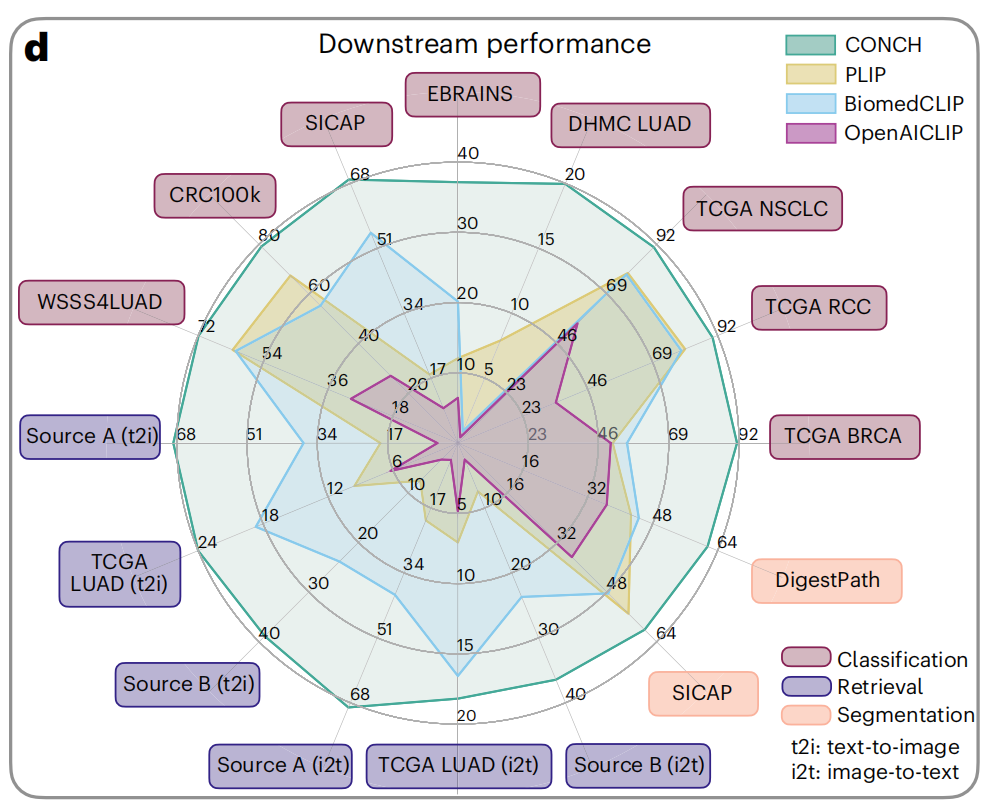
任务和数据集
- SICAP:前列腺癌的Gleason分级。
- EBRAINS:脑肿瘤的细粒度亚型分类。
- DHMC LUAD:肺腺癌的组织学模式分类。
- TCGA NSCLC:非小细胞肺癌的亚型分类。
- TCGA RCC:肾细胞癌的亚型分类。
- TCGA BRCA:乳腺癌的亚型分类。
- CRC100k:结直肠腺癌的组织分类。
- WSS4LUAD:肺腺癌的图像块分类。
- Source A (t2i) 和 **Source B (t2i)**:两个用于文本到图像和图像到文本检索的数据集。
- **TCGA LUAD (i2t)**:肺腺癌的图像块,用于图像到文本的检索。
性能指标
- 每个任务的得分范围从0到100,反映了模型在该任务上的性能。
- 图中每个轴代表一个任务,不同颜色的区域表示不同模型在该任务上的性能。
模型比较
- CONCH在大多数任务上都显示出较好的性能,特别是在分类任务上。
- PLIP和BiomedCLIP在某些任务上表现接近,但在其他任务上CONCH明显领先。
- OpenACLIP在某些任务上表现较弱,特别是在检索任务上。
二、零样本分类和监督分类的实验设置和结果
2-1:零样本分类示意图
这张图展示了一个用于区域感兴趣(ROI)级别的零样本(zero-shot)分类的模型架构。为每个类别构建一个提示(prompt),并根据共享嵌入空间中图像与提示嵌入的接近程度对图像进行分类。
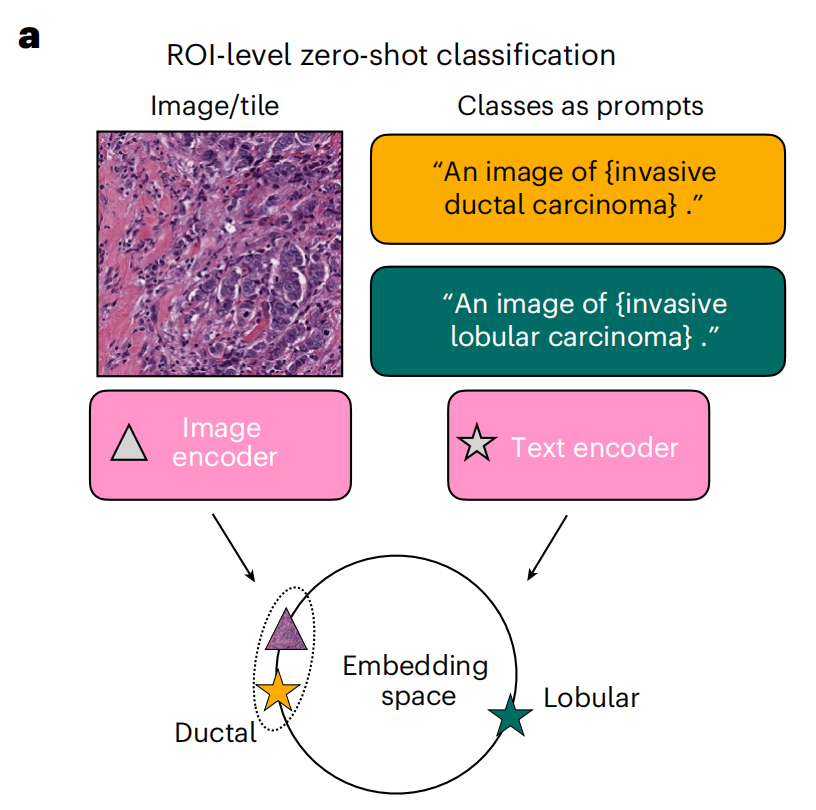
以下是对图中各部分的详细分析:
- 图像/图块(Image/tile):- 左上半部分显示了一个病理图像样本,对应roi区域。
- 类别作为提示(Classes as prompts):- 右上半部分显示了两个文本提示,分别描述了两种不同的癌症类型:“侵袭性导管癌(invasive ductal carcinoma)”和“侵袭性小叶癌(invasive lobular carcinoma)”。这些文本提示用于指导模型识别图像中的特征。
- 图像编码器(Image encoder):- 图像经过图像编码器处理,转换为可以被模型进一步处理的嵌入形式。
- 文本编码器(Text encoder):- 文本提示也通过文本编码器处理,转换为嵌入形式。文本编码器能够理解文本数据,并将其转换为模型可以处理的数值型特征。
- 嵌入空间(Embedding space):- 图像和文本编码器的输出在嵌入空间中相遇。这个空间是一个多维向量空间,其中每个维度代表数据的一个特征。- 在这个空间中,相关的图像和文本嵌入应该彼此靠近,这是通过对比学习实现的,目的是使模型能够理解图像内容和相关文本描述之间的关系。
- 分类过程:- 模型通过比较图像嵌入和文本嵌入在嵌入空间中的相似性来进行分类。- 根据图像嵌入和文本嵌入之间的距离或相似性得分,模型可以判断图像最可能属于的类别。
整体上,这个架构利用了深度学习中的转移学习和零样本学习的概念,允许模型在没有看到特定类别的样本的情况下,仅通过文本描述来识别和分类新的图像。这对于病理学图像分析尤其有用,因为获取大量标注数据往往是昂贵和耗时的。通过这种方法,模型可以更快地适应新的癌症类型或罕见病例,只需提供相应的文本描述。
2-2:对全玻片图像(WSIs)进行零样本分类
这张图展示了在全切片图像(WSI)级别上执行零样本(zero-shot)分类的流程,使用top-K池化技术汇总瓦片的相似度分数,形成切片级别的相似度分数,其中最高的分数对应于切片级别的预测。
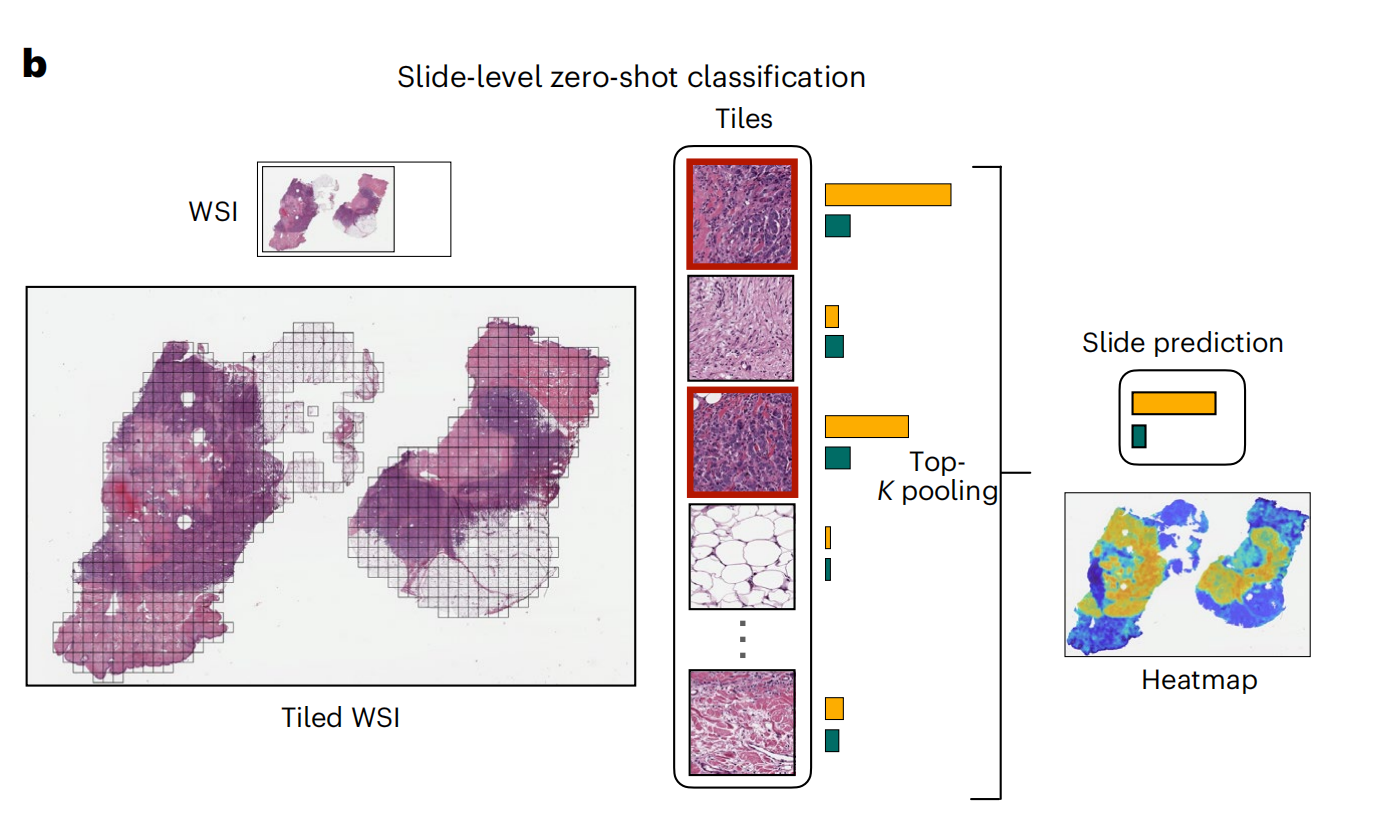
以下是对图中各部分的详细分析:
- 全切片图像(WSI):- 左上角显示了一个全切片图像的缩略图,这是病理学分析中常用的高分辨率图像,用于诊断和研究。
- 图块划分(Tiled WSI):- 左下角展示了全切片图像被划分成多个小图块(Tiles),每个图块可以单独进行分析。
- 图块分类(Tiles):- 中间部分显示了从全切片图像中提取的几个代表性图块,每个图块都用边界框标出。- 每个图块都通过模型进行分类,生成一个分类分数或嵌入向量。
- Top-K池化(Top-K pooling):- 为了进行全切片级别的分类,模型对所有图块的分类分数执行Top-K池化,这意味着从每个类别中选择最高K个分数,然后对这些分数进行汇总或平均,以生成整个切片的分类预测。
- 全切片预测(Slide prediction):- 根据Top-K池化的结果,模型为整个切片提供一个最终的分类预测结果。
- 热图(Heatmap):- 右下角展示了一个热图,它可视化了全切片图像上各个区域与特定类别关联的强度。- 热图中的颜色变化(例如从蓝色到黄色)表示了模型对于不同区域属于某一预测类别的信心程度,颜色越暖表示信心越高。
整体上,这个流程图说明了如何将全切片图像细分为可管理的图块,单独分析每个图块,然后汇总信息以进行整个切片的分类。这种方法允许模型在没有针对特定任务进行训练的情况下,利用其在大量数据上学到的通用特征提取和匹配能力,来预测新图像的类别。
这对于自动化病理图像分析尤其有用,因为它可以减少对大量标注数据的依赖,同时提供关于整个切片的详细信息。
2-3:零样本分类在下游亚型分类和分级任务上的性能
这张图展示了四种不同模型在多个下游任务上的零样本(zero-shot)性能。
这些模型包括CONCH(青色)、PLIP(黄色)、BiomedCLIP(蓝色)和OpenAICLIP(紫色)。横轴列出了不同的数据集和任务,包括TCGA BRCA、TCGA RCC、TCGA NSCLC、DHMC LUAD、SICAP、CRC100k和WSS4LUAD。
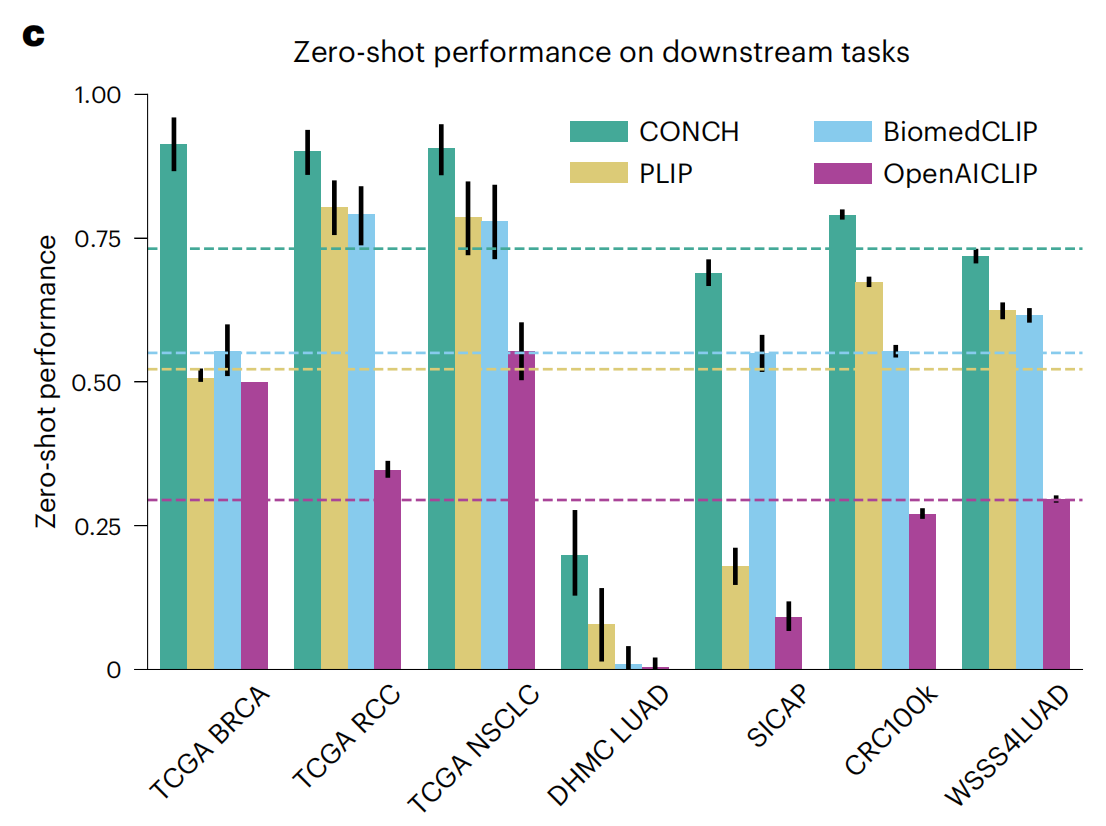
性能指标
- Y轴表示零样本性能,范围从0到1,其中1代表完美性能。
- 每种颜色的柱状图代表一个模型在特定任务上的性能。
- 每个任务上的柱状图都带有一个误差棒,表示性能的变动或不确定性。
模型比较
- CONCH(青色)在大多数任务上显示出最高的性能,特别是在TCGA BRCA、TCGA RCC和CRC100k数据集上。
- PLIP(黄色)在所有任务上表现中等,没有在任何特定任务上明显领先。
任务比较
- 在TCGA NSCLC任务上,所有模型的性能都相对较高,这表明这些模型能够较好地泛化到这些癌症类型的分类。
- 在DHMC LUAD任务上,所有模型的性能都明显下降,这可能是由于该数据集的特定挑战或模型对该数据集的泛化能力有限。
- SICAP任务上,CONCH和BiomedCLIP的性能相近,而PLIP和OpenAICLIP的性能较低。
- CRC100k和WSS4LUAD任务上,CONCH的性能明显优于其他模型,显示了它在这些任务上的优势。
2-4:监督评估模型的嵌入
这张图展示了不同模型在多个下游任务上的监督学习性能。这些任务包括TCGA BRCA、TCGA RCC、TCGA NSCLC、SICAP和CRC100k。
图中比较了五种模型:CONCH(青色)、PLIP(黄色)、BiomedCLIP(蓝色)、OpenAICLIP(紫色)以及ResNet50(绿色)。
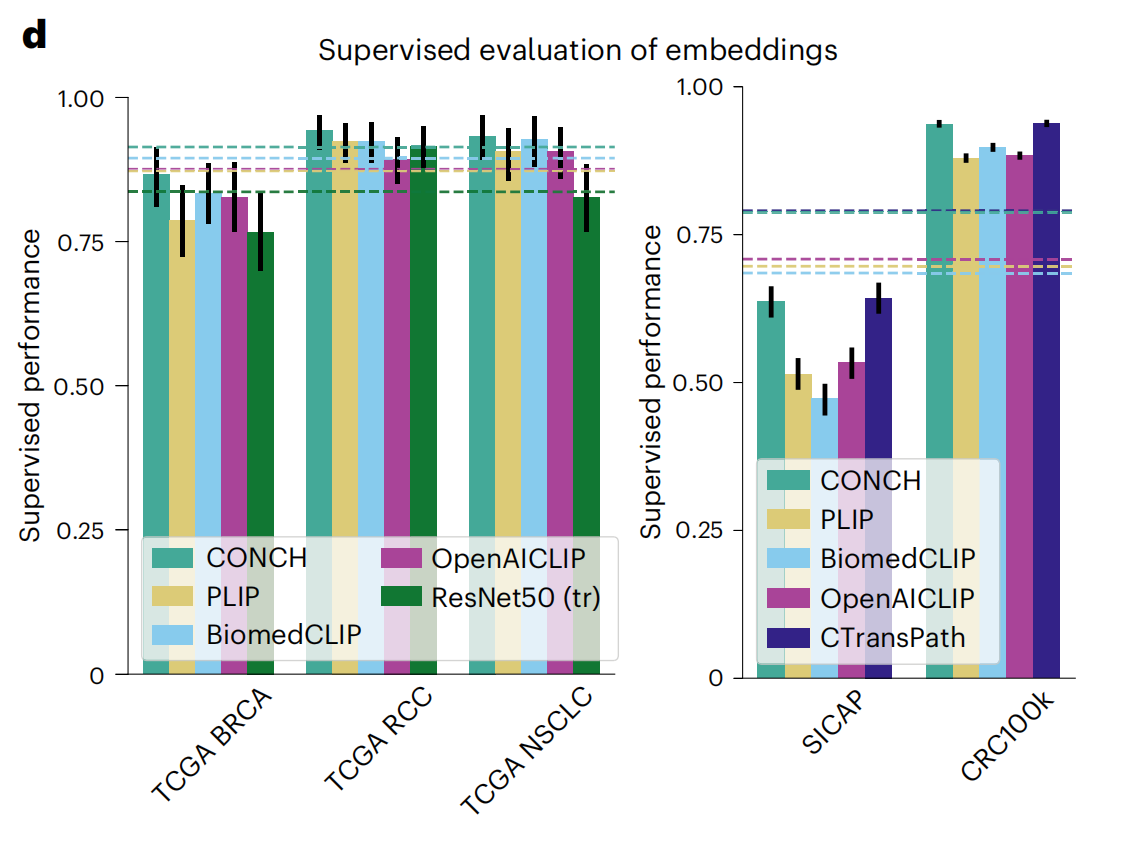
性能指标
- Y轴表示监督性能,范围从0到1,其中1代表完美性能。
- 每个任务上的柱状图代表一个模型的性能。
- 每个柱状图都带有一个误差棒,表示性能的变动或不确定性。
模型比较
- CONCH(青色)在所有任务上普遍显示出较高的性能,尤其是在TCGA RCC和CRC100k上。
- PLIP(黄色)在所有任务上表现中等,没有在任何特定任务上明显领先。
- BiomedCLIP(蓝色)在某些任务上,如TCGA NSCLC,表现接近或略逊于CONCH。
- OpenAICLIP(紫色)在大多数任务上表现较弱,尤其是在SICAP上。
- ResNet50(绿色)作为基线,通常表现低于其他视觉-语言模型,这强调了视觉-语言预训练的优势。
任务比较
- 在TCGA BRCA和TCGA RCC任务上,所有模型的性能都相对较高,这表明这些模型能够较好地泛化到这些癌症类型的分类。
- 在SICAP任务上,CONCH和CTranspath的性能相近,而PLIP和OpenACLIP的性能较低。
2-5:病理学家标注的IDC、对应的热图和选定的瓦片高倍观察
这张图由三个部分组成,展示了病理学图像分析中的热图和示例感兴趣区域(ROIs)。
由下图可知,标注的图像与高相似度区域之间有很好的一致性,高相似度区域内的瓦片展示了典型的IDC形态,而低相似度区域内则展示了乳腺的基质或其他正常成分。
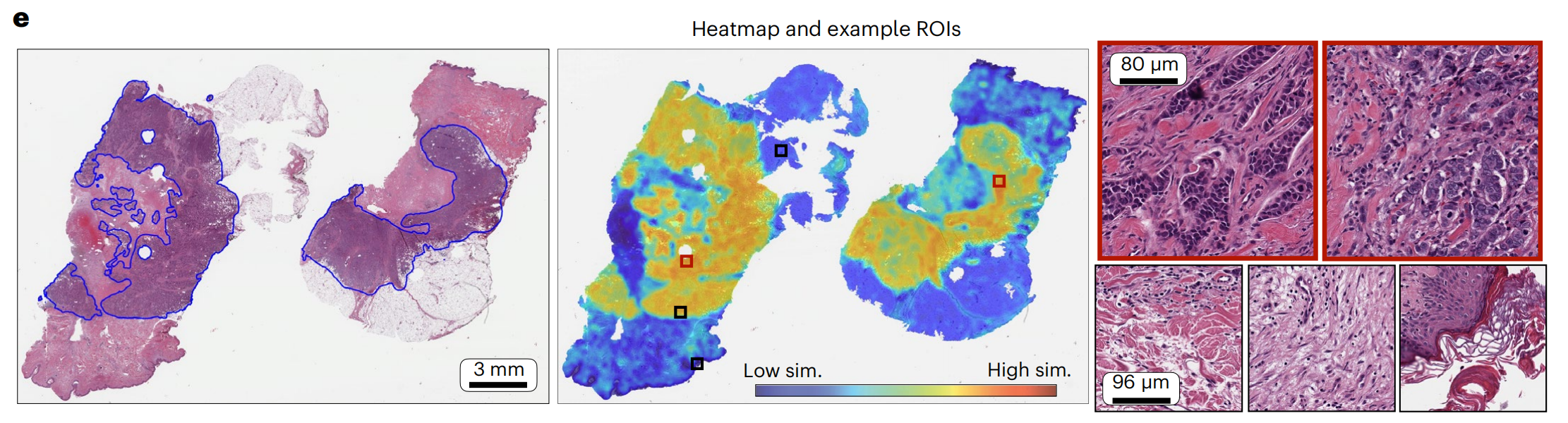
- 全切片图像(WSI):- 左图展示了两张手动标注的切片
- 热图(Heatmap):- 中间图显示了与全切片图像相对应的热图,颜色表示了模型对于图像中各个区域的分类信心程度。- 颜色从蓝色(低相似性)到红色(高相似性)变化,表示模型对于该区域属于特定类别(如癌症)的信心程度。- 可以看到颜色较暖的区域,与手动勾画的区域基本一致
- 示例感兴趣区域(Example ROIs):- 右图显示了从热图中选取的两个示例ROIs的放大图。- 这些放大的区域提供了更详细的组织结构视图,帮助病理学家进一步分析和诊断。- 标注的尺度(如80 μm和96 μm)提供了这些区域的实际大小信息。
整体上,这种热图方法允许研究者和病理学家快速识别和定位病理学图像中的关键区域,从而更有效地进行诊断和分析。通过将全切片图像、热图和示例ROIs结合起来,这张图提供了一个全面的视觉工具,用于理解和解释病理学图像数据。
三、零样本跨模态检索的性能评估
3-1:跨模态检索性能评估
这张图展示了不同模型在跨模态检索任务中的性能,具体包括文本到图像(Text-to-image)和图像到文本(Image-to-text)的检索。
图中比较了四种模型:CONCH(青色)、PLIP(黄色)、BiomedCLIP(蓝色)和OpenAICLIP(紫色)。
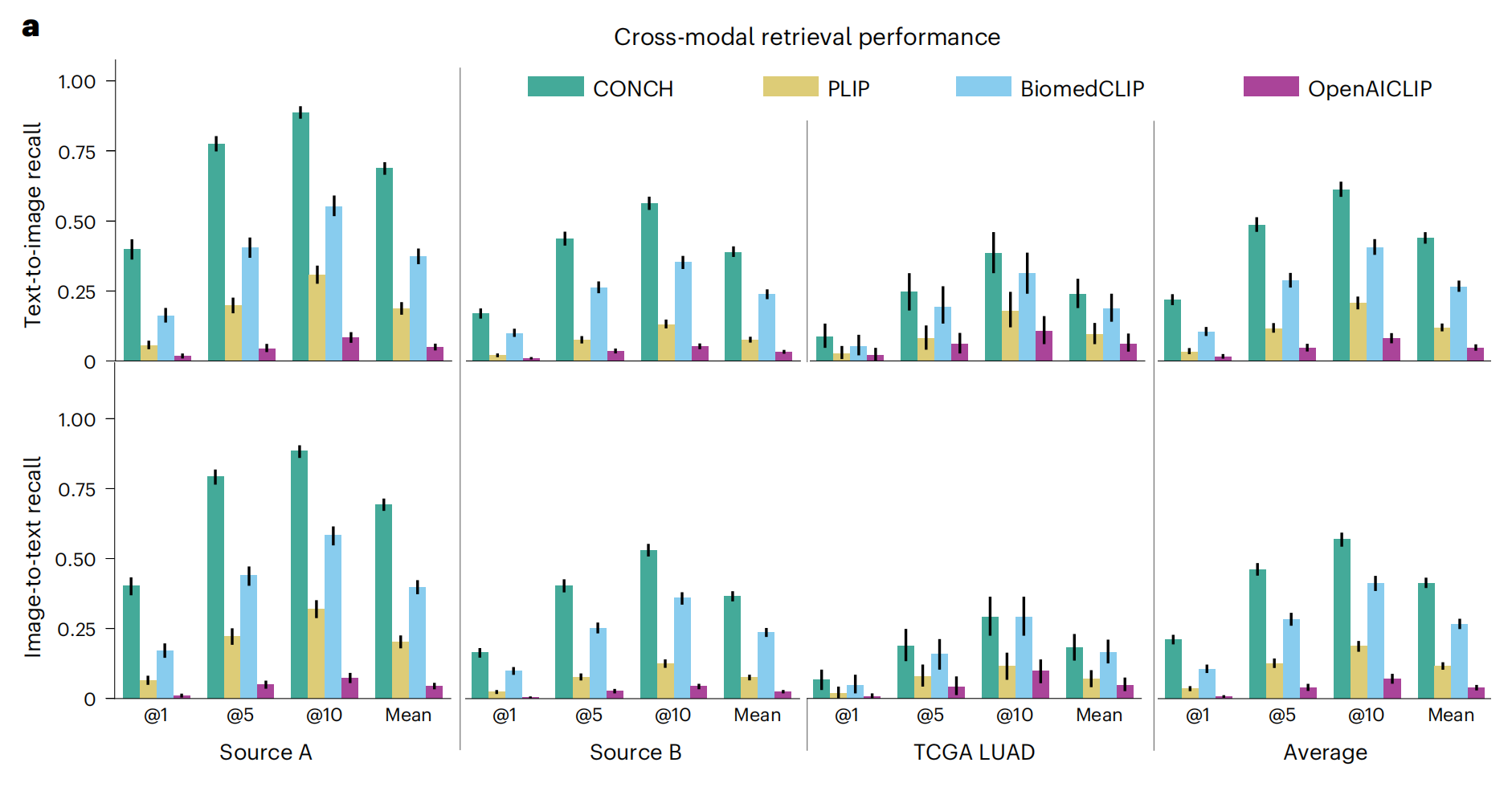
性能指标
- Y轴表示检索性能,采用召回率(recall)作为衡量标准,范围从0到1,其中1代表完美检索性能。
- 图中每个柱状图代表一个模型在一个特定数据集上的性能。
- 每个柱状图都带有一个误差棒,表示性能的变动或不确定性。
模型比较
- CONCH(青色)在多数任务上显示出较高的性能,尤其是在Source A和Source B的文本到图像检索任务上。
- PLIP(黄色)在所有任务上表现中等,没有在任何特定任务上明显领先。
- BiomedCLIP(蓝色)在某些任务上,如TCGA LUAD的图像到文本检索,表现接近或略逊于CONCH。
- OpenAICLIP(紫色)在大多数任务上表现较弱,尤其是在Source A和Source B的文本到图像检索任务上。
任务比较
- 在Source A和Source B数据集上,CONCH在文本到图像检索任务上的性能明显高于其他模型,表明它在理解文本描述和检索相关图像方面具有较强的能力。
- 在TCGA LUAD数据集上,CONCH在图像到文本检索任务上的性能也优于其他模型,显示了它在将图像内容转换为文本描述方面的优势。
- 平均值(Average)显示了模型在所有任务上的平均性能,CONCH再次显示出最高的平均检索性能。
3-2:零样本图像到文本检索的示意图
这张图展示了跨模态检索的概念和流程,用于在病理学图像分析中根据文本描述检索相应的图像,或根据图像检索相应的文本描述。
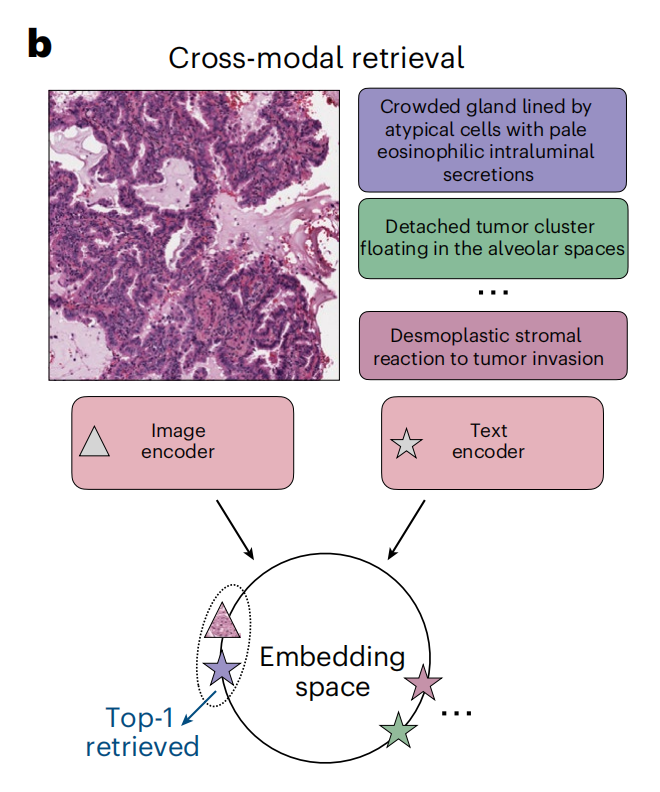
以下是对图中各部分的详细分析:
- 文本描述(Text):- 右侧列出了与图像相关的文本描述,包括: - “Crowded gland lined by atypical cells with pale eosinophilic intraluminal secretions”(由非典型细胞排列的拥挤腺体,内部有淡染的分泌物)- “Detached tumor cluster floating in the alveolar spaces”(在肺泡空间中漂浮的脱离肿瘤团块)- “Desmoplastic stromal reaction to tumor invasion”(对肿瘤侵入的间质性基质反应)- 这些描述用于训练模型识别图像中的特征。
- 编码器(Encoders):- 图像编码器(Image encoder):处理输入的病理图像,将其转换为嵌入向量,以便模型可以理解和处理。- 文本编码器(Text encoder):处理文本描述,将其转换为嵌入量,模型可以通过这些嵌入量理解文本内容。
- 嵌入空间(Embedding space):- 图像和文本的嵌入被映射到一个共同的嵌入空间中,这个空间允许模型比较图像和文本的相似性。- 在这个空间中,相关的图像和文本嵌入应该彼此靠近,模型可以据此进行检索。
- 检索过程(Retrieval):- 模型在嵌入空间中找到与输入文本描述最匹配的图像,或找到与输入图像最匹配的文本描述。- 图中显示了“Top-1 retrieved”(检索到的顶部结果),意味着模型为给定的查询找到了最相关的图像。
整体上,这个图示意了如何使用深度学习模型进行跨模态检索,在病理学图像和文本描述之间建立联系,从而实现精确的检索功能。这种技术在自动化病理图像分析和辅助诊断中具有重要应用价值。
3-3:TCGA LUAD数据集的检索示例
这张图展示了根据特定文本提示检索到的病理图像示例,这些图像与“肺腺癌(lung adenocarcinoma)”相关联。
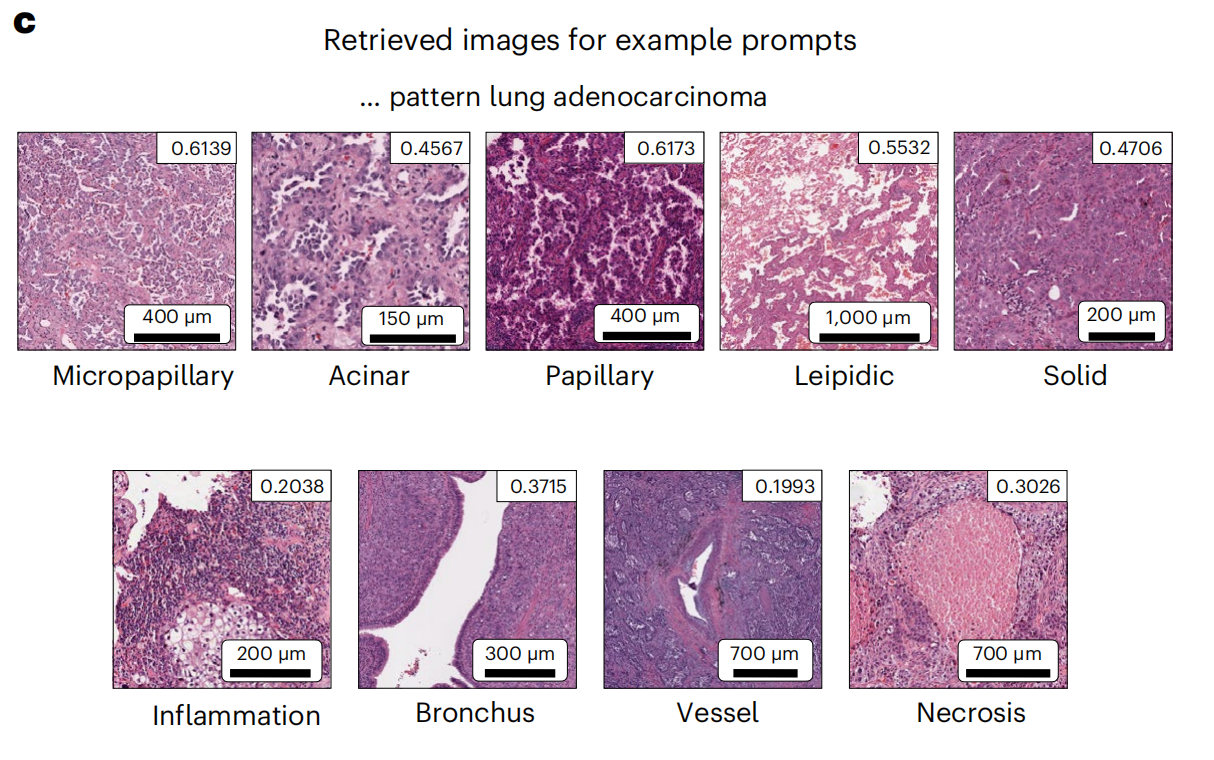
每个图像都附有一个分数,表示与文本提示的匹配程度,以及图像的尺度条,显示了图像实际大小。以下是对图中内容的详细分析:
- Micropapillary(微乳头状):- 分数:0.6139- 尺度:400微米(μm)
- Acinar(腺泡状):- 分数:0.4567- 尺度:150微米(μm)
- Papillary(乳头状):- 分数:0.6173- 尺度:400微米(μm)
- Leipidic(类脂质):- 分数:0.5532- 尺度:1000微米(μm)
- Solid(实性):- 分数:0.4706- 尺度:200微米(μm)
- Inflammation(炎症):- 分数:0.2038- 尺度:200微米(μm)
- Bronchus(支气管):- 分数:0.3715- 尺度:300微米(μm)
- Vessel(血管):- 分数:0.1993- 尺度:700微米(μm)
- Necrosis(坏死):- 分数:0.3026- 尺度:700微米(μm)
分析
- 这些图像是根据与“肺腺癌”相关模式的文本提示进行检索得到的,分数反映了图像与文本提示之间的相似度。
- 分数越高,表示图像与文本提示的相关性越高。
- 图像的尺度提供了图像在显微镜下的实际大小,有助于理解图像中特征的可见度和细节水平。
- 检索结果包括了多种病理学特征,如微乳头状、腺泡状、乳头状、类脂质、实性、炎症、支气管、血管和坏死,这些都是在肺癌病理诊断中可能会观察到的特征。
四、预训练数据集中不同类别标题内容的定性可视化分析
这张图是一个词云,展示了与病理学相关的不同主题和概念。
词云中词语的大小通常表示其在数据集中出现的频率或重要性,较大的词语意味着它们在数据集中更为常见或关键。

以下是对图中主要内容的分析:
- 主要主题:- 图中包含了多个与病理学相关的主题,如“肺(lung)”、“肝(liver)”、“皮肤(skin)”、“胃肠道(gastrointestinal tract)”等。
- 常见疾病和概念:- 词云中突出了一些常见的疾病和病理学概念,例如“癌症(cancer)”、“腺泡(acinar)”、“乳头状(papillary)”、“类脂质(leipidic)”、“炎症(inflammation)”、“坏死(necrosis)”等。
- 人体系统和部位:- 多个人体系统和部位被提及,如“皮肤(skin)”、“胃肠道(gastrointestinal tract)”、“肝和胆道(liver and biliary tract)”、“骨、关节和软组织(bone, joints, soft tissue)”等。
- 数据集大小:- 图中还提供了一些数据集的大小,如“所有类别(1,170,647)”、“胃肠道轨迹(121,209)”、“皮肤(90,585)”等,这指的是与这些主题相关的图像或文本数据对的数量。
- 颜色和分组:- 不同颜色用于区分不同的主题或概念群,帮助视觉化地组织信息。例如,“胃肠道轨迹”、“骨、关节和软组织”、“肺”等每个都有自己独特的颜色。
- 细节和模式:- 一些词语描述了病理学图像中可能观察到的特定模式或特征,如“微乳头状(micropapillary)”、“实性(solid)”、“类脂质(leipidic)”等。
整体上,这张词云图提供了一个视觉化的方式来概览病理学数据集中的关键主题和概念,帮助理解数据集的组成和研究的重点领域。通过这种可视化,研究者和医生可以快速识别出在病理学图像分析中常见的诊断类型和重要的组织学特征。
五、CONCH模型在肾细胞癌病理图像上的热力图分析
这张图展示了病理图像分析中的一个具体例子,包括了整体图像、热图和几个放大的感兴趣区域(ROIs)。
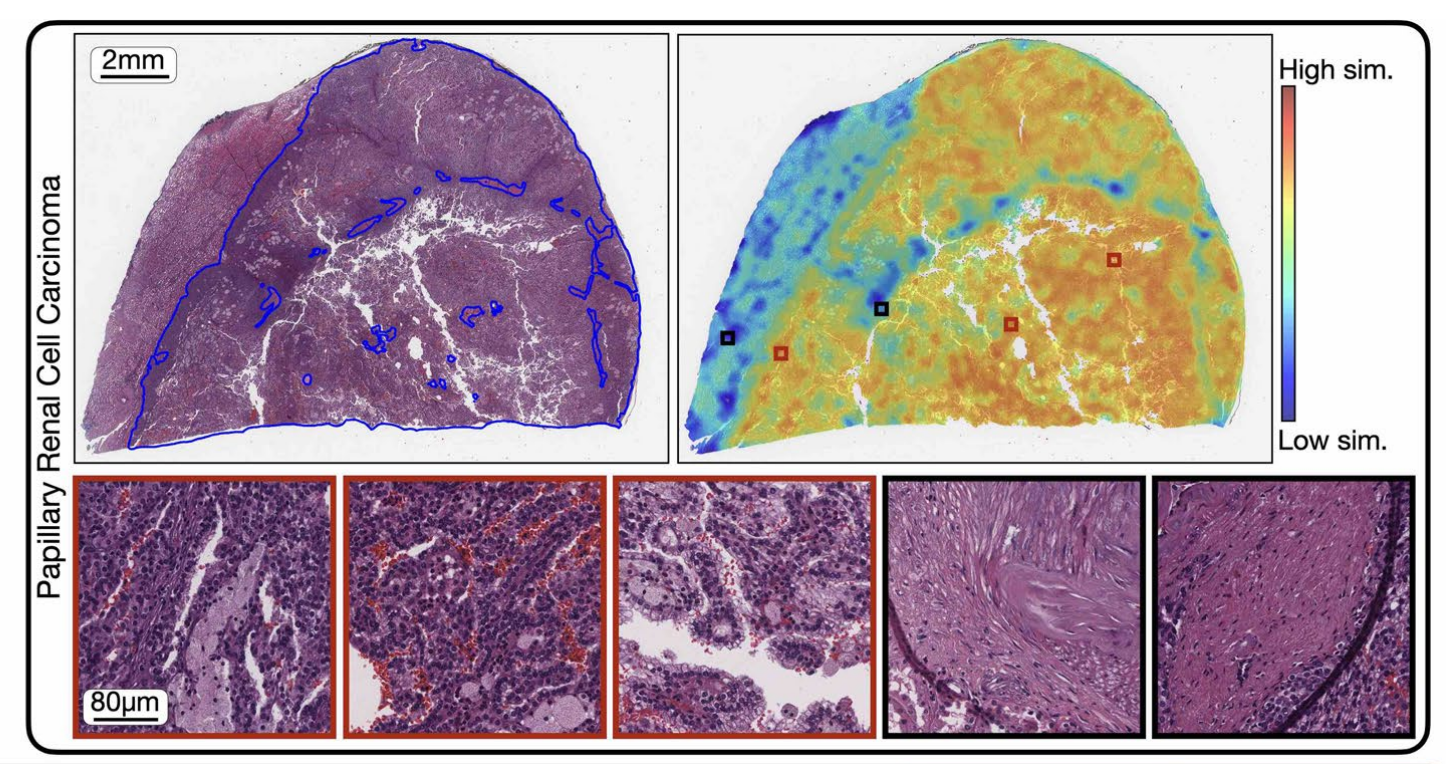
以下是对图中各部分的详细分析:
- 整体图像(Whole Slide Image): - 左上角显示了一个标记为“Papillary Renal Cell Carcinoma”(乳头状肾细胞癌)的全切片图像。- 图像中用蓝色轮廓线标出了肿瘤区域。- 左上角有一个尺度标记,指示图像大小为2毫米。
- 热图(Heatmap): - 右侧显示了与全切片图像相对应的热图,颜色表示了模型对于图像中各个区域的分类信心程度。- 颜色从蓝色(低相似性)到红色(高相似性)变化,表示模型对于该区域属于特定类别(如癌症)的信心程度。- 热图中有几个用黑色方块标出的区域,可能表示模型识别出的关键特征区域。
- 感兴趣区域(ROIs): - 下方显示了从热图中选取的三个感兴趣区域的放大图像,提供了更详细的组织结构视图。- 这些放大的区域提供了对肿瘤特征的更详细观察,如细胞形态、组织结构和可能的病理变化。
整体上,这种可视化方法允许研究者和病理学家快速识别和定位病理图像中的关键区域,从而更有效地进行诊断和分析。通过结合全切片图像、热图和示例ROIs,这张图提供了一个全面的视觉工具,用于理解和解释病理图像数据。
六、CONCH模型在图像字幕生成任务上的性能结果
6-1:图像字幕生成性能
这张图展示了三个不同模型在图像描述生成任务上的性能比较,使用的评估指标是METEOR和ROUGE。
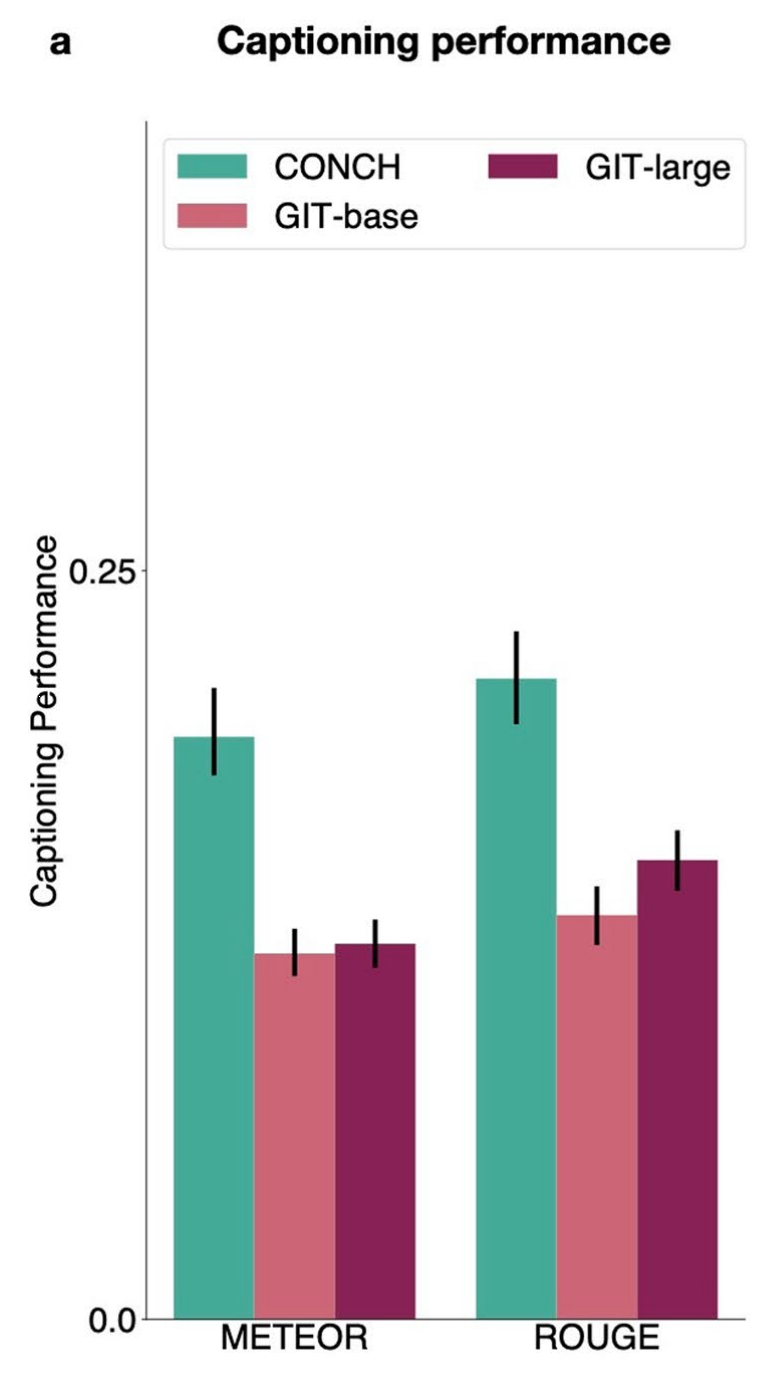
以下是对图中内容的详细分析:
- 模型: - CONCH(青色)- GIT-large(紫色)- GIT-base(红色)
- 评估指标: - METEOR:一种评估机器翻译质量的自动度量,考虑了同义词和词形变化,值越高表示生成的描述与真实描述越接近。- ROUGE:基于n-gram的评估方法,计算生成描述和真实描述之间的重叠程度,值越高表示两者越相似。
- 误差棒: - 每个柱状图上的误差棒表示性能评估的置信区间,较长的误差棒意味着模型性能的变动较大。
- 性能比较: - CONCH模型在图像描述生成任务上的性能优于GIT的两个版本,特别是在METEOR评估标准下。- GIT-large和GIT-base的性能相对接近,但都不如CONCH。
这张图表明CONCH模型在图像描述任务上具有更好的泛化能力和描述精度,能够生成与真实情况更为接近的文本描述。这对于需要精确描述生成病理图像内容的应用场景(如自动报告生成)特别有用。
6-2:高质量字幕示例
这张图展示了模型在病理图像分析中的几个正确预测的例子,每个例子都包括了图像和对应的文本描述。

- Glioblastoma(星形细胞瘤):- 图像显示了高细胞密度的星形细胞瘤,细胞具有丝状活性,血管明显,出血区域周围有少量纤维组织。- 文本描述强调了星形细胞瘤的细胞学特征,包括高度细胞性、丝状活跃的细胞、明显的血管和出血区域。
- Oncocarcytoma(癌泡状瘤):- 图像展示了癌泡状瘤的特征,细胞形成离散的巢状结构,位于肾实质内,由类似多边形细胞和类似肝细胞的细胞组成,具有癌泡状外观。- 文本描述指出了癌泡状瘤的组织学特征,包括在肾实质内形成的离散巢状结构和细胞的多边形性及肝样细胞特征。
- Renal oncocytoma(肾癌):- 图像显示了肾癌的特征,多边形细胞形成离散的巢状结构,类似肝细胞,具有癌泡状外观。- 文本描述了肾癌的组织学特征,包括多边形细胞形成的离散巢状结构和癌泡状外观。
6-3:部分正确字幕示例
这张图展示了模型在病理图像分析中的几个部分正确预测的例子,每个例子都包括了图像和对应的文本描述。
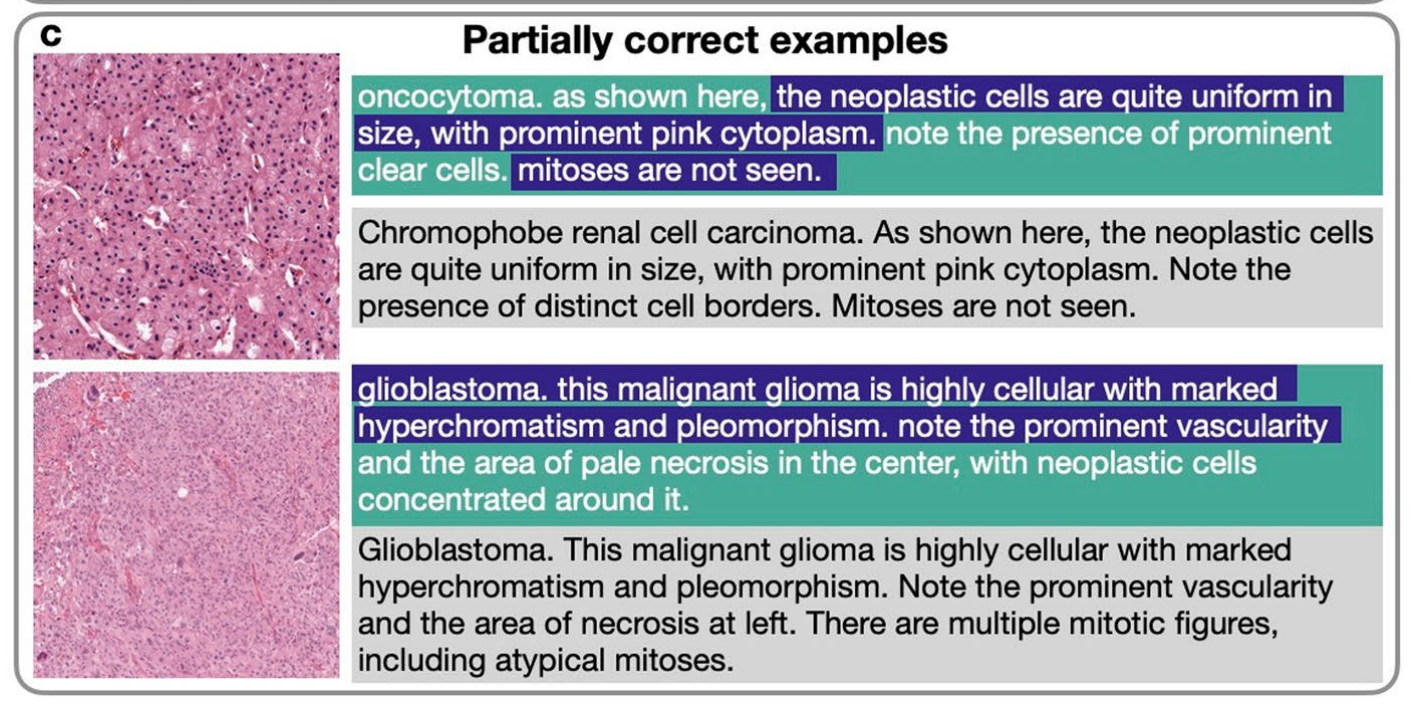
以下是对图中内容的详细分析:
- 癌泡状瘤(oncocytoma):- 图像显示了大小一致的肿瘤细胞,具有明显的粉红色细胞质。文本描述指出了明显的透明细胞存在,但未观察到丝状分裂像。
- 染色体性肾细胞癌(Chromophobe renal cell carcinoma):- 图像展示了大小一致的肿瘤细胞,具有明显的粉红色细胞质。文本描述强调了明显的细胞边界,但同样未观察到丝状分裂像。
- 星形细胞瘤(glioblastoma):- 图像显示了高度细胞性的肿瘤,具有明显的超染色体和多形性。文本描述了明显的血管和中心的淡染坏死区域,肿瘤细胞集中于此。
- 星形细胞瘤(glioblastoma):- 图像显示了高度细胞性的肿瘤,具有明显的超染色体和多形性。文本描述了明显的血管和左侧的坏死区域,存在多个丝状分裂像,包括非典型的丝状分裂像。
此外,一些生成的文本几乎是训练数据集中的原文复述,这可能是由于微调数据集规模有限(训练集:n=558)。
由于当前的预训练规模相对于通用视觉-语言领域的工作来说仍然较小,预计随着更多高质量训练数据的增加,微调后的字幕生成性能有可能会显著提高。
版权归原作者 罗小罗同学 所有, 如有侵权,请联系我们删除。