· Hands-On Machine Learning with Scikit-Learn, Keras, and TensorFlow, 2nd Edition, by Aurélien Géron (O’Reilly). Copyright 2019 Aurélien Géron, 978-1-492-03264-9.
· 《机器学习》周志华
· 环境:Anaconda(Python 3.8) + Pycharm
· 学习时间:2022.05.07~2022.05.08
第八章 降维
许多机器学习问题涉及每个训练实例的成千上万甚至数百万个特征。正如我们将看到的那样,所有这些特征不仅使训练变得极其缓慢,而且还会使找到好的解决方案变得更加困难。这个问题通常称为维度的诅咒。
幸运的是,在实际问题中,通常可以大大减少特征的数量,从而将棘手的问题转变为易于解决的问题。例如,考虑MNIST图像(在第3章中介绍):图像边界上的像素几乎都是白色,因此你可以从训练集中完全删除这些像素而不会丢失太多信息。图7-6确认了这些像素对于分类任务而言完全不重要。另外,两个相邻的像素通常是高度相关的,如果将它们合并为一个像素(例如,通过取两个像素的平均值),不会丢失太多信息。
数据降维确实会丢失一些信息(就好比将图像压缩为JPEG会降低其质量一样),所以,它虽然能够加速训练,但是也会轻微降低系统性能。同时它也让流水线更为复杂,维护难度上升。因此,如果训练太慢,你首先应该尝试的还是继续使用原始数据,然后再考虑数据降维。不过在某些情况下,降低训练数据的维度可能会滤除掉一些不必要的噪声和细节,从而导致性能更好(但通常来说不会,它只会加速训练)。
除了加快训练,降维对于数据可视化(或称DataViz)也非常有用。将维度降到两个(或三个),就可以在图形上绘制出高维训练集,通过视觉来检测模式,常常可以获得一些十分重要的洞察,比如聚类。此外,DataViz对于把你的结论传达给非数据科学家至关重要,尤其是将使用你的结果的决策者。
本章将探讨维度的诅咒,简要介绍高维空间中发生的事情。然后,我们将介绍两种主要的数据降维方法(投影和流形学习),并学习现在最流行的三种数据降维技术:PCA、Kernal PCA以及LLE。
文章目录
8.1 维度的诅咒
我们太习惯三维空间的生活,所以当我们试图去想象一个高维空间时,直觉思维很难成功。即使是一个基本的四维超立方体,我们也很难在脑海中想象出来,更不用说在一个千维空间中弯曲的二百维椭圆体。
事实证明,在高维空间中,许多事物的行为都迥然不同。例如,如果你在一个单位平面(1×1的正方形)内随机选择一个点,那么这个点离边界的距离小于0.001的概率只有约0.4%(也就是说,一个随机的点不大可能刚好位于某个维度的“极端”)。但是,在一个10 000维的单位超立方体(1×1…×1立方体,一万个1)中,这个概率大于99.99999%。高维超立方体中大多数点都非常接近边界。
还有一个更麻烦的区别:如果你在单位平面中随机挑两个点,这两个点之间的平均距离大约为0.52。如果在三维的单位立方体中随机挑两个点,两点之间的平均距离大约为0.66。但是,如果在一个100万维的超立方体中随机挑两个点呢?不管你相信与否,平均距离大约为408.25(约等于
1000000
/
6
\sqrt{1000000/6}
1000000/6)!这是非常违背直觉的:位于同一个单位超立方体中的两个点,怎么可能距离如此之远?这个事实说明高维数据集有很大可能是非常稀疏的:大多数训练实例可能彼此之间相距很远。当然,这也意味着新的实例很可能远离任何一个训练实例,导致跟低维度相比,预测更加不可靠,因为它们基于更大的推测。简而言之,训练集的维度越高,过拟合的风险就越大。
理论上来说,通过增大训练集,使训练实例达到足够的密度,是可以解开维度的诅咒的。然而不幸的是,实践中,要达到给定密度,所需要的训练实例数量随着维度的增加呈指数式上升。仅仅100个特征下(远小于MNIST问题),要让所有训练实例(假设在所有维度上平均分布)之间的平均距离小于0.1,你需要的训练实例数量就比可观察宇宙中的原子数量还要多。
8.2 降维的主要方法
在深入研究特定的降维算法之前,让我们看一下减少维度的两种主要方法:投影和流形学习。
8.2.1 投影
在大多数实际问题中,训练实例并不是均匀地分布在所有维度上。许多特征几乎是恒定不变的,而其他特征则是高度相关的(如之前针对MNIST所述)。结果,所有训练实例都位于(或接近于)高维空间的低维子空间内。这听起来很抽象,所以让我们看一个示例。在下图中,你可以看到由圆圈表示的3D数据集。
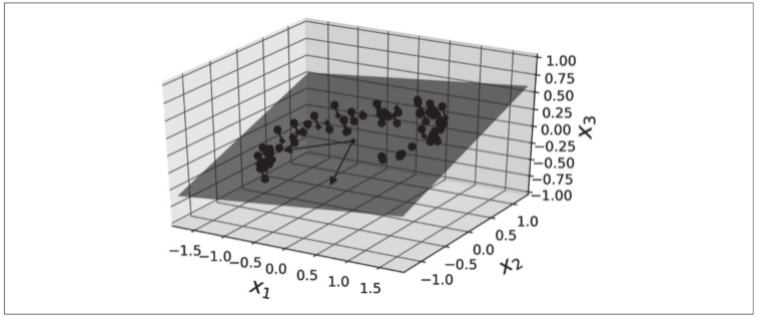
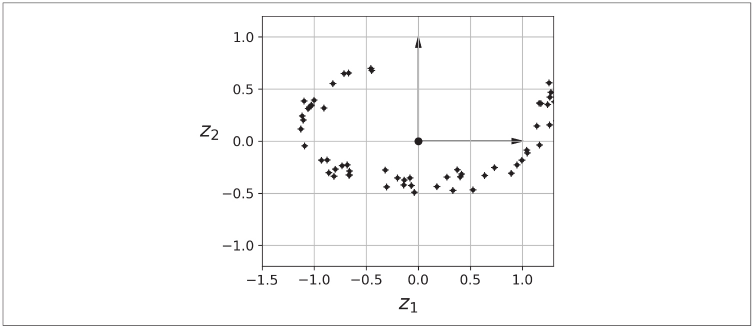
请注意,所有训练实例都位于一个平面附近:这是高维(3D)空间的低维(2D)子空间。如果我们将每个训练实例垂直投影到该子空间上(如实例连接到平面的短线所示),我们将获得如下图所示的新2D数据集——我们刚刚将数据集的维度从3D减少到2D。注意,轴对应于新特征z1和z2(平面上投影的坐标)。
但是,投影并不总是降低尺寸的最佳方法。在许多情况下,子空间可能会发生扭曲和转动,例如在下图中所示的著名的瑞士卷小数据集中。
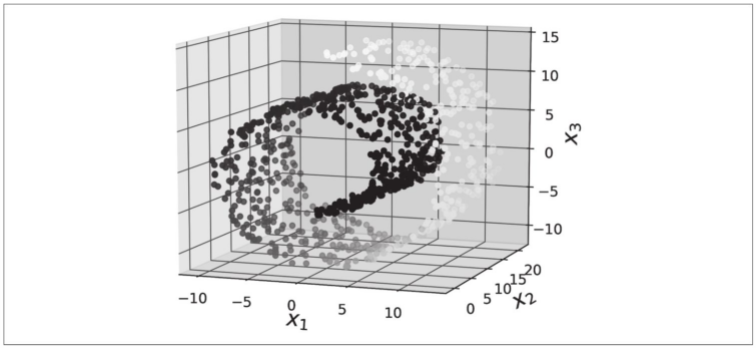
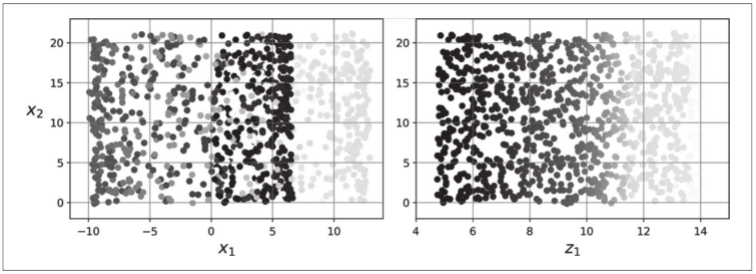
如下图左侧所示,简单地投影到一个平面上(例如,去掉x3维度)会将瑞士卷的不同层挤压在一起。你真正想要的是展开瑞士卷,得到下图右侧的2D数据集。
8.2.2 流形学习
瑞士卷是2D流形的一个示例。简而言之,2D流形是可以在更高维度的空间中弯曲和扭曲的2D形状。更一般而言,d维流形是n维空间(其中d<n)的一部分,局部类似于d维超平面。在瑞士卷的情况下,d=2且n=3时,它局部类似于2D平面,但在第三维中弯曲。
许多降维算法通过对训练实例所在的流形进行建模来工作。这称为流形学习。它依赖于流形假设(也称为流形假说),该假设认为大多数现实世界的高维数据集都接近于低维流形。通常这是根据经验观察到的这种假设。
再次考虑一下MNIST数据集:所有手写数字图像都有一些相似之处。它们由连接的线组成,边界为白色,并且或多或少居中。如果你随机生成图像,那么其中只有一小部分看起来像手写数字。换句话说,如果你试图创建数字图像,可用的自由度大大低于允许你生成任何图像的自由度。这些约束倾向于将数据集压缩为低维流形。
流形假设通常还伴随着另一个隐式假设:如果用流形的低维空间表示,手头的任务(例如分类或回归)将更加简单。例如,在下图的上面一行中,瑞士卷分为两类:在3D空间(左侧)中,决策边界会相当复杂,而在2D展开流形空间中(右侧),决策边界是一条直线。
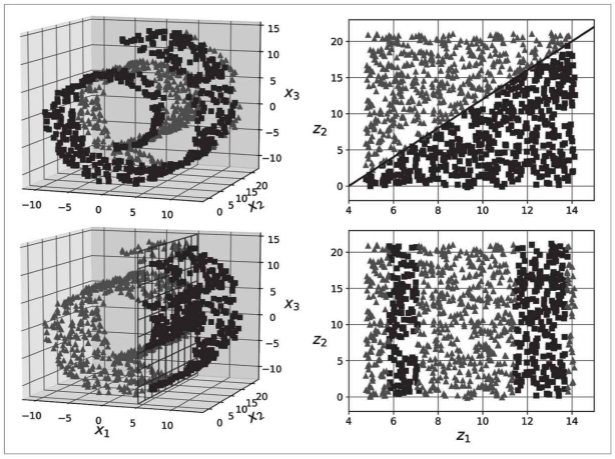
但是,这种隐含假设并不总是成立。例如,在上图的下面一行中,决策边界位于x1=5处。此决策边界在原始3D空间(垂直平面)中看起来非常简单,但在展开流形中看起来更加复杂(四个独立线段的集合)。
简而言之,在训练模型之前降低训练集的维度肯定可以加快训练速度,但这并不总是会导致更好或更简单的解决方案,它取决于数据集。
希望现在你对于维度的诅咒有了一个很好的理解,也知道降维算法是怎么解决它的,特别是当流形假设成立的时候应该怎么处理。本章剩余部分将逐一介绍几个最流行的算法。
8.3 主成分分析 PCA
主成分分析(PCA)是迄今为止最流行的降维算法。首先,它识别最靠近数据的超平面,然后将数据投影到其上。
8.3.1 保留差异性
将训练集投影到低维超平面之前需要选择正确的超平面。例如下图的左图代表一个简单的2D数据集,沿三条不同的轴(即一维超平面)。右图是将数据集映射到每条轴上的结果。正如你所见,在实线上的投影保留了最大的差异性,而点线上的投影只保留了非常小的差异性,虚线上的投影的差异性居中。
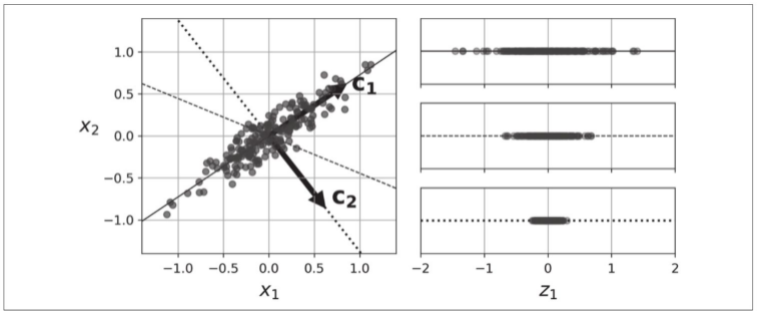
选择保留最大差异性的轴看起来比较合理,因为它可能比其他两种投影丢失的信息更少。要证明这一选择,还有一种方法,即比较原始数据集与其轴上的投影之间的均方距离,使这个均方距离最小的轴是最合理的选择,也就是实线代表的轴。这也正是PCA背后的简单思想。
8.3.2 主要成分
主成分分析可以在训练集中识别出哪条轴对差异性的贡献度最高。在上图中是由实线表示的轴。同时它也找出了第二条轴,与第一条轴垂直,它对剩余差异性的贡献度最高。因为这个示例是二维的,所以除了这条点线再没有其他。如果是在更高维数据集中,PCA还会找到与前两条都正交的第三条轴,以及第四条、第五条,等等——轴的数量与数据集维度数量相同。
第i个轴称为数据的第i个主要成分(PC)。在上图中,第一个PC是向量c1所在的轴,第二个PC是向量c2所在的轴。
对于每个主要成分,PCA都找到一个指向PC方向的零中心单位向量。由于两个相对的单位向量位于同一轴上,因此PCA返回的单位向量的方向不稳定:如果稍微扰动训练集并再次运行PCA,则单位向量可能会指向原始向量的相反方向。但是,它们通常仍位于相同的轴上。在某些情况下,一对单位向量甚至可以旋转或交换(如果沿这两个轴的方差接近),但是它们定义的平面通常保持不变。
那么如何找到训练集的主要成分呢?幸运的是,有一种称为奇异值分解(SVD)的标准矩阵分解技术,该技术可以将训练集矩阵
X
X
X分解为三个矩阵
U
⋅
Σ
V
T
U·ΣV^T
U⋅ΣVT的矩阵乘法,其中
V
V
V包含定义所有主要成分的单位向量。如公式所示。

以下Python代码使用NumPy的
svd()
函数来获取训练集的所有主要成分,然后提取定义前两个PC的两个单位向量:
import numpy as np
# 构建数据集
np.random.seed(4)
m =60
w1, w2 =0.1,0.3
noise =0.1
angles = np.random.rand(m)*3* np.pi /2-0.5
X = np.empty((m,3))
X[:,0]= np.cos(angles)+ np.sin(angles)/2+ noise * np.random.randn(m)/2
X[:,1]= np.sin(angles)*0.7+ noise * np.random.randn(m)/2
X[:,2]= X[:,0]* w1 + X[:,1]* w2 + noise * np.random.randn(m)# 调用奇异值分解SVD
X_centered = X - X.mean(axis=0)
U, s, Vt = np.linalg.svd(X_centered)
c1 = Vt.T[:,0]
c2 = Vt.T[:,1]
PCA假定数据集以原点为中心。正如我们将看到的,Scikit-Learn的PCA类负责为你居中数据。如果你自己实现PCA(如上例所示),或者使用其他库,请不要忘记首先将数据居中(标准化)。
8.3.3 向下投影到d维度
一旦确定了所有主要成分,你就可以将数据集投影到前d个主要成分定义的超平面上,从而将数据集的维度降低到d维。选择这个超平面可确保投影将保留尽可能多的差异性。例如,在上图(8.2.1)中,将3D数据集投影到由前两个主成分定义的2D平面上,从而保留了数据集大部分的差异性。最终,2D投影看起来非常类似于原始3D数据集。
要将训练集投影到超平面上并得到维度为
d
d
d的简化数据集
X
d
−
p
r
o
j
X_{d-proj}
Xd−proj,计算训练集矩阵
X
X
X与矩阵
W
d
W_d
Wd的矩阵相乘,矩阵
W
d
W_d
Wd定义为包含
V
V
V的前
d
d
d列的矩阵,如公式所示。
X
d
−
p
r
o
j
=
X
W
d
X_{d-proj} = XW_d
Xd−proj=XWd
以下Python代码将训练集投影到由前两个主要成分定义的平面上:
W2 = Vt.T[:,:2]
X2D = X_centered.dot(W2)
你现在知道了如何将任何数据集的维度减少到任意数量的维度,同时保留尽可能多的差异性。
8.3.4 使用Scikit-Learn
就像我们在本章前面所做的那样,Scikit-Learn的PCA类使用SVD分解来实现PCA。以下代码应用PCA将数据集的维度降到二维(请注意,它会自动处理数据居中的问题):
from sklearn.decomposition import PCA
pca = PCA(n_components=2)# 将数据集的维度降到2维
X2D = pca.fit_transform(X)
将PCA转换器拟合到数据集后,其
components_
属性是
W
d
W_d
Wd的转置(例如,定义第一个主成分的单位向量等于
pca.components.T[:, 0]
)。
完整代码如下:
import numpy as np # 构建数据集 np.random.seed(4) m =60 w1, w2 =0.1,0.3 noise =0.1 angles = np.random.rand(m)*3* np.pi /2-0.5 X = np.empty((m,3)) X[:,0]= np.cos(angles)+ np.sin(angles)/2+ noise * np.random.randn(m)/2 X[:,1]= np.sin(angles)*0.7+ noise * np.random.randn(m)/2 X[:,2]= X[:,0]* w1 + X[:,1]* w2 + noise * np.random.randn(m)# 调用奇异值分解SVD X_centered = X - X.mean(axis=0) U, s, Vt = np.linalg.svd(X_centered) c1 = Vt.T[:,0] c2 = Vt.T[:,1]print('X: ', X) W2 = Vt.T[:,:2]# X2D = X_centered.dot(W2)print('X_centered:', X_centered)from sklearn.decomposition import PCA pca = PCA(n_components=2) X2D = pca.fit_transform(X)print('X2D: ', X2D)
8.3.5 可解释方差比
另一个有用的信息是每个主成分的可解释方差比,可以通过
explained_variance_ratio_
变量来获得。该比率表示沿每个成分的数据集方差的比率。例如,让我们看一下图8-2中表示的3D数据集的前两个成分的可解释方差比:
print('输出:\n', pca.explained_variance_ratio_)# 输出:# [0.84248607 0.14631839]
此输出告诉你,数据集方差的84.2%位于第一个PC上,而14.6%位于第二个PC上。对于第三个PC,这还不到1.2%,因此可以合理地假设第三个PC携带的信息很少。
即用来解释成分的重要性占比。
8.3.6 选择正确的维度
与其任意选择要减小到的维度,不如选择相加足够大的方差部分(例如95%)的维度。当然,如果你是为了数据可视化而降低维度,这种情况下,需要将维度降低到2或3。
以下代码在不降低维度的情况下执行PCA,然后计算保留95%训练集方差所需的最小维度:
from sklearn.datasets import fetch_openml
from sklearn.model_selection import train_test_split
# 导入MINST数据集
mnist = fetch_openml('mnist_784', version=1)
mnist.target = mnist.target.astype(np.uint8)# 划分训练集和测试集
X = mnist["data"]
y = mnist["target"]
X_train, X_test, y_train, y_test = train_test_split(X, y)# 应用(在不降低维度的情况下执行PCA,然后计算保留95%训练集方差所需的最小维度)
pca.fit(X_train)
cumsum = np.cumsum(pca.explained_variance_ratio_)
d = np.argmax(cumsum >=0.95)+1print(d)
然后,你可以设置n_components=d并再次运行PCA。但是还有一个更好的选择:**将
n_components
设置为0.0到1.0之间的浮点数来表示要保留的方差率,而不是指定要保留的主成分数**:
pca = PCA(n_components=0.95)
X_reduced = pca.fit_transform(X_train)
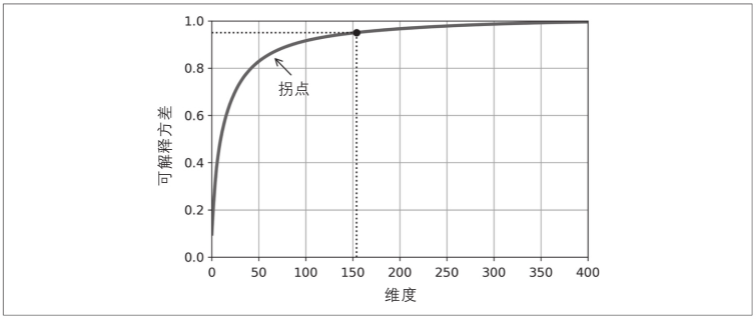
另一个选择是将可解释方差绘制成维度的函数(简单地用cumsum绘制,见下图)。曲线上通常会出现一个拐点,其中可解释方差会停止快速增大。在这种情况下,你可以看到将维度降低到大约100而不会损失太多的可解释方差。
8.3.7 PCA压缩
降维后,训练集占用的空间要少得多。例如,将PCA应用于MNIST数据集,同时保留其95%的方差。你会发现每个实例将具有150多个特征,而不是原始的784个特征。因此,尽管保留了大多数方差,但数据集现在不到其原始大小的20%!这是一个合理的压缩率,你可以看到这种维度减小极大地加速了分类算法(例如SVM分类器)。
通过应用PCA投影的逆变换,还可以将缩减后的数据集解压缩回784维。由于投影会丢失一些信息(在5%的方差被丢弃),因此这不会给你原始的数据,但可能会接近原始数据。原始数据与重构数据(压缩后再解压缩)之间的均方距离称为重构误差。
以下代码将MNIST数据集压缩为154个维度,然后使用
inverse_transform()
方法将其解压缩回784个维度:
pca = PCA(n_components=154)
X_reduced = pca.fit_transform(X_train)# 逆变换: 解压缩
X_recovered = pca.inverse_transform(X_reduced)
下图显示了原始训练集的一些数字(左侧),以及压缩和解压缩后的相应数字。你会看到图像质量略有下降,但是数字仍然大部分保持完好。

逆变换的公式如下所示。
X
r
e
c
o
v
e
r
e
d
=
X
d
−
p
r
o
j
W
d
X_{recovered} = X_{d-proj}W_d
Xrecovered=Xd−projWd
8.3.8 随机PCA
如果将超参数
svd_solver
设置为"randomized",则Scikit-Learn将使用一种称为Randomized PCA的随机算法,该算法可以快速找到前d个主成分的近似值。它的计算复杂度为
O
(
m
×
d
2
)
+
O
(
d
3
)
O(m×d^2)+O(d^3)
O(m×d2)+O(d3),而不是完全SVD方法的
O
(
m
×
n
2
)
+
O
(
n
3
)
O(m×n^2)+O(n^3)
O(m×n2)+O(n3),因此,当
d
d
d远远小于
n
n
n时,它比完全的SVD快得多:
# 随机PCA
rnd_pca = PCA(n_components=154, svd_solver="randomized")
X_reduced = rnd_pca.fit_transform(X_train)
默认情况下,
svd_solver
实际上设置为"auto":如果m或n大于500并且d小于m或n的80%,则Scikit-Learn自动使用随机PCA算法,否则它将使用完全的SVD方法。如果要强制Scikit-Learn使用完全的SVD,可以将
svd_solver
超参数设置为"full"。
8.3.9 增量PCA(IPCA)
前面的PCA实现的一个问题是,它们要求整个训练集都放入内存才能运行算法。幸运的是已经开发了增量PCA(IPCA)算法,它们可以使你把训练集划分为多个小批量,并一次将一个小批量送入IPCA算法。这对于大型训练集和在线(即在新实例到来时动态运行)应用PCA很有用。
以下代码将MNIST数据集拆分为100个小批量(使用NumPy的array_split()函数),并将其馈送到Scikit-Learn的IncrementalPCA类,来把MNIST数据集的维度降低到154(就像之前做的那样)。请注意,你必须在每个小批量中调用
partial_fit()
方法,而不是在整个训练集中调用
fit()
方法:
# IPCAfrom sklearn.decomposition import IncrementalPCA
n_batches =100
inc_pca = IncrementalPCA(n_components=154)for X_batch in np.array_split(X_train, n_batches):
inc_pca.partial_fit(X_batch)
X_reduced = inc_pca.transform(X_train)
另外,你可以使用NumPy的memmap类,该类使你可以将存储在磁盘上的二进制文件中的大型数组当作完全是在内存中一样来操作,该类仅在需要时才将数据加载到内存中。由于IncrementalPCA类在任何给定时间仅使用数组的一小部分,因此内存使用情况处于受控状态。如以下代码所示,这使得调用通常的
fit()
方法成为可能:
filename ="my_mnist.data"
X_mm = np.memmap(filename, dtype="float32", mode="readonly", shape=(m, n))
batch_size = m // n_batches
inc_pca = IncrementalPCA(n_components=154, batch_size=batch_size)
inc_pca.fit(X_mm)
8.4 内核PCA(kPCA)
在第5章中,我们讨论了内核,这是一种数学技术,它可以将实例隐式映射到一个高维空间(称为特征空间),从而可以使用支持向量机来进行非线性分类和回归。回想一下,高维特征空间中的线性决策边界对应于原始空间中的复杂非线性决策边界。
事实证明,可以将相同的技术应用于PCA,从而可以执行复杂的非线性投影来降低维度。这叫作内核PCA(kPCA)。它通常擅长在投影后保留实例的聚类,有时甚至可以展开位于扭曲流形附近的数据集。
下面的代码使用Scikit-Learn的KernelPCA类以及用RBF内核来执行kPCA(有关RBF内核和其他内核的更多详细信息,请参见第5章):
from sklearn.decomposition import KernelPCA
rbf_pca = KernelPCA(n_components=2, kernel="rbf", gamma=0.04)
X_reduced = rbf_pca.fit_transform(X)
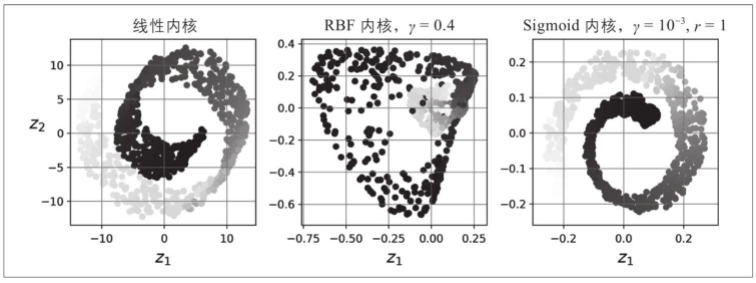
下图显示了瑞士卷,它使用线性内核(相当于简单地使用PCA类)、RBF内核和sigmoid内核减小为二维。
选择内核并调整超参数
由于kPCA是一种无监督学习算法,因此没有明显的性能指标可以帮助你选择最好的内核和超参数值。也就是说,降维通常是有监督学习任务(例如分类)的准备步骤,因此你可以使用网格搜索来选择在该任务上能获得最佳性能的内核和超参数。以下代码创建了一个两步流水线,首先使用kPCA将维度减少到二维,然后使用逻辑回归来分类。它使用GridSearchCV来查找kPCA的最佳内核和gamma值,以便在流水线的最后得到最好的分类准确率:
from sklearn.model_selection import GridSearchCV
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import Pipeline
clf = Pipeline([("kpca", KernelPCA(n_components=2)),("log_reg", LogisticRegression())])
param_grid =[{"kpca__gamma": np.linspace(0.03,0.05,10),"kpca__kernel":["rbf","sigmoid"]}]
grid_search = GridSearchCV(clf, param_grid, cv=3)
grid_search.fit(X, y)
然后,可以通过
best_params_
变量来得到最佳内核和超参数:
print(grid_search.best_params_)# {'kpca__gamma': 0.043333333333333335, 'kpca__kernel': 'rbf'}
另一种完全无监督的方法是选择产生最低重构误差的内核和超参数。请注意,重建并不像使用线性PCA那样容易。以下是原因。
下图显示了原始的瑞士卷3D数据集(左上)和使用RBF内核应用kPCA之后得到的2D数据集(右上)。多亏了内核技术,此变换在数学上等效于使用特征图φ将训练集映射到无限维特征空间(右下),然后使用线性PCA将变换后的训练集投影到2D。
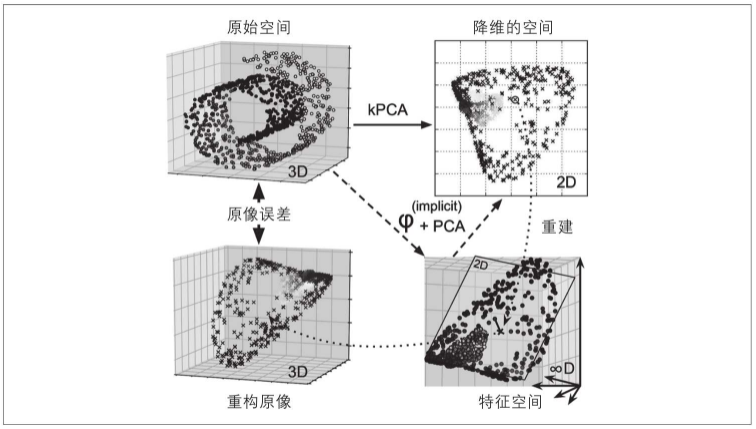
请注意,如果我们可以对一个给定的实例在缩小的空间中反转线性PCA,则重构点将位于特征空间中,而不是原始空间中(例如,如图中的X所示)。由于特征空间是无限维的,因此我们无法计算重构点,无法计算真实的重构误差。幸运的是,有可能在原始空间中找到一个点,该点将映射到重建点附近,这一点称为重建原像。一旦你有了原像,就可以测量其与原始实例的平方距离。然后可以选择内核和超参数,最大限度地减少此重构原像误差。
你可能想知道如何执行这个重构。一种解决方案是训练有监督的回归模型,其中将投影实例作为训练集,将原始实例作为目标值。如果设置fit_inverse_transform=True,Scikit-Learn会自动执行此操作,如以下代码所示:
rbf_pca = KernelPCA(n_components=2, kernel="rbf", gamma=0.0433, fit_inverse_transform=True)
X_reduced = rbf_pca.fit_transform(X)
X_preimage = rbf_pca.inverse_transform(X_reduced)
默认情况下,
fit_inverse_transform=False
,并且KernelPCA没有
inverse_transform()
方法。仅当你设置
fit_inverse_transform=True
时,才会创建此方法。
计算重建原像误差:
from sklearn.metrics import mean_squared_error
print(mean_squared_error(X, X_preimage))# 输出:32.786308795766132
现在,你可以使用网格搜索与交叉验证来找到可最大限度减少此错误的内核和超参数。
8.5 局部线性嵌入LLE
局部线性嵌入(LLE)是另一种强大的非线性降维(NLDR)技术。它是一种流形学习技术,不像以前的算法那样依赖于投影。简而言之,LLE的工作原理是首先测量每个训练实例如何与其最近的邻居(c.n.)线性相关,然后寻找可以最好地保留这些局部关系的训练集的低维表示形式(稍后会详细介绍)。这种方法特别适合于展开扭曲的流形,尤其是在没有太多噪声的情况下。
以下代码使用Scikit-Learn的LocallyLinearEmbedding类来展开瑞士卷:
from sklearn.manifold import LocallyLinearEmbedding
lle = LocallyLinearEmbedding(n_components=2, n_neighbors=10)
X_reduced = lle.fit_transform(X)
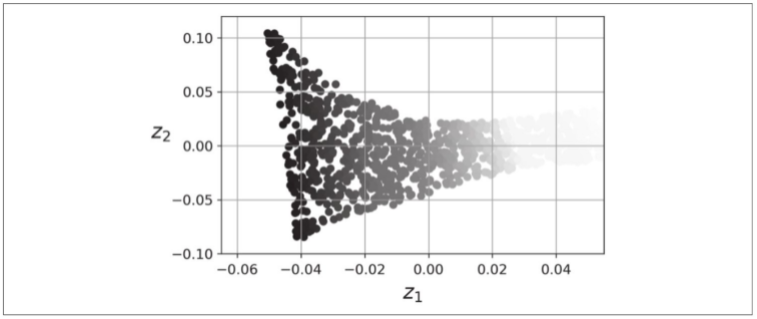
生成的2D数据集下图所示。如你所见,瑞士卷已完全展开,并且实例之间的距离在局部得到了很好的保留。但是,距离并没有在更大范围内保留:展开的瑞士卷的左侧部分被拉伸,而右侧部分被挤压。尽管如此,LLE在流形建模方面做得很好。
LLE的工作方式如下:对于每个训练实例
x
(
i
)
x^{(i)}
x(i),算法都会识别出其k个最近的邻居(在前面的代码k=10中),然后尝试将
x
(
i
)
x^{(i)}
x(i)重构为这些邻居的线性函数。更具体地说,它找到权重
w
i
,
j
w_{i,j}
wi,j,使得
x
(
i
)
x^{(i)}
x(i)与
∑
j
=
1
m
w
i
,
j
x
j
\sum^m_{j=1}w_{i,j}x^{j}
∑j=1mwi,jxj之间的平方距离尽可能小,并假设如果
x
(
j
)
x^{(j)}
x(j)不是
x
(
i
)
x^{(i)}
x(i)的k个最接近的邻居之一则
w
i
,
j
=
0
w_{i,j}=0
wi,j=0。因此,LLE的第一步是下面公式中描述的约束优化问题,其中W是包含所有权重
w
i
,
j
w_{i,j}
wi,j的权重矩阵。第二个约束条件只是简单地将每个训练实例
x
(
i
)
x^{(i)}
x(i)的权重归一化。
W
′
=
a
r
g
m
i
n
∑
i
=
1
m
(
x
(
i
)
−
∑
j
=
1
m
w
i
,
j
x
(
j
)
)
2
W' = argmin\sum^m_{i=1}(x^{(i)}-\sum^m_{j=1}w_{i,j}x^{(j)})^2
W′=argmini=1∑m(x(i)−j=1∑mwi,jx(j))2
在此步骤之后,权重矩阵
W
′
W'
W′(包含权重
w
i
,
j
′
w'_{i,j}
wi,j′)对训练实例之间的局部线性关系进行编码。第二步是将训练实例映射到d维空间(其中d<n),同时尽可能保留这些局部关系。如果
z
(
i
)
z^{(i)}
z(i)是此d维空间中
x
(
i
)
x^{(i)}
x(i)的图像,则我们希望
z
(
i
)
z^{(i)}
z(i)与
∑
j
=
1
m
w
i
,
j
′
z
j
\sum^m_{j=1}w'_{i,j}z^{j}
∑j=1mwi,j′zj之间的平方距离尽可能小。
这种想法导致了下面公式中描述的无约束优化问题。它看起来与第一步非常相似,但是我们没有保持实例固定并找到最佳权重,而是相反:保持权重固定并找到实例图像在低维空间中的最佳位置。注意
Z
Z
Z是包含所有
z
(
i
)
z^{(i)}
z(i)的矩阵。
Z
′
=
a
r
g
m
i
n
∑
i
=
1
m
(
z
(
i
)
−
∑
j
=
1
m
w
i
,
j
z
(
j
)
)
2
Z' = argmin\sum^m_{i=1}(z^{(i)}-\sum^m_{j=1}w_{i,j}z^{(j)})^2
Z′=argmini=1∑m(z(i)−j=1∑mwi,jz(j))2
Scikit-Learn的LLE实现具有以下计算复杂度:
O
(
m
⋅
l
o
g
m
⋅
n
⋅
l
o
g
k
)
O(m·log^m·n·log^k)
O(m⋅logm⋅n⋅logk)用于找到k个最近的邻居,
O
(
m
⋅
n
⋅
k
3
)
O(m·n·k^3)
O(m⋅n⋅k3)用于优化权重,
O
(
d
⋅
m
2
)
O(d·m^2)
O(d⋅m2)用于构造低维表示。不幸的是,最后一项中的
m
2
m^2
m2使该算法很难扩展到非常大的数据集。
8.6 其他降维技术
还有许多其他降维技术,Scikit-Learn中提供了其中几种。以下是一些很受欢迎的降维技术:
随机投影
- 顾名思义,使用随机线性投影将数据投影到较低维度的空间。这听起来可能很疯狂,但事实证明,这样的随机投影实际上很有可能很好地保持距离,就如William B.Johnson和Joram Lindenstrauss在著名引理中的数学证明。降维的质量取决于实例数目和目标维度,令人惊讶的不取决于初始维度。请查看
sklearn.random_projection软件包的文档以获取更多详细信息。
多维缩放(MDS)
- 当尝试保留实例之间的距离时降低维度。
等度量映射Isomap
- 等度量映射(Isornetric Mapping,简称 Isomap)【Tenenbaum et al,2000】 的基本出发点,是认为低维流形嵌入到高维空问之后,直接在高维空间中计算直线距离具有误导性,对为高维空间中的直线距离在低维嵌入流形上是不可达的。
- 通过将每个实例与其最近的邻居连接来创建一个图,然后在尝试保留实例之间的测地距离的同时降低维度。
t分布随机近邻嵌入(t-SNE)
- 降低了维度,同时使相似实例保持接近,异类实例分开。它主要用于可视化,特别是在高维空间中可视化实例的聚类(例如,以2D可视化MNIST图像)。
线性判别分析(LDA)
- LDA是一种分类算法,但是在训练过程中,它会学习各类之间最有判别力的轴,然后可以使用这些轴来定义要在其上投影数据的超平面。这种方法的好处是投影将使类保持尽可能远的距离,因此LDA是在运行其他分类算法(例如SVM分类器)之前降低维度的好技术。
8.7 练习题
问题
- 减少数据集维度的主要动机是什么?主要缺点是什么?
- 维度的诅咒是什么?
- 一旦降低了数据集的维度,是否可以逆操作?如果可以,怎么做?如果不能,为什么?
- 可以使用PCA来减少高度非线性的数据集的维度吗?
- 假设你在1000维的数据集上执行PCA,将可解释方差比设置为95%。结果数据集将具有多少个维度?
- 在什么情况下,你将使用常规PCA、增量PCA、随机PCA或内核PCA?
- 如何评估数据集中的降维算法的性能?
- 链接两个不同的降维算法是否有意义?
- 加载MNIST数据集(在第3章中介绍),并将其分为训练集和测试集(使用前60 000个实例进行训练,其余10 000个进行测试)。在数据集上训练随机森林分类器,花费多长时间,然后在测试集上评估模型。接下来,使用PCA来减少数据集的维度,可解释方差率为95%。在精简后的数据集上训练新的随机森林分类器,查看花费了多长时间。训练速度提高了吗?接下来,评估测试集上的分类器。与之前的分类器相比如何?
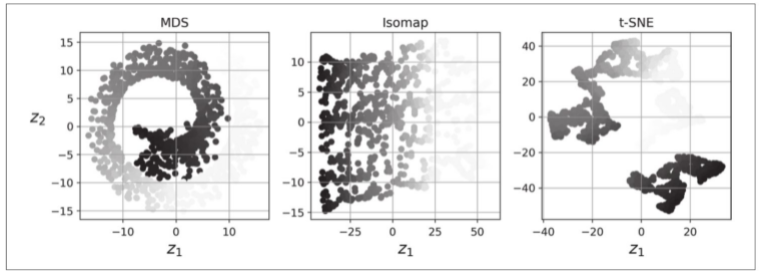
- 使用t-SNE将MNIST数据集降至两个维度,然后用Matplotlib绘制结果。你可以通过散点图用10个不同的颜色来代表每个图像的目标类,或者也可以用对应实例的类(从0到9的数字)替换散点图中的每个点,甚至你还可以绘制数字图像本身的缩小版(如果你绘制所有数字,视觉效果会太凌乱,所以你要么绘制一个随机样本,要么选择单个实例,但是这个实例的周围最好没有其他绘制的实例)。现在你应该得到了一个很好的可视化结果及各自分开的数字集群。尝试使用其他降维算法,如PCA、LLE或MDS等,比较可视化结果。
答案
- 降维的主要动机是:- 为了加速后续的训练算法(在某些情况下,也可能为了消除噪声和冗余特征,使训练算法性能更好)。- 为了将数据可视化,并从中获得洞见,了解最重要的特征。- 为了节省空间(压缩)。主要的弊端是:- 丢失部分信息,可能使后续训练算法的性能降低。- 可能是计算密集型的。- 为机器学习流水线增添了些许复杂度。- 转换后的特征往往难以解释。
- 维度的诅咒是指许多在低维空间中不存在的问题,在高维空间中发生。在机器学习领域,一个常见的现象是随机抽样的高维向量通常非常稀疏,提升了过拟合的风险,同时也使得在没有充足训练数据的情况下,要识别数据中的模式非常困难。
- 一旦使用我们讨论的任意算法减少了数据集的维度,就几乎不可能再将操作完美地逆转,因为在降维过程中必然丢失了一部分信息。此外,虽然有一些算法(例如PCA)拥有简单的逆转换过程,可以重建出与原始数据集相似的数据集,但是也有一些算法不能实现逆转(例如T-SNE)。
- 对大多数数据集来说,PCA可以用来进行显著降维,即便是高度非线性的数据集,因为它至少可以消除无用的维度。但是如果不存在无用的维度(例如瑞士卷),那么使用PCA降维将会损失太多信息。你希望的是将瑞士卷展开,而不是将其压扁。
- 这是个不好回答的问题,它取决于数据集。我们来看看两个极端的示例。首先,假设数据集是由几乎完全对齐的点组成的,在这种情况下,PCA可以将数据集降至一维,同时保留95%的方差。现在,试想数据集由完全随机的点组成,分散在1000个维度上,在这种情况下,需要在950个维度上保留95%的方差。所以,这个问题的答案是:取决于数据集,它可能是1到950之间的任何数字。将解释方差绘制成关于维度数量的函数,可以对数据集的内在维度获得一个粗略的概念。
- 常规PCA是默认选择,但是它仅适用于内存足够处理训练集的时候。增量PCA对于内存无法支持的大型数据集非常有用,但是它比常规PCA要慢一些,所以如果内存能够支持,还是应该使用常规PCA。当你需要随时应用PCA来处理每次新增的实例时,增量PCA对于在线任务同样有用。当你想大大降低维度数量,并且内存能够支持数据集时,使用随机PCA非常有效,它比常规PCA快得多。最后,对于非线性数据集,使用核化PCA非常有效。
- 直观来说,如果降维算法能够消除许多维度并且不会丢失太多信息,那么这就算一个好的降维算法。进行衡量的方法之一是应用逆转换然后测量重建误差。然而并不是所有的降维算法都提供了逆转换。还有另一种选择,如果你将降维当作一个预处理过程,用在其他机器学习算法(比如随机森林分类器)之前,那么可以通过简单测量第二个算法的性能来进行评估。如果降维过程没有损失太多信息,那么第二个算法的性能应该跟使用原始数据集一样好。
- 链接两个不同的降维算法绝对是有意义的。常见的示例是使用PCA快速去除大量无用的维度,然后应用另一种更慢的降维算法,如LLE。这种两步走的策略产生的结果可能与仅使用LLE相同,但是时间要短得多。
有关练习9和10的解答,请参见https://github.com/ageron/handson-ml2上提供的Jupyternotebook。
8.8 阅读材料
主成分分析是一种无监督的绒性降维方法,监督线性降维方法最著名的是线性判别分析(LDA)Fisher,1936】,其核化版本KLDA 【Baudatand Anouar,2000】。通过最大化两个变量集合之间的相关性,则可得到"典型相关分析"(Canonical Correlation Analysis,简称 CCA)Hotelling,1936】 及其核化版本KCCA 【Harden et al.,2004,该方法在多视图学习(multi-view learning)中有广泛应用.在模式识别领域人们发现,直接对矩阵对象(例如一幅图像)进行降维操作会比将其拉伸为向量(例如把图像逐行拼接成一个向量)再进行降维操作有更好的性能,于是产生了2DPCA Yang et al.,204】、2DLDA 【Ye et al,2005】、(2D)PCA 【Zhang and Zhou,2005】 等方法,以及基于张量(ten3or)的方法【Kolda and Bader,2009】。
除了 Isomap和LLE,常见的流形学习方法还有拉普拉斯特征映射 (Laplcian Eigenmaps,简称LE)【【Belkin and Niyogi,2003】、局部切空间对齐(LocalTangent Space Alignment,简称 LTSA)【Zhang and Zha,2004等】。局部保持投影(Locality Preserving Projections,简称 LPP)【He and Niyogi,2004】 是基于LE的线性降维方法.对监督学习而言,根据类别信息扭曲后的低维空间常比本真低维空间更有利【Geng et al.,2005】。值得注意的是,流形学习欲有效进行邻域保持则需样本密采样、而这恰是高维情形下面临的重大障碍,因此流形学习方法在实践中的降维性能往往没有预期的好:但邻域保持的想法对机器学习的其他分支产生了重要影响,例如半监督学习中有著名的流形假设、流形正则化【Belkin et al,2006】。【Yan et al,2007】从图嵌入的角度给出了降维方法的一个统一框架。
将必连关系、勿连关系作为学习任务优化目标的约束,在半监督聚类的研究中使用得更早【Wagstaf et al,2001】。在度量学习中,由于这些约束是对所有样本同时发生作用 【Xing et al,2003】,因此相应的方法被称为全局度量学习方法。人们也尝试利用局部约束(例如邻域内的三元关系),从而产生了局部距离度量学习方法【Weinberger and Saul,2009】,甚至有一些研究试图为每个样本产生最合适的距离度量Frome et al,2007;Zhan et a,2009】。在具体的学习与优化求解方面,不同的度量学习方法往往采用了不同的技术,例如 【Yang etal.,2006】将度量学习转化为判别式概率模型框架下基于样本对的二分类问题求解,【Davis et al,2007】将度量学习转化为信息论框架下的 Bregman 优化问题,能方便地进行在线学习。
版权归原作者 新四石路打卤面 所有, 如有侵权,请联系我们删除。