根据训练样本中是否包含标签信息,机器学习可以分为监督学习和无监督学习。聚类算法是典型的无监督学习,目的是想将那些相似的样本尽可能聚在一起,不相似的样本尽可能分开。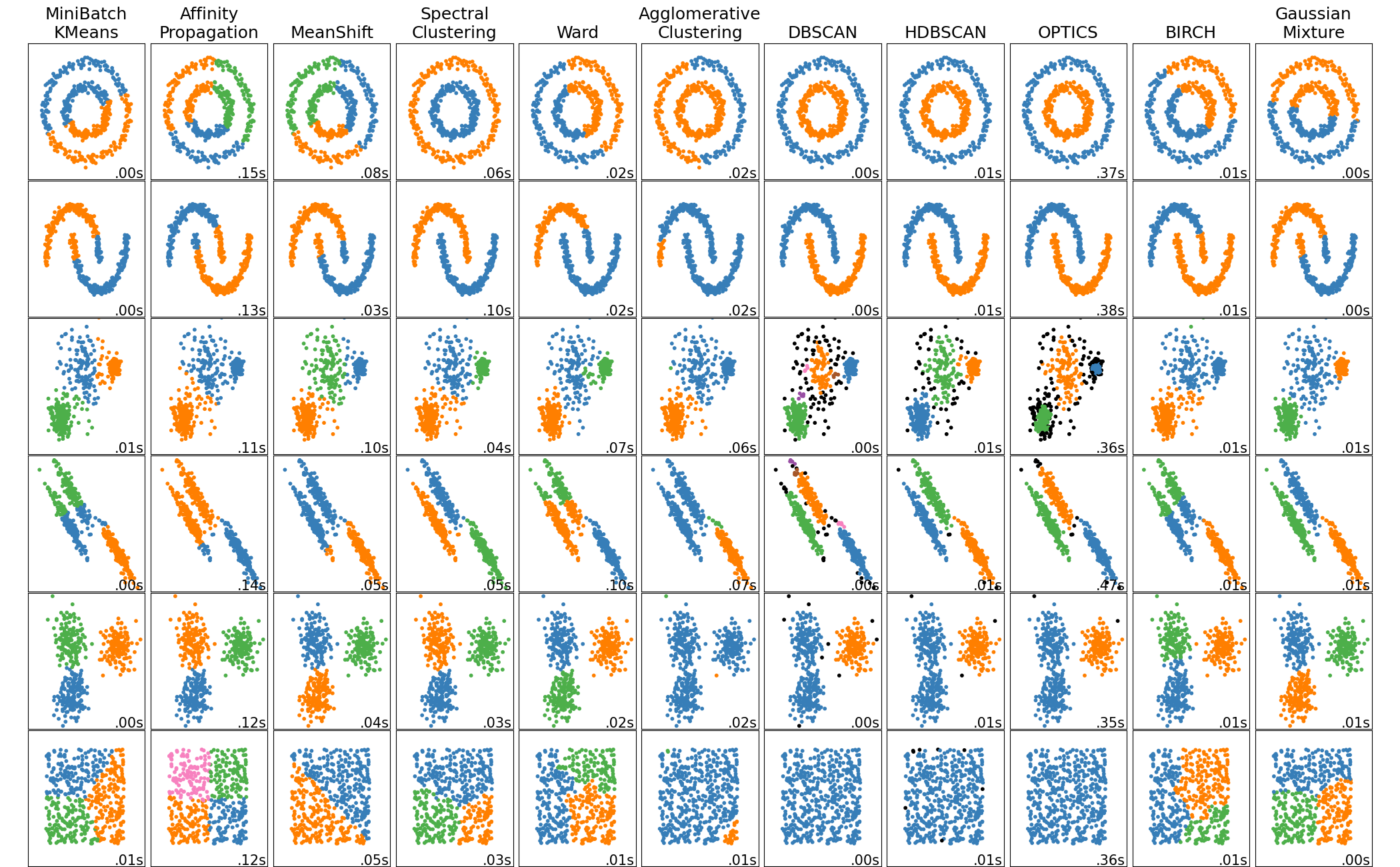
相似度或距离
聚类的核心概念是相似度(similarity)或距离(distance),常用的距离有以下几种:
闵可夫斯基距离(Minkowski distance)
d
(
x
,
y
)
=
(
∑
i
=
1
m
∣
x
i
−
y
i
∣
p
)
1
/
p
d(\mathbf x,\mathbf y)=\left(\sum_{i=1}^m|x_i-y_i|^p\right)^{1/p}
d(x,y)=(i=1∑m∣xi−yi∣p)1/p
其中
p
∈
R
p\in\R
p∈R 且
p
⩾
1
p\geqslant 1
p⩾1 。下面是三个常见的例子
当
p
=
1
p=1
p=1 时,表示曼哈顿距离(Manhattan)。即
d
(
x
,
y
)
=
∑
i
=
1
m
∣
x
i
−
y
i
∣
d(\mathbf x,\mathbf y)=\sum_{i=1}^m|x_i-y_i|
d(x,y)=i=1∑m∣xi−yi∣
当
p
=
2
p=2
p=2 时,表示欧几里德距离(Euclidean)。即
d
(
x
,
y
)
=
∑
i
=
1
m
(
x
i
−
y
i
)
2
d(\mathbf x,\mathbf y)=\sqrt{\sum_{i=1}^m(x_i-y_i)^2}
d(x,y)=i=1∑m(xi−yi)2
当
p
→
∞
p\to\infty
p→∞ 时,表示切比雪夫距离(Chebyshev)。即
d
(
x
,
y
)
=
max
(
∣
x
i
−
y
i
∣
)
d(\mathbf x,\mathbf y)=\max(|x_i-y_i|)
d(x,y)=max(∣xi−yi∣)
马哈拉诺比斯距离(Mahalanobis distance)也是另一种常用的相似度,考虑各个分量(特征)之间的相关性并与各个分量的尺度无关。
d
(
x
,
y
)
=
(
x
−
y
)
Σ
−
1
(
x
−
y
)
T
d(\mathbf x,\mathbf y)=(\mathbf x-\mathbf y)\Sigma^{-1}(\mathbf x-\mathbf y)^T
d(x,y)=(x−y)Σ−1(x−y)T
其中
Σ
−
1
\Sigma^{-1}
Σ−1 是协方差矩阵的逆。
余弦距离(Cosine distance)也可以用来衡量样本之间的相似度。夹角余弦越接近于1,表示样本越相似;越接近于0,表示样本越不相似。
cos
(
x
,
y
)
=
x
⋅
y
∥
x
∥
⋅
∥
y
∥
\cos(\mathbf x,\mathbf y)=\frac{\mathbf x\cdot\mathbf y}{\|\mathbf x\|\cdot\|\mathbf y\|}
cos(x,y)=∥x∥⋅∥y∥x⋅y
K-means
基础算法
K-means是一种广泛使用的聚类算法。将样本集合
X
=
{
x
1
,
x
2
,
⋯
,
x
N
}
X=\{x_1,x_2,\cdots,x_N\}
X={x1,x2,⋯,xN} 划分到
K
K
K个子集中
{
C
1
,
C
2
,
⋯
,
C
K
}
\{C_1,C_2,\cdots,C_K\}
{C1,C2,⋯,CK},目标函数为
min
∑
k
=
1
K
∑
x
∈
C
k
∥
x
−
μ
k
∥
2
\min\sum_{k=1}^K\sum_{x\in C_k}\|x-\mu_k\|^2
mink=1∑Kx∈Ck∑∥x−μk∥2
其中
μ
k
\mu_k
μk是簇
C
k
C_k
Ck的中心(centroid)向量
μ
k
=
1
N
k
∑
x
∈
C
k
x
\mu_k=\frac{1}{N_k}\sum_{x\in C_k}x
μk=Nk1x∈Ck∑x
直观的看相似样本被划到同一个簇,损失函数最小。但这是一个NP-hard问题,因此K-means采用迭代策略来近似求解。
K-Means的思想十分简单,首先随机指定类中心,根据样本与类中心的距离划分类簇,接着重新计算类中心,迭代直至收敛。

算法中使用到的距离可以是任何的距离计算公式,最常用的是欧氏距离,应用时具体应该选择哪种距离计算方式,需要根据具体场景确定。
聚类数k的选择:K-Means中的类别数需要预先指定,而在实际中,最优的
K
K
K值是不知道的,解决这个问题的一个方法是肘部法则(Elbow method):尝试用不同的
K
K
K值聚类,计算损失函数,拐点处即为推荐的聚类数 (即通过此点后,聚类数的增大也不会对损失函数的下降带来很大的影响,所以会选择拐点) 。但是也有损失函数随着$
K
K
K的增大平缓下降的例子,此时通过肘部法则选择
K
K
K的值就不是一个很有效的方法了。

优缺点小结
K-Means是个简单实用的聚类算法,这里对K-Means的优缺点做一个总结。
K-Means的主要优点有:
- 原理比较简单,实现也是很容易,收敛速度快(在大规模数据集上收敛较慢,可尝试使用Mini Batch K-Means算法)。
- 聚类效果较优。
- 算法的可解释度比较强。
- 主要需要调参的参数仅仅是簇数k。
K-Means的主要缺点有:
- K值的选取不好把握(实际中K值的选定是非常难以估计的,很多时候,事先并不知道给定的数据集应该分成多少个类别才最合适)。
- 对于不是凸的数据集比较难收敛(密度的聚类算法更加适合,比如DBSCAN算法,后面再介绍DBSCAN算法)
- 如果各隐含类别的数据不平衡,比如各隐含类别的数据量严重失衡,或者各隐含类别的方差不同,则聚类效果不佳。
- 采用迭代方法,得到的结果只是局部最优。(可以尝试采用二分K-Means算法)
- 对噪音和异常点比较敏感。(改进1:离群点检测的LOF算法,通过去除离群点后再聚类,可以减少离群点和孤立点对于聚类效果的影响;改进2:改成求点的中位数,这种聚类方式即K-Mediods聚类)
- 对初始值敏感(初始聚类中心的选择,可以尝试采用二分K-Means算法或K-Means++算法)
- 时间复杂度高 O ( N K t ) O(NKt) O(NKt),其中N是对象总数,K是簇数,t是迭代次数。
- 只适用于数值型数据,只能发现球型类簇。
K-means++
K-means 初始化质心位置的选择对最后的聚类结果和运行时间都有很大的影响,因此需要选择合适的K个质心。
K-Means++算法就是对K-Means随机初始化质心的方法的优化。K-Means++算法在聚类中心的初始化过程中使得初始的聚类中心之间的相互距离尽可能远,这样可以避免出现上述的问题。
二分K-means
为克服K-Means算法收敛于局部最小的问题,有人提出了另一种称为二分K-Means(bisecting K-Means)的算法。该算法首先将所有点作为一个簇,然后将该簇一分为二。之后选择其中一个簇继续进行划分,直到产生
K
K
K 个簇为止。
至于簇的分裂准则,取决于对其划分的时候可以最大程度降低 SSE(平方和误差)的值。

Elkan K-Means
在传统的K-Means算法中,我们在每轮迭代时,要计算所有的样本点到所有的质心的距离,这样会比较的耗时。Elkan K-Means算法利用了两边之和大于等于第三边,以及两边之差小于第三边的三角形性质,来减少不必要的距离的计算。
- 对于一个样本点x和两个质心 μ 1 \mu_1 μ1、 μ 2 \mu_2 μ2,如果我们预先计算出这两个质心之间的距离 d ( μ 1 , μ 1 ) d(\mu_1,\mu_1) d(μ1,μ1),如果发现 2 d ( x , μ 1 ) ≤ d ( μ 1 , μ 2 ) 2d(x,\mu_1) \leq d(\mu_1,\mu_2) 2d(x,μ1)≤d(μ1,μ2),那么我们便能得到 d ( x , μ 1 ) ≤ d ( x , μ 2 ) d(x,\mu_1)\leq d(x,\mu_2) d(x,μ1)≤d(x,μ2)。此时我们不再计算 d ( x , μ 2 ) d(x,\mu_2) d(x,μ2),也就节省了一步距离计算。
- 对于一个样本点x和两个质心 μ 1 \mu_1 μ1、 μ 2 \mu_2 μ2,我们能够得到 d ( x , μ 2 ) ≤ max { 0 , d ( x , μ 1 ) − d ( μ 1 , μ 2 ) } d(x,\mu_2)\leq \max{0,d(x,\mu_1)-d(\mu_1,\mu_2)} d(x,μ2)≤max{0,d(x,μ1)−d(μ1,μ2)}。这个从三角形的性质也很容易得到。
Elkan K-Means迭代速度比传统K-Means算法迭代速度有较大提高,但如果我们的样本特征是稀疏的,或者有缺失值的话,此种方法便不再使用。
Mean Shift
占位
ISODATA
DBSCAN
K-Means算法是基于距离的聚类算法,当数据集中的聚类结果是非球状结构时,聚类效果并不好。而基于密度的聚类算法可以发现任意形状的聚类,它是从样本密度的角度来考察样本之间的可连接性,并基于可连接性(密度可达)不断扩展聚类簇以获得最终的聚类结果.。
基本概念
DBSCAN(Density-Based Spatial Clustering of Application with Noise)是一种典型的基于密度的聚类算法,它基于一组邻域参数
(
ϵ
,
M
i
n
P
t
s
)
(\epsilon,MinPts)
(ϵ,MinPts) 刻画样本分布的紧密程度。
在DBSCAN算法中将数据点分为三类:
- 核心点(Core point):若样本 x i x_i xi 的 ϵ \epsilon ϵ 邻域内至少包含了MinPts 个样本,即 N ϵ ( x i ) ⩾ M i n P t s N_\epsilon(x_i)\geqslant MinPts Nϵ(xi)⩾MinPts,则称样本点 x i x_i xi 为核心点。
- 边界点(Border point):若样本 x i x_i xi 的 ϵ \epsilon ϵ 邻域内包含的样本数目小于MinPts,但是它在其他核心点的邻域内,则称样本点 x i x_i xi 为边界点。
- 噪音点(Noise):既不是核心点也不是边界点的点。
在这里有两个量,一个是半径 Eps (
ϵ
\epsilon
ϵ),另一个是指定的数目 MinPts。
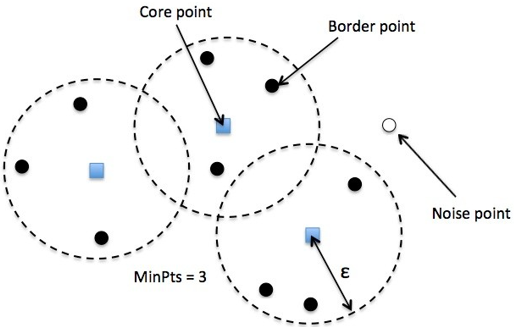
在DBSCAN算法中,由核心对象出发,找到与该核心对象密度可达的所有样本集合形成簇。
在DBSCAN算法中,还定义了如下一些概念:
- 密度直达(directly density-reachable):若样本点 x j x_j xj 在核心点 x i x_i xi 的邻域内( x j ∈ U ( x i , ϵ ) x_j\in U(x_i,\epsilon) xj∈U(xi,ϵ)),则称 x j x_j xj 由 x i x_i xi 密度直达。
- 密度可达(density-reachable):存在样本序列 p 1 , p 2 , ⋯ , p n p_1,p_2,\cdots,p_n p1,p2,⋯,pn,若样本点 p i + 1 p_{i+1} pi+1 由 p i p_i pi 密度直达,则称 p n p_n pn 由 p 1 p_1 p1 密度可达。
- 密度相连(density-connected):若样本 x i x_i xi 和 x j x_j xj 均可由核心点 p p p 密度可达,则称样本 x i x_i xi 和 x j x_j xj 密度相连。
因此,DBSCAN的一个簇为最大密度相连的样本集合。该算法可在具有噪声的空间数据库中发现任意形状的簇。
算法流程
DBSCAN算法的流程为:
- 根据给定的邻域参数Eps和MinPts确定所有的核心对象;
- 选择一个未处理过的核心对象,找到由其密度可达的的所有样本生成聚类簇。
- 重复以上过程,直到所有的核心对象都遍历完。
下图与原来的DBSCAN算法用了相同的概念并发现相同的簇,但为了简洁而不是有效,做了一些调整。
优缺点小结
优点:
- 相比K-Means,DBSCAN 不需要预先声明聚类数量。
- 可以对任意形状的稠密数据集进行聚类,相对的,K-Means之类的聚类算法一般只适用于凸数据集。
- 可以在聚类的同时发现异常点,对数据集中的异常点不敏感。
- 聚类结果没有偏倚,相对的,K-Means之类的聚类算法初始值对聚类结果有很大影响。
缺点:
- 当空间聚类的密度不均匀、聚类间距相差很大时,聚类质量较差,因为这种情况下参数MinPts和Eps选取困难。
- 如果样本集较大时,聚类收敛时间较长,此时可以对搜索最近邻时建立的KD树或者球树进行规模限制来改进。
- 在两个聚类交界边缘的点会视它在数据库的次序决定加入哪个聚类,幸运地,这种情况并不常见,而且对整体的聚类结果影响不大(DBSCAN变种算法,把交界点视为噪音,达到完全决定性的结果。)
- 调参相对于传统的K-Means之类的聚类算法稍复杂,主要需要对距离阈值eps,邻域样本数阈值MinPts联合调参,不同的参数组合对最后的聚类效果有较大影响。
HDBSCAN
占位
OPTICS
在前面介绍的DBSCAN算法中,有两个初始参数Eps(邻域半径)和minPts(Eps邻域最小点数)需要手动设置,并且聚类的结果对这两个参数的取值非常敏感。为了克服DBSCAN算法这一缺点,提出了OPTICS算法(Ordering Points to identify the clustering structure),翻译过来就是,对点排序以此来确定簇结构。
OPTICS是对DBSCAN的一个扩展算法,该算法可以让算法对半径Eps不再敏感。只要确定minPts的值,半径Eps的轻微变化,并不会影响聚类结果。OPTICS并不显式的产生结果类簇,而是为聚类分析生成一个增广的簇排序(比如,以可达距离为纵轴,样本点输出次序为横轴的坐标图),这个排序代表了各样本点基于密度的聚类结构。它包含的信息等价于从一个广泛的参数设置所获得的基于密度的聚类,换句话说,从这个排序中可以得到基于任何参数Eps和minPts的DBSCAN算法的聚类结果。
要搞清楚OPTICS算法,需要搞清楚2个新的定义:核心距离和可达距离。
核心距离 (core distance):一个对象
P
P
P的核心距离是使得其成为核心对象的最小半径,即对于集合
M
M
M而言,假设让其中
x
x
x点作为核心,找到以
x
x
x点为圆心,且刚好满足最小邻点数minPts的最外层的一个点为
x
′
x'
x′,则
x
x
x点到
x
′
x'
x′点的距离称为核心距离
c
d
(
x
)
=
d
(
x
,
U
ϵ
(
x
)
)
,
if
N
ϵ
(
x
)
⩾
m
i
n
P
t
s
cd(x)=d(x,U_\epsilon(x)),\quad\text{if }N_\epsilon(x)\geqslant minPts
cd(x)=d(x,Uϵ(x)),if Nϵ(x)⩾minPts
可达距离(reachability distance):是根据核心距离来定义的。对于核心点
x
x
x的邻点
x
1
,
x
2
,
⋯
,
x
n
x_1,x_2,\cdots,x_n
x1,x2,⋯,xn 而言:
如果他们到点 x x x的距离大于点 x ′ x' x′的核心距离,则其可达距离为该点到点 x x x的实际距离;
如果他们到点 x x x的距离核心距离小于点 x ′ x' x′的核心距离的话,则其可达距离就是点 x x x的核心距离,即在 x ′ x' x′以内的点到 x x x的距离都以此核心距离来记录。
r d ( y , x ) = max { c d ( x ) , d ( x , y ) } , if N ϵ ( x ) ⩾ m i n P t s rd(y,x)=\max\{cd(x),d(x,y)\},\quad\text{if }N_\epsilon(x)\geqslant minPtsrd(y,x)=max{cd(x),d(x,y)},if Nϵ(x)⩾minPts
举例,下图中假设
m
i
n
P
t
s
=
3
minPts=3
minPts=3,半径是
ϵ
\epsilon
ϵ。那么
P
P
P点的核心距离是
d
(
1
,
P
)
d(1,P)
d(1,P),点2的可达距离是
d
(
1
,
P
)
d(1,P)
d(1,P),点3的可达距离也是
d
(
1
,
P
)
d(1,P)
d(1,P),点4的可达距离则是
d
(
4
,
P
)
d(4,P)
d(4,P)。

OPTICS的核心思想:
- 较稠密簇中的对象在簇排序中相互靠近;
- 一个对象的最小可达距离给出了一个对象连接到一个稠密簇的最短路径。
每个团簇的深浅代表了团簇的紧密程度,我们可以在这基础上采用DBSCAN(选取最优的Eps)或层次聚类方法对数据进行聚类。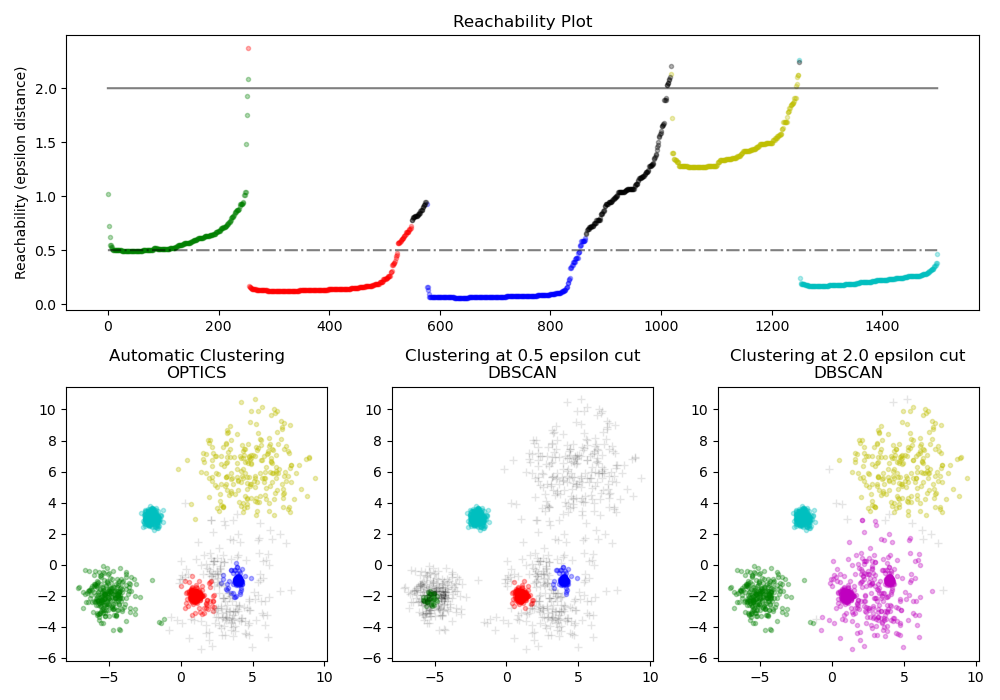
层次聚类
层次聚类(hierarchical clustering) 假设簇之间存在层次结构,后面一层生成的簇基于前面一层的结果。层次聚类算法一般分为两类:
- 自顶向下(top-down)的分拆(divisive)层次聚类:开始将所有样本分到一个类,之后将己有类中相距最远的样本分到两个新的类,重复此操作直到满足停止条件(如每个样本均为一类)。
- 自底向上(bottom-up)的聚合(Agglomerative)层次聚类:开始将每个样本各自分到一个类,之后将相距最相近的两个类合并生成一个新的类,重复此操作直到满足停止条件(如所有样本聚为一类)。
类之间的距离
除了需要衡量样本之间的距离之外,层次聚类算法还需要衡量类之间的距离,称为 linkage。
假设
C
i
C_i
Ci和
C
j
C_j
Cj为两个类,常见的类之间距离的衡量方法有

最短距离或单连接(single-linkage):两个类的最近样本间的距离
d
(
C
i
,
C
j
)
=
min
{
d
(
x
,
y
)
∣
x
∈
C
i
,
y
∈
C
j
}
d(C_i, C_j)=\min\{ d(x, y)|x \in C_i, y \in C_j \}
d(Ci,Cj)=min{d(x,y)∣x∈Ci,y∈Cj}
最长距离或完全连接(complete-linkage):两个类的最远样本间的距离
d
(
C
i
,
C
j
)
=
max
{
d
(
x
,
y
)
∣
x
∈
C
i
,
y
∈
C
j
}
d(C_i, C_j) = \max\{ d(x, y)|x \in C_i, y \in C_j \}
d(Ci,Cj)=max{d(x,y)∣x∈Ci,y∈Cj}
平均距离(average-linkage):两个类所有样本间距离的平均值
d
(
C
i
,
C
j
)
=
1
N
i
N
j
∑
x
∈
C
i
∑
y
∈
C
j
d
(
x
,
y
)
d(C_i,C_j)=\frac{1}{N_iN_j}\sum_{x\in C_i}\sum_{y\in C_j}d(x,y)
d(Ci,Cj)=NiNj1x∈Ci∑y∈Cj∑d(x,y)
AGNES
AGNES 是一种采用自底向上聚合策略的层次聚类算法,它先将数据集中的每个样本看作一个初始聚类簇,然后在每一步中找出距离最近的两个类簇进行合并,该过程不断重复,直至达到预设的类簇个数。

BIRCH
占位
Ward 方法
Ward方法 提出的动机是最小化每次合并时的信息损失。 SSE 作为衡量信息损失的准则:
S
S
E
(
C
)
=
∑
x
∈
C
(
x
−
μ
C
)
T
(
x
−
μ
C
)
SSE(C) = \sum_{x \in C}(x-\mu_C)^T(x-\mu_C)
SSE(C)=x∈C∑(x−μC)T(x−μC)
其中
μ
C
\mu_C
μC为簇
C
C
C 中样本点的均值
μ
C
=
1
N
C
∑
x
∈
C
x
\mu_C=\frac{1}{N_C}\sum_{x\in C}x
μC=NC1x∈C∑x
可以看到 SSE 衡量的是一个类内的样本点的聚合程度,样本点越聚合,SSE 的值越小。Ward 方法则是希望找到一种合并方式,使得合并后产生的新的一系列的类的 SSE 之和相对于合并前的类的 SSE 之和的增长最小。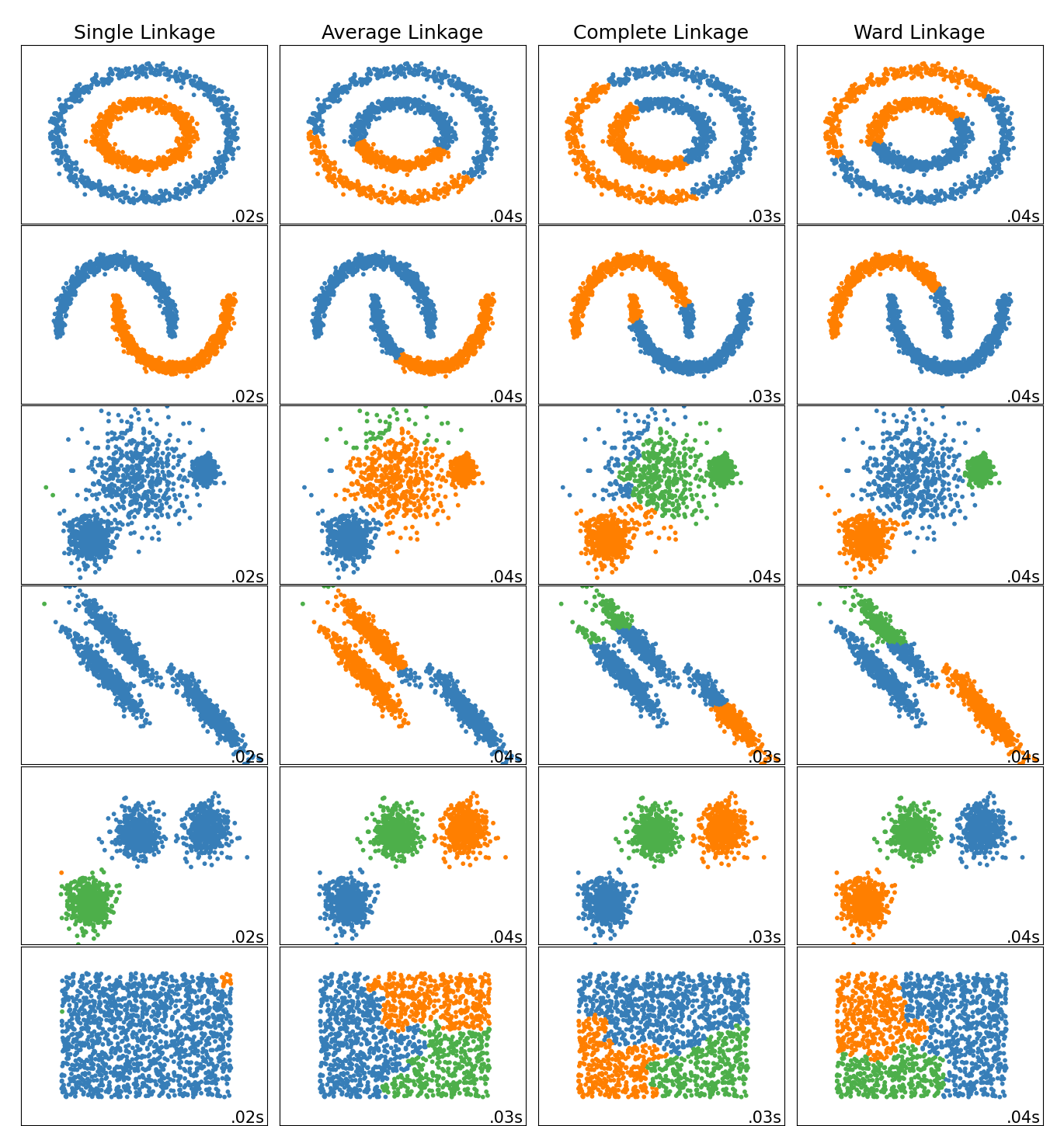
高斯混合聚类
高斯混合聚类是基于概率模型的贝叶斯定理来表达聚类原型,将在高斯混合模型章节详细介绍。
Affinity Propagation

谱聚类
Spectral clustering
聚类性能评估
在学习聚类算法得时候并没有涉及到评估指标,主要原因是聚类算法属于非监督学习,并不像分类算法那样可以使用训练集或测试集中的数据计算准确率、召回率等。那么如何评估聚类算法得好坏呢?好的聚类算法一般要求类簇具有:
- 高的类内 (intra-cluster) 相似度
- 低的类间 (inter-cluster) 相似度
对于聚类算法大致可分为两类度量标准:内部指标和外部指标。
内部指标
内部指标(internal index)即基于数据聚类自身进行评估,不依赖任何外部模型。如果某个聚类算法的结果是类间相似性低,类内相似性高,那么内部指标会给予较高的分数评价。
SSE
SSE:(Sum of Squared Error)衡量的是一个类内的样本点的聚合程度。SSE值越小表示数据点越接近他们的质心,聚类效果也最好。
S
S
E
=
∑
i
=
1
K
∑
x
∈
C
i
d
2
(
μ
i
,
x
)
SSE = \sum_{i=1}^K\sum_{x\in C_i}{d^2(\mu_i,x)}
SSE=i=1∑Kx∈Ci∑d2(μi,x)
轮廓系数
轮廓系数(Silhouette Coefficient)适用于实际类别信息未知的情况。对于单个样本,设a是与它同类别中其他样本的平均距离,b是与它距离最近不同类别中样本的平均距离,其轮廓系数为:
s
=
b
−
a
max
{
a
,
b
}
s=\frac{b-a}{\max\{a,b\}}
s=max{a,b}b−a
对于一个样本集合,它的轮廓系数是所有样本轮廓系数的平均值。轮廓系数的取值范围是[-1,1],同类别样本距离越相近不同类别样本距离越远,分数越高。缺点:不适合基高密度的聚类算法DBSCAN。
实际应用中,查查把 SSE(Sum of Squared Errors,误差平方和) 与轮廓系数(Silhouette Coefficient)结合使用,评估聚类模型的效果。
Calinski-Harabasz Index
Calinski-Harabasz定义为簇间离散均值与簇内离散均值之比。通过计算类中各点与类中心的距离平方和来度量类内离散,通过计算各类中心点与数据集中心点距离平方和来度量数据集的离散度,CHI越大代表着类自身越紧密,类与类之间越分散,即更优的聚类结果。
C
H
I
=
tr
(
B
k
)
tr
(
W
k
)
⋅
N
−
k
k
−
1
CHI=\frac{\text{tr}(B_k)}{\text{tr}(W_k)}\cdot\frac{N-k}{k-1}
CHI=tr(Wk)tr(Bk)⋅k−1N−k
其中N为训练样本数,k是类别个数,
B
k
B_k
Bk是类别之间协方差矩阵,
W
k
W_k
Wk是类别内部数据协方差矩阵。
B
k
=
∑
i
=
1
k
∑
x
∈
C
i
d
2
(
x
,
μ
i
)
W
k
=
∑
i
=
1
k
N
i
d
2
(
μ
i
,
μ
X
)
B_k=\sum_{i=1}^k\sum_{x\in C_i} d^2(x,\mu_i) \\ W_k=\sum_{i=1}^kN_id^2(\mu_i,\mu_X)
Bk=i=1∑kx∈Ci∑d2(x,μi)Wk=i=1∑kNid2(μi,μX)
CHI数值越小可以理解为:组间协方差很小,组与组之间界限不明显。
Davies-Bouldin Index
DB指数计算任意两类别的类内平均距离(CP)之和除以两类中心距离求最大值。该指标的计算公式:
D
B
I
=
1
K
∑
i
=
1
K
max
i
≠
j
s
i
+
s
j
d
(
C
i
,
C
j
)
DBI=\frac{1}{K}\sum_{i=1}^K\max_{i\neq j}\frac{s_i+s_j}{d(C_i,C_j)}
DBI=K1i=1∑Ki=jmaxd(Ci,Cj)si+sj
其中K是类别个数,
s
i
s_i
si是类
C
i
C_i
Ci中所有的点到类中心的平均距离,
d
(
C
i
,
C
j
)
d(C_i,C_j)
d(Ci,Cj)是类
C
i
,
C
j
C_i,C_j
Ci,Cj中心点间的距离。DBI越小意味着类内距离越小同时类间距离越大。缺点:因使用欧式距离所以对于环状分布聚类评测很差。
外部指标
外部指标(external index)通过是通过使用没被用来做训练集的数据进行评估。例如已知样本点的类别信息和一些外部的基准。这些基准包含了一些预先分类好的数据,比如由人基于某些场景先生成一些带label的数据,因此这些基准可以看成是金标准。这些评估方法是为了测量聚类结果与提供的基准数据之间的相似性。然而这种方法也被质疑不适用真实数据。
Rand index
兰德指数(Rand index, RI)可认为是计算精确率,因此我们借用二分类问题的混淆矩阵概念:
Positive 包括 TP 和 TN
Negative 包括 FP 和 FN
R I = T P + T N T P + F P + T N + F N RI=\frac{TP+TN}{TP+FP+TN+FN}RI=TP+FP+TN+FNTP+TN
RI取值范围为 [0,1],值越大意味着聚类结果与真实情况越吻合。
对于随机结果,RI并不能保证分数接近零。为了实现“在聚类结果随机产生的情况下,指标应该接近零”,调整兰德系数(Adjusted rand index)被提出,它具有更高的区分度:
A
R
I
=
R
I
−
E
[
R
I
]
max
R
I
−
E
[
R
I
]
ARI=\frac{RI-\mathbb E[RI]}{\max RI-\mathbb E[RI]}
ARI=maxRI−E[RI]RI−E[RI]
ARI取值范围为[-1,1],值越大意味着聚类结果与真实情况越吻合。从广义的角度来讲,ARI衡量的是两个数据分布的吻合程度。
互信息
互信息(Mutual Information)是用来衡量两个数据分布的吻合程度。有两种不同版本的互信息以供选择,一种是Normalized Mutual Information(NMI),一种是Adjusted Mutual Information(AMI)。
假设U与V是对N个样本标签的分配情况,
N
M
I
(
U
,
V
)
=
M
I
(
U
,
V
)
(
H
(
U
)
+
H
(
V
)
)
/
2
NMI(U,V)=\frac{MI(U,V)}{(H(U)+H(V))/2}
NMI(U,V)=(H(U)+H(V))/2MI(U,V)
其中,MI表示互信息,H为熵。
A
M
I
=
M
I
−
E
[
M
I
]
(
H
(
U
)
+
H
(
V
)
)
/
2
−
E
[
R
I
]
AMI=\frac{MI-\mathbb E[MI]}{(H(U)+H(V))/2-\mathbb E[RI]}
AMI=(H(U)+H(V))/2−E[RI]MI−E[MI]
利用基于互信息的方法来衡量聚类效果需要实际类别信息,MI与NMI取值范围为[0,1],AMI取值范围为[-1,1],它们都是值越大意味着聚类结果与真实情况越吻合。
V-measure
说V-measure之前要先介绍两个指标:
- 同质性(homogeneity):每个群集只包含单个类的成员。
- 完整性(completeness):给定类的所有成员都分配给同一个群集。
同质性和完整性分数基于以下公式得出:
h
=
1
−
H
(
C
∣
K
)
H
(
C
)
c
=
1
−
H
(
K
∣
C
)
H
(
K
)
h=1-\frac{H(C|K)}{H(C)} \\ c=1-\frac{H(K|C)}{H(K)}
h=1−H(C)H(C∣K)c=1−H(K)H(K∣C)
V-measure是同质性和完整性的调和平均数:
v
=
2
h
c
h
+
c
v=\frac{2hc}{h+c}
v=h+c2hc
V-measure 实际上等同于互信息 (NMI)。取值范围为 [0,1],越大越好,但当样本量较小或聚类数据较多的情况,推荐使用AMI和ARI。
Fowlkes-Mallows scores
Fowlkes-Mallows Scores(FMI) 是成对的precision(精度)和recall(召回)的几何平均数。取值范围为 [0,1],越接近1越好。定义为:
F
M
I
=
T
P
(
T
P
+
F
P
)
(
T
P
+
F
N
)
FMI=\frac{TP}{\sqrt{(TP+FP)(TP+FN)}}
FMI=(TP+FP)(TP+FN)TP
版权归原作者 WilenWu 所有, 如有侵权,请联系我们删除。