一、背景
BRATS(Brain Tumor Segmentation)是一个公开的医学图像分割挑战赛,旨在促进脑肿瘤的自动分割和治疗规划技术的发展。该挑战赛已经举办多年,是学习医学图像分割最前沿的平台之一。
二、BRATS 2021
BRATS 2021数据集是一个医学图像分割数据集,该数据集由2000例患者脑部MRI(核磁共振成像)扫描构成。其中训练集有1251例、验证集219例、测试集530例,每例MRI扫描有4个模态的3D图像。其中训练集是包含3D图像和分割标签的,而验证集和测试集则不包含分割标签,验证集被用于公共排行榜,测试集不公开,用作参赛者的最终排名评测。
训练集其中一例MRI扫描(包含4个模态的3D图像以及1个共享标签)
三、数据详解
1、在训练集中包含1251例核磁共振成像扫描,每例扫描包含4种模态的3D图像以及1个共享标签,其中4种模态分别是t1、t1ce、t2、flair:
T1加权成像(T1-weighted imaging, **T1**):这种成像方式通过对人体进行磁共振成像,使用对T1信号敏感的成像方法,呈现出人体组织的强度。T1序列主要用于显示解剖结构。 T1对比增强成像(T1-weighted imaging with contrast enhancement, **T1CE**):这种成像方式是在T1序列的基础上加入了造影剂,能够帮助检测到肿瘤和炎症等病变。T1CE序列主要用于显示肿瘤和炎症等异常组织。 T2加权成像(T2-weighted imaging, **T2**):这种成像方式通过对人体进行磁共振成像,使用对T2信号敏感的成像方法,呈现出人体组织的水分布情况。T2序列主要用于显示病变的水肿或液体。 **FLAIR**(Fluid Attenuated Inversion Recovery)成像:这种成像方式通过将T2序列与T1序列结合起来,使用反转恢复脉冲序列,增强了病变和脑脊液的对比度。FLAIR序列主要用于显示肿瘤、炎症和其他异常组织。
BraTS2021_00002_t1.nii.gz
BraTS2021_00002_t1ce.nii.gz
BraTS2021_00002_t2.nii.gz
BraTS2021_00002_flair.nii.gz
其中共享标签(shared label)是指在所有模态中共享的标签或标注,包括四种分类标签[0、1、2、4],这些标签用于对肿瘤进行分割和分级,以帮助医生制定最佳治疗方案。共享标签表示在整个数据集中,不同模态的标签都是相同的,并且每个模态都会收到相同的标注,这有助于保证数据的一致性和可比性。此外,共享标签也有助于研究者比较不同算法在相同数据集上的性能,以及进一步提高肿瘤分割的准确性和可靠性。
BraTS2021_00002_seg.nii.gz
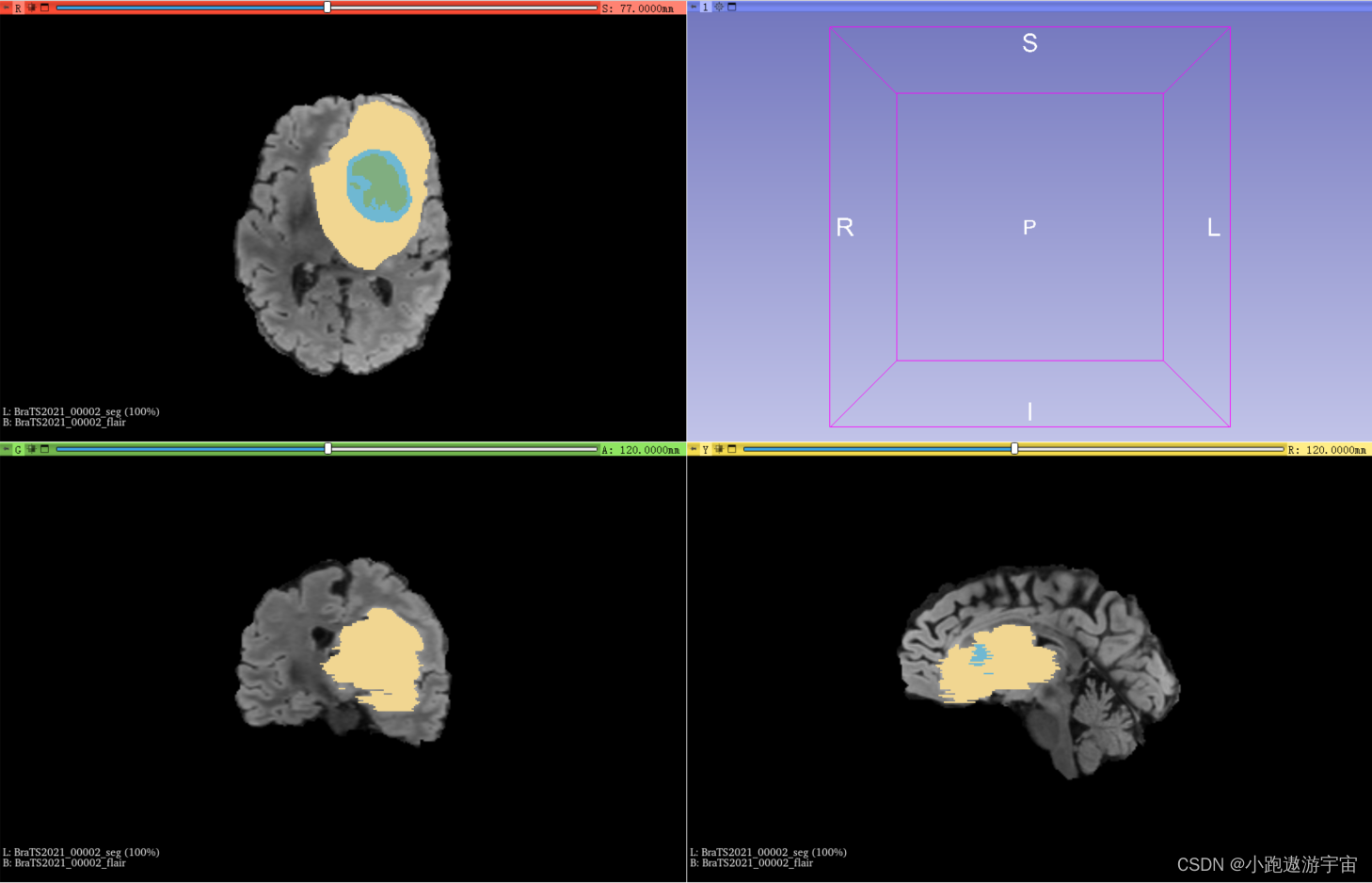
** label 0**:背景(Background)呈现黑色
** label 1**:坏死肿瘤核心(NCR, necrotic tumor core)呈现绿色 ** label 2**:瘤周水肿(ED,peritumoral edema)呈现淡金色 **label 4**:增强肿瘤(ET,enhancing tumor)呈现蓝色 根据比赛要求按区域进行分割,最终需识别出三个子区域:增强肿瘤**ET**(只有label4)、肿瘤核心**TC**(包括label1、4)、整个肿瘤**WT**(包括label1、2、4)。2、每个模态都是核磁共振成像,大小都为 240 x 240 x 155(长 宽 高)的3D图像,可以理解成一个240 x 240的二维切片沿z轴叠加成的155层三维立体。每个二维切片都是由一个二维矩阵表示的,每个像素点包含在x、y、z轴上的位置信息和该点的强度值。该点的强度值指的是该点的灰度强度,取值范围一般为0-255。
3、4个模态的3D图像以及共享标签都是以 .nii 文件格式存储在 .nii.gz 压缩包中,对于一个.nii.gz文件,它包含的数据可以分为两部分:头文件(Header)和图像数据(Image Data)。头文件包含了一些描述数据集的元数据,例如数据集的大小、数据类型、空间分辨率、坐标系等等;而图像数据则是由一系列二维切片构成的三维图像数据。三维图像数据以体素(Voxel)为单位储存,每个体素包含在三个坐标轴上的位置信息和该点的强度值,这些体素可以看作是三维空间中的像素点,因此也被称为“三维像素”。这三个坐标轴分别为x轴、y轴和z轴,它们的方向和空间分辨率都可以在头文件中指定。例如,对于一个大小为240x240x155的.nii.gz文件,其中的一个体素可以表示为(10, 20, 30, 128),表示该体素在x轴上的位置为10,在y轴上的位置为20,在z轴上的位置为30,其强度值为128。这个体素可以在三维空间中表示为一个立方体,其边长为该.nii.gz文件的空间分辨率,例如,如果空间分辨率为1mm,那么该立方体的边长为1mm,它的位置为(x, y, z),其颜色或灰度强度为该体素的强度值。通过这种方式,每个.nii.gz文件都可以被看作是一个由体素构成的三维立体,每个体素包含在三个坐标轴上的位置信息和该点的强度值,这些信息可以用来表示不同组织和结构在三维空间中的分布和形态。
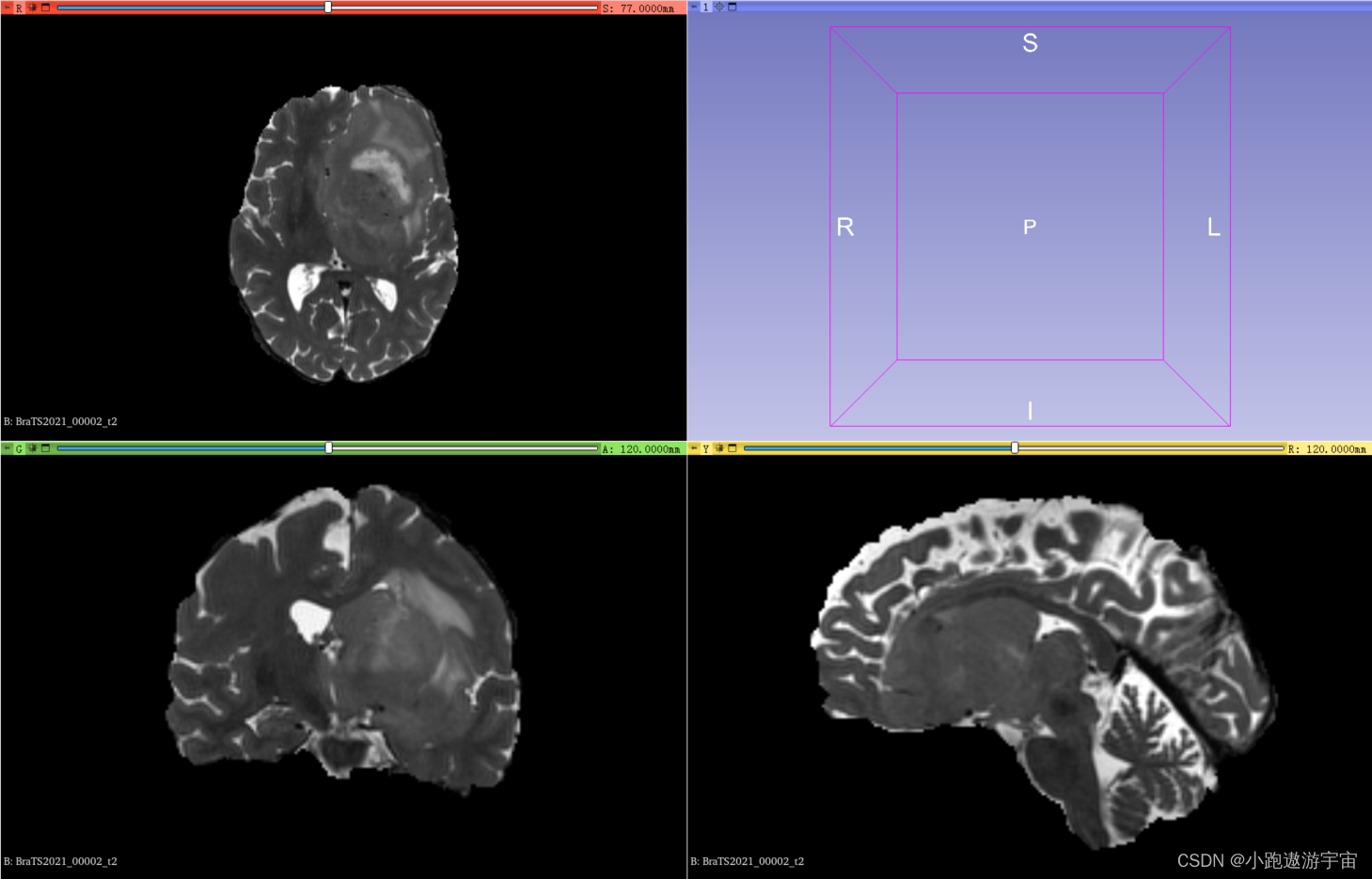
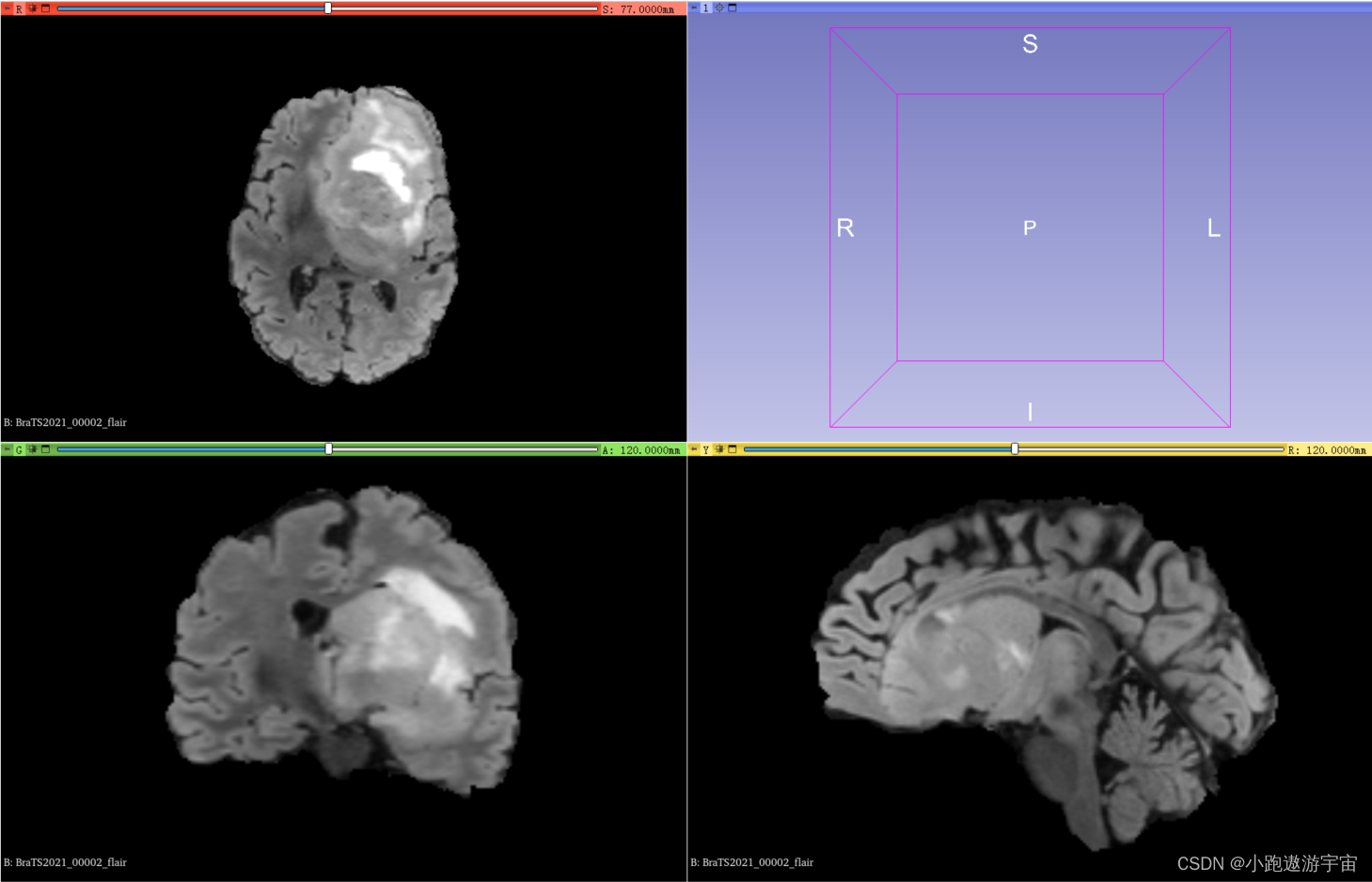
4、可视化使用3D Slicer查看图像和标签,可以直观的了解自己要用的数据集。左上角代表在Axial(轴向)视角下,可以查看由上到下的脑部二维切片;左下角代表在Coronal(冠状)视角下,可以查看由前到后的脑部二维切片;右下角代表在Sagittal(矢状)视角下,可以查看由左到右的脑部二维切片。
5、用3D Slicer打开BraTS2021_00002_flair.nii.gz,紧接着打开BraTS2021_00002_seg.nii.gz,可以看到在FLAIR成像时所标注的标签,其他模态亦是如此。
四、其他理解
1、如何理解一个由像素组成的二维矩阵?
一个由像素组成的二维矩阵可以理解为一个由数字组成的矩形网格,其中每个数字代表该位置的像素值。在医学影像学中,这些像素值通常表示该位置的信号强度,例如在MRI扫描中,信号强度可以反映组织的密度、水含量、磁共振性质等信息。 对于灰度图像,每个像素值通常是一个介于0到255之间的整数,其中0代表黑色,255代表白色,其他的数字则代表不同灰度级别的灰色。因此,一个由像素组成的二维矩阵可以看作是一个灰度图像的数字表示,它可以通过对像素值进行操作和处理来实现对图像的各种分析和处理。 在医学影像学中,常常使用的是三维矩阵,即由像素组成的立体网格。一个三维矩阵可以看作是一组由二维矩阵组成的立方体,其中每个像素值表示该位置的信号强度。类似地,可以对三维矩阵进行各种图像处理操作,如分割、配准、重建等。2、可视化.nii.gz文件时为什么可以在三个视角看到?
.nii.gz文件是医学影像中常用的数据格式之一,通常包含了三维图像数据,即由一系列的二维切片(或称为“层”)构成的图像。因此,在可视化.nii.gz文件时,可以通过不同的视角(或称为“面”)来查看不同的二维切片,从而观察到整个三维图像。 具体地说,在常见的医学图像处理软件中,如ITK、Nibabel或SimpleITK,可以通过改变三维图像的“切面”来实现不同视角的查看。例如,在Axial(轴向)视角下,可以查看由上到下的二维切片,即沿着脑部的水平平面进行切片;在Coronal(冠状)视角下,可以查看由前到后的二维切片,即沿着脑部的冠状面进行切片;在Sagittal(矢状)视角下,可以查看由左到右的二维切片,即沿着脑部的矢状面进行切片。 因此,通过在不同的视角下查看.nii.gz文件,可以获得不同的图像信息,并且可以更全面地了解医学影像的特征和结构。3、.nii.gz文件由一系列的二维切片(或称为“层”)构成的图像,那么为什么每个视角都能切片呢?
具体来说,切片过程实质上是将三维体数据中某个方向上的所有像素点排列成一个二维矩阵,然后将该矩阵作为一个二维图像来展示。不同方向的切片所对应的就是不同方向上的二维矩阵,因此可以通过改变视角来切换不同方向的切片,以查看不同方向上的图像。4、举个例子分别说明二维图像的像素点和三维空间的像素点?
假设我们有一个二维图像,大小为5x5像素,其像素点的坐标范围为x=[1,5], y=[1,5]。我们可以用一个二维矩阵来表示该图像,其中每个元素代表一个像素点的灰度值。[ 0 128 255 128 0 ] [128 255 128 255 128 ] [255 128 255 128 255 ] [128 255 128 255 128 ] [ 0 128 255 128 0 ]其中,每个数字代表该像素点的灰度值,取值范围一般为0-255。 现在,我们将这个二维图像想象成一个物体,该物体在三维空间中有一个位置和方向。我们可以将这个物体想象成一张厚度很小的纸片,在三维空间中的位置和方向决定了我们从哪个方向来观察该物体,从而得到不同的二维切片图像。 假设我们从Axial(轴向)视角来观察该物体,也就是从上往下看,那么我们可以将该物体切成一系列的二维切片,每个切片对应一个二维图像。这些切片沿z轴(高度方向)排列,其z坐标范围为z=[1,1]。每个切片对应的二维矩阵可以看做是从该物体的顶部往下看的一个二维图像,其中每个像素点的灰度值仍然表示该点的颜色强度。 类似地,如果我们从Coronal(冠状)视角或Sagittal(矢状)视角来观察该物体,那么就会得到不同方向上的二维切片图像,每个切片对应的二维矩阵也会有所不同。 需要注意的是,二维图像中的像素点只有x和y坐标,而没有z坐标,因为二维图像只有两个维度。而在三维空间中,每个像素点除了有x和y坐标,还有一个z坐标,表示该点在三维空间中的高度。因此,在处理三维图像时,需要考虑到像素点在三维空间中的位置信息。5、强度值指的是什么,具体解释一下?
在医学图像领域中,.nii.gz文件中的强度值通常指的是图像中每个像素点或体素的灰度值或颜色值。在计算机视觉领域,强度值也可以指像素点的RGB值或其他颜色空间的值。 对于MRI或CT图像,强度值反映了组织或结构在该点处对应的物理性质。在MRI图像中,强度值通常反映了组织或结构在该点处的T1或T2信号强度,也就是其对应的磁共振信号强度。例如,对于脑部MRI图像中的白质和灰质组织,在T1加权图像中,白质通常表现为较高的信号强度,而灰质则表现为较低的信号强度。在CT图像中,强度值通常反映了组织或结构在该点处的X射线吸收程度,也就是其对应的CT值。 因此,在医学图像中,强度值通常被用来表示不同组织或结构的特定物理性质,具有重要的临床意义。通过对这些强度值的分析和处理,可以帮助医生判断疾病的类型和严重程度,制定治疗方案和手术计划等。6、强度值的范围?
强度值的范围取决于具体的医学图像和采集设备,不同的图像类型和设备可能有不同的强度值范围。 以MRI图像为例,一般情况下,MRI图像中的强度值是一个无符号的整数,通常在0到255之间,代表不同灰度级别。不同MRI序列的强度值范围可能会有所不同,例如T1加权图像中的白质信号强度通常在150到250之间,而灰质信号强度通常在50到120之间。T2加权图像中的白质信号强度通常在200到250之间,而灰质信号强度通常在70到120之间。 对于CT图像,强度值通常也是一个无符号的整数,范围在-1024到+3071之间,代表不同的CT值。在CT图像中,不同的组织和结构对应不同的CT值,例如骨骼和钙化物质通常具有较高的CT值,而软组织和液体则具有较低的CT值。 需要注意的是,不同的医学图像和采集设备可能会有不同的强度值范围和取值方式,因此在处理和分析医学图像时需要对不同的数据进行适当的预处理和标准化,以保证结果的可靠性和可重复性。7、强度值和RGB区别?
RGB需要三个0-255的分量来表示一个像素的颜色,而强度值只需要一个0-255的量来表示一个像素的灰度强度。因此,RGB图像中的一个像素由三个数值组成,而医学图像中的一个像素只有一个数值。 所以MRI和CT等医学图像中的像素强度值并不是用来表示颜色的,而是用来表示图像中不同组织或结构的密度、吸收、反射等特性。因此,虽然两者都是0-255的取值范围,但含义是不同的。 在医学图像中,像素的强度值通常与该像素所代表的组织或结构的特性相关,例如CT图像中的骨骼、软组织、肿瘤等,MRI图像中的脑脊液、白质、灰质等。因此,医学图像中的像素强度值通常用于定量分析、诊断和治疗等临床应用,而不是用于颜色表示。8、空间分辨率和二维分辨率的区别以及各自的作用?
空间分辨率(spatial resolution)和二维分辨率(2D resolution)是图像处理中的两个重要概念,它们都与图像的分辨率有关,但含义和作用略有不同。 空间分辨率指的是图像中最小可分辨的物理距离,通常用线对线距离或者点对点距离表示,也可以理解为图像中最小物理结构的大小。空间分辨率越高,表示图像中可分辨出的物体大小越小,图像细节也越多,图像清晰度也越高。在医学图像中,空间分辨率可以影响到图像诊断和治疗的准确性,因为它决定了医生在图像上能够看到多少细节和结构。 二维分辨率指的是图像在水平和垂直方向上的像素数目,也称为像素分辨率。它决定了图像在屏幕上显示的大小和清晰度。二维分辨率越高,图像显示越清晰,图像大小也越大。在数字图像处理中,二维分辨率也会影响到算法的效果和速度。 总的来说,空间分辨率关注图像的物理分辨率,二维分辨率则关注图像在计算机上的像素分辨率。两者都是影响图像质量的重要因素,需要根据不同的应用场景和需求进行选择和优化。
版权归原作者 小跑遨游宇宙 所有, 如有侵权,请联系我们删除。