文章目录
前言
临时接到一个分支任务,那就是解决TSP问题,来作为人工智能课程的期中测试。是的这不时巧了嘛,我Hou(第三声)恰好略懂一二。那么今天的话,咱们就用好几个方案来解决这个问题吧,首先是咱们的遗传算法,之后是咱的PSO算法,最后是咱们的一个衍生想法,就是使用强化学习来做(这里选取的是DQN,我们采用3个网络并行解决问题),同样我们分三篇博文说明。
TSP问题
那么在开始之前的话,咱们来仔细描述一下这个TSP问题。这个打过数模,或者接触过智能优化或者机器学习方面的朋友应该都知道,当然为了本文的受众普适性,咱们在这里尽可能去完善一点,说清楚,这样便于咱们去实际解决问题。
那么这个问题的其实简单,是这样子的: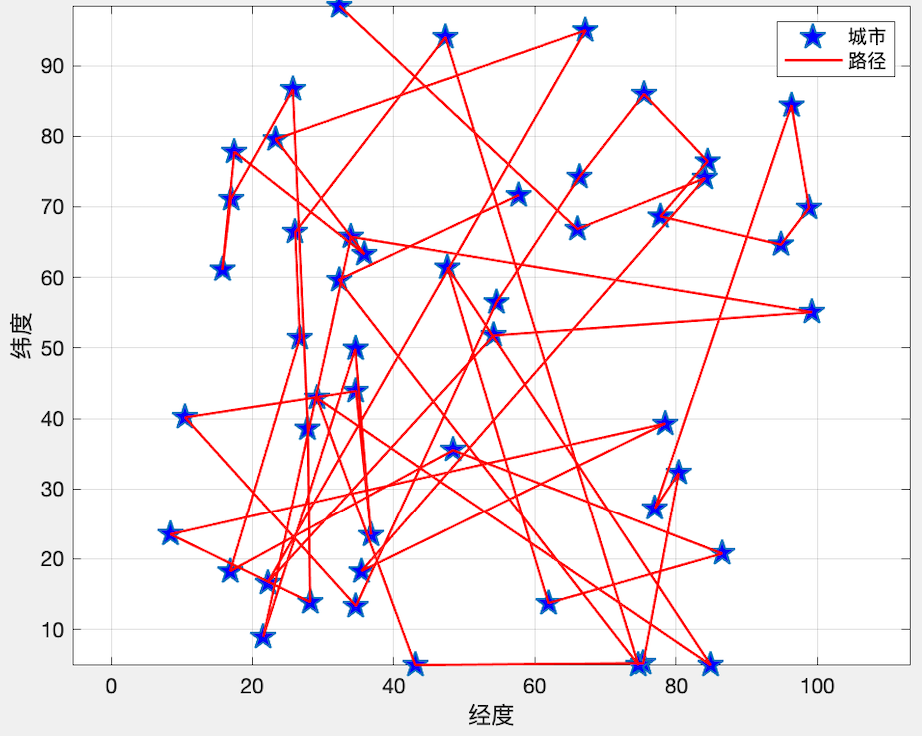
在我们的N维平面是,咱们今天的话是拿这个二维平面来的,在这个平面上有很多个城市,城市之间是彼此联通的,我们现在要找出一条最短的路径可以将全部城市都走完。例如我们有城市A,B,C,D,E。现在知道了城市间的坐标,也就是相当于知道了城市之间的距离,那么现在找到一个顺序,能够使得走完A,B,C,D,E所有城市的路径和最短。比如计算完之后可能是B-->A-->C-->E-->D。换一句话说找到这个顺序。
枚举
首先要解决这个问题的话,其实方案有很多,说变白了,咱们就是要找到一个顺序,能够让路径之和最小,那么最容易想到的自然就是枚举,例如让A先走然后看距离A最近的假设是B,那么就走B,然后从B走。当然这个是局部贪心策略,很容易走到局部最优,那么这个时候我们可以考虑DP,也就是说依然假设从A开始,然后确保2个城市最短,3个城市最短,4个5个。最后在假设从B开始同理。或者直接 枚举全部情况,计算距离。但是无论如何,随着城市数量的上升,他们的复杂度都会增加,所以这个时候我们就要想办法让计算能不能发挥一下咱们人类的专长了。我称之为“瞎蒙”。
智能算法
现在我们来聊聊这个智能算法,以及为什么要用着玩意,刚刚咱们说了前面的方案对于大量的数据计算量会很大,也不见得编写简单。那么这个时候,首先单单对于TSP问题来说,我们要的就是一个序列,一个不会重复的序列。那么这个时候,有什么一个更加简便的方案,而且在数据足够大的情况下,我们也不见得需要一个完全精准,完全最小的解,只要接近就好。那么这个时候,采用传统的那些算法的话,一就是一,他只会按照我们的规则去计算,而且我们也确实不知道标准答案是什么,对传统算法也比较难去设定一个阈值去停止运算。但是对我们人来说,有一种东西叫做“运气”,有的人运气贼好,可能一发入魂,一下子就懵出了答案。那么我们的智能算法其实就是有点类似于“蒙”。但是人家蒙是讲究技巧的,例如经验告诉我们,三长一短选最短,通过这个技巧可以去蒙一下答案,或者找男盆友的时候像博主一样帅气的男孩纸,只要一张40系(30也可以)显卡就可以轻松带走一样。蒙是要技巧的,我们管这个叫做策略。
策略
那么我们刚刚说的这个技巧,这个蒙的技巧。在智能算法里面,这个蒙,就是我们的一种策略。我们要怎么去蒙才能够让我们的解更加合理。那么这个时候,就开始百花齐放了,这里我就不念经了,我们拿最金典的两个算法为例子,一个是遗传算法,一个是粒子群算法(PSO)。为例子,他们就是采用了一种策略去蒙,例如遗传算法,通过模拟物竞天择,一开始先随机蒙出一堆解,一堆序列,然后按照咱们的这个物竞天择的策略出筛选这些解,然后通过这些解再去蒙出新的更好的解。如此往复,之后蒙出不错的解。粒子群也是类似的,这些部分咱们用的时候再详细说明。
算法
现在咱们已经知道了这个策略,那么算法是啥,其实就是实现这些策略的步骤啊,就是咱们的代码,咱们的循环,数据结构。我们要去实现刚刚说的例如物竞天择,例如咱们TSP,如何随机生成一堆解。
数据样例
ok,到这里咱们已经说完了,基本的一些概念,那么这个时候的话,咱们来看看咱们如何表示这个TSP的问题,这个其实很简单,咱们这边的话就简单的准备一个测试数据,我们这里假设有14个城市,那么我们的这些城市的数据如下:
data = np.array([16.47,96.10,16.47,94.44,20.09,92.54,22.39,93.37,25.23,97.24,22.00,96.05,20.47,97.02,17.20,96.29,16.30,97.38,14.05,98.12,16.53,97.38,21.52,95.59,19.41,97.13,20.09,92.55]).reshape((14,2))
我们后面都用这组数据进行测试,现在在上面已经有了14个城市。
那么接下来我们开始我们的解决方案
遗传算法
ok,那么我们来说一说咱们的这个遗传算法是怎么一回事,之后的话,咱们用这个来解决这个TSP问题。
那么现在的话,我们来看看我们的遗传算法是怎么蒙的。
算法流程
遗传算法其实是在用计算机模拟我们的物种进化。其实更加通俗的说法是筛选,这个就和我们袁隆平老爷爷种植水稻一样。有些个体发育良好,有些个体发育不好,那么我就先筛选出发育好的,然后让他们去繁衍后代,然后再筛选,最后得到高产水稻。其实也和我们社会一样,不努力就木有女朋友就不能保留自己的基因,然后剩下的人就是那些优秀的人和富二代的基因,这就是现实呀。所以得好好学习,天天向上!
那么回到主题,我们的遗传算法就是在模拟这一个过程,模拟一个物竞天择的过程。
所以在我们的算法里面也是分为几大块
繁殖
首先我们的种群需要先繁殖。这样才能不断产生优良基于,那么对应我们的算法,假设我们需要求取
Y = np.sin(10 * x) * x + np.cos(2 * x) * x
的最大值(在一个范围内)那么我们的个体就是一组(X1)的解。好的个体就会被保留,不好的就会被pass,选择标准就是我们的函数 Y 。那么问题来了如何模拟这个过程?我们都知道在繁殖后代的时候我们是通过DNA来保留我们的基因信息,在这个过程当中,父母的DNA交互,并且在这个过程当中会产生变异,这样一来,父母双方的优秀基于会被保存,并且产生的变异有可能诞生更加优秀的后代。
所以接下来我们需要模拟我们的DNA,进行交叉和变异。
交叉
这个交叉过程和我们的生物其实很像,当然我们在我们的计算机里面对于数字我们可以将其转化为二进制,当做我们的DNA
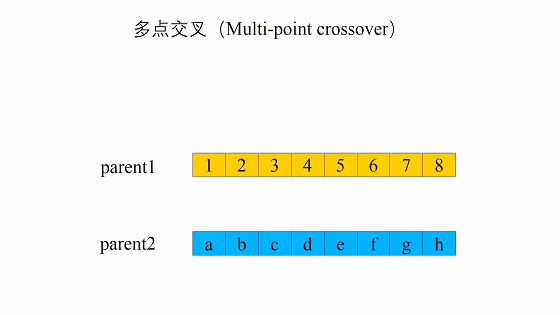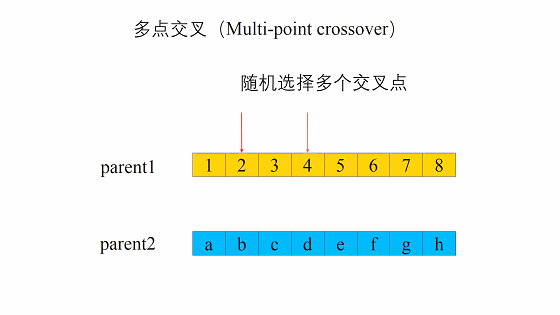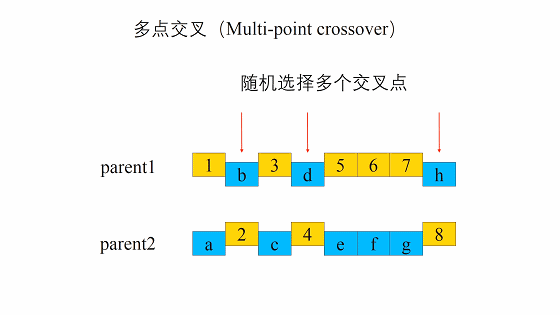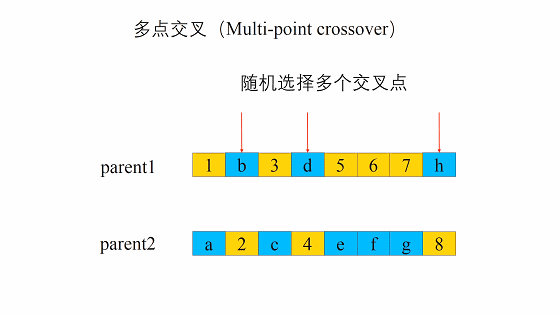
交叉的方式有很多,我们这边选择这一个,进行交叉。
变异
那这个在我们这里就更加简单了
我们只需要在交叉之后,再随机选择几个位置进行改变值就可以了。当然变异的概率是很小的,并且是随机的,这一点要注意。并且由于变异是随机的,所以不排除生成比原来还更加糟糕的个体。
选择
最后我们按照一定的规则去筛选这个些个体就可以了,然后淘汰原来的个体。那么在我们的计算机里面是使用了两个东西,首先我们要把原来二进制的玩意,给转化为我们原来的十进制然后带入我们的函数运算,然后保存起来,之后再每一轮统一筛选一下就好了。
逆转
这个咋说呢,说好听点叫逆转,难听点就算,对于一些新的生成的不好的解,我们是要舍弃的。
代码
那么这部分用代码描述的话就是这样的:
import numpy as np
import matplotlib.pyplot as plt
Population_Size =100
Iteration_Number =200
Cross_Rate =0.8
Mutation_Rate =0.003
Dna_Size =10
X_Range=[0,5]defF(x):'''
目标函数,需要被优化的函数
:param x:
:return:
'''return np.sin(10* x)* x + np.cos(2* x)* x
defCrossOver(Parent,PopSpace):'''
交叉DNA,我们直接在种群里面选择一个交配
然后就生出孩子了
:param parent:
:param PopSpace:
:return:
'''if(np.random.rand())< Cross_Rate:
cross_place = np.random.randint(0,2, size=Dna_Size).astype(np.bool)
cross_one = np.random.randint(0, Population_Size, size=1)#选择一位男/女士交配
Parent[cross_place]= PopSpace[cross_one,cross_place]return Parent
defMutate(Child):'''
变异
:param Child:
:return:
'''for point inrange(Dna_Size):if np.random.rand()< Mutation_Rate:
Child[point]=1if Child[point]==0else0return Child
defTranslateDNA(PopSpace):'''
把二进制转化为十进制方便计算
:param PopSpace:
:return:
'''return PopSpace.dot(2** np.arange(Dna_Size)[::-1])/float(2** Dna_Size -1)* X_Range[1]defFitness(pred):'''
这个其实是对我们得到的F(x)进行换算,其实就是选择的时候
的概率,我们需要处理负数,因为概率不能为负数呀
pred 这是一个二维矩阵
:param pred:
:return:
'''return pred +1e-3- np.min(pred)defSelect(PopSpace,Fitness):'''
选择
:param PopSpace:
:param Fitness:
:return:
''''''
这里注意的是,我们先按照权重去选择我们的优良个体,所以我们这里选择的时候允许重复的元素出现
之后我们就可以去掉这些重复的元素,这样才能实现保留良种去除劣种。100--》70(假设有30个重复)
如果不允许重复的话,那你相当于没有筛选
'''
Better_Ones = np.random.choice(np.arange(Population_Size), size=Population_Size, replace=True,
p=Fitness / Fitness.sum())# np.unique(Better_Ones) #这个是我后面加的return PopSpace[Better_Ones]if __name__ =='__main__':
PopSpace = np.random.randint(2, size=(Population_Size, Dna_Size))# initialize the PopSpace DNA
plt.ion()
x = np.linspace(X_Range,200)# plt.plot(x, F(x))
plt.xticks([0,10])
plt.yticks([0,10])for _ inrange(Iteration_Number):
F_values = F(TranslateDNA(PopSpace))# something about plottingif'sca'inglobals():
sca.remove()
sca = plt.scatter(TranslateDNA(PopSpace), F_values, s=200, lw=0, c='red', alpha=0.5)
plt.pause(0.05)# GA part (evolution)
fitness = Fitness(F_values)print("Most fitted DNA: ", PopSpace[np.argmax(fitness)])
PopSpace = Select(PopSpace, fitness)
PopSpace_copy = PopSpace.copy()for parent in PopSpace:
child = CrossOver(parent, PopSpace_copy)
child = Mutate(child)
parent[:]= child
plt.ioff()
plt.show()
这个代码是以前写的,逆转没有写上(下面的有)
TSP遗传算法
ok,刚刚的例子是拿的解方程,也就是说是一个连续问题吧,当然那个连续处理的话并不是很好,只是一个演示。那么我们这个的话其实类似的。首先我们的DNA,是城市的路径,也就是A-B-C-D等等,当然我们用下标表示城市。
种群表示
首先我们确定了使用城市的序号作为我们的个体DNA,例如咱们种群大小为100,有ABCD四个城市,那么他就是这样的,我们先随机生成种群,长这个样:
123423453214...
那个1,2,3,4是ABCD的序号。
交叉,变异
这里面的话,值得一提的就是,由于暂定城市需要是不能重复的,且必须是完整的,所以如果像刚刚那样进行交叉或者变异的话,那么实际上会出点问题,我们不允许出现重复,且必须完整,对于我们的DNA,也就是咱们瞎蒙的个体。
代码
由于咱们每一步在代码里面都有注释,所以的话咱们在这里就不再进行复述了。
from math import floor
import numpy as np
import matplotlib.pyplot as plt
classGena_TSP(object):"""
使用遗传算法解决TSP问题
"""def__init__(self, data, maxgen=200,
size_pop=200, cross_prob=0.9,
pmuta_prob=0.01, select_prob=0.8):
self.maxgen = maxgen # 最大迭代次数
self.size_pop = size_pop # 群体个数,(一次性瞎蒙多少个解)
self.cross_prob = cross_prob # 交叉概率
self.pmuta_prob = pmuta_prob # 变异概率
self.select_prob = select_prob # 选择概率
self.data = data # 城市的坐标数据
self.num =len(data)# 有多少个城市,对应多少个坐标,对应染色体的长度(我们的解叫做染色体)"""
计算城市的距离,我们用矩阵表示城市间的距离
"""
self.__matrix_distance = self.__matrix_dis()
self.select_num =int(self.size_pop * self.select_prob)# 通过选择概率确定子代的选择个数"""
初始化子代和父代种群,两者相互交替
"""
self.parent = np.array([0]* self.size_pop * self.num).reshape(self.size_pop, self.num)
self.child = np.array([0]* self.select_num * self.num).reshape(self.select_num, self.num)"""
负责计算每一个个体的(瞎蒙的解)最后需要多少距离
"""
self.fitness = np.zeros(self.size_pop)
self.best_fit =[]
self.best_path =[]# 保存每一步的群体的最优路径和距离def__matrix_dis(self):"""
计算14个城市的距离,将这些距离用矩阵存起来
:return:
"""
res = np.zeros((self.num, self.num))for i inrange(self.num):for j inrange(i +1, self.num):
res[i, j]= np.linalg.norm(self.data[i,:]- self.data[j,:])
res[j, i]= res[i, j]return res
defrand_parent(self):"""
初始化种群
:return:
"""
rand_ch = np.array(range(self.num))for i inrange(self.size_pop):
np.random.shuffle(rand_ch)
self.parent[i,:]= rand_ch
self.fitness[i]= self.comp_fit(rand_ch)defcomp_fit(self, one_path):"""
计算,咱们这个路径的长度,例如A-B-C-D
:param one_path:
:return:
"""
res =0for i inrange(self.num -1):
res += self.__matrix_distance[one_path[i], one_path[i +1]]
res += self.__matrix_distance[one_path[-1], one_path[0]]return res
defout_path(self, one_path):"""
输出我们的路径顺序
:param one_path:
:return:
"""
res =str(one_path[0]+1)+'-->'for i inrange(1, self.num):
res +=str(one_path[i]+1)+'-->'
res +=str(one_path[0]+1)+'\n'print(res)defSelect(self):"""
通过我们的这个计算的距离来计算出概率,也就是当前这些个体DNA也就瞎蒙的解
之后我们在通过概率去选择个体,放到child里面
:return:
"""
fit =1./(self.fitness)# 适应度函数
cumsum_fit = np.cumsum(fit)
pick = cumsum_fit[-1]/ self.select_num *(np.random.rand()+ np.array(range(self.select_num)))
i, j =0,0
index =[]while i < self.size_pop and j < self.select_num:if cumsum_fit[i]>= pick[j]:
index.append(i)
j +=1else:
i +=1
self.child = self.parent[index,:]defCross(self):"""
模仿DNA交叉嘛,就是交换两个瞎蒙的解的部分的解例如
A-B-C-D
C-D-A-B
我们选几个交叉例如这样
A-D-C-B
1,3号交换了位置,当然这里注意可不能重复啊
:return:
"""if self.select_num %2==0:
num =range(0, self.select_num,2)else:
num =range(0, self.select_num -1,2)for i in num:if self.cross_prob >= np.random.rand():
self.child[i,:], self.child[i +1,:]= self.intercross(self.child[i,:],
self.child[i +1,:])defintercross(self, ind_a, ind_b):"""
这个是我们两两交叉的具体实现
:param ind_a:
:param ind_b:
:return:
"""
r1 = np.random.randint(self.num)
r2 = np.random.randint(self.num)while r2 == r1:
r2 = np.random.randint(self.num)
left, right =min(r1, r2),max(r1, r2)
ind_a1 = ind_a.copy()
ind_b1 = ind_b.copy()for i inrange(left, right +1):
ind_a2 = ind_a.copy()
ind_b2 = ind_b.copy()
ind_a[i]= ind_b1[i]
ind_b[i]= ind_a1[i]
x = np.argwhere(ind_a == ind_a[i])
y = np.argwhere(ind_b == ind_b[i])iflen(x)==2:
ind_a[x[x != i]]= ind_a2[i]iflen(y)==2:
ind_b[y[y != i]]= ind_b2[i]return ind_a, ind_b
defMutation(self):"""
之后是变异模块,这个就是按照某个概率,去替换瞎蒙的解里面的其中几个元素。
:return:
"""for i inrange(self.select_num):if np.random.rand()<= self.cross_prob:
r1 = np.random.randint(self.num)
r2 = np.random.randint(self.num)while r2 == r1:
r2 = np.random.randint(self.num)
self.child[i,[r1, r2]]= self.child[i,[r2, r1]]defReverse(self):"""
近化逆转,就是说下一次瞎蒙的解如果没有更好的话就不进入下一代,同时也是随机选择一个部分的
我们不是一次性全部替换
:return:
"""for i inrange(self.select_num):
r1 = np.random.randint(self.num)
r2 = np.random.randint(self.num)while r2 == r1:
r2 = np.random.randint(self.num)
left, right =min(r1, r2),max(r1, r2)
sel = self.child[i,:].copy()
sel[left:right +1]= self.child[i, left:right +1][::-1]if self.comp_fit(sel)< self.comp_fit(self.child[i,:]):
self.child[i,:]= sel
defBorn(self):"""
替换,子代变成新的父代
:return:
"""
index = np.argsort(self.fitness)[::-1]
self.parent[index[:self.select_num],:]= self.child
defmain(data):
Path_short = Gena_TSP(data)# 根据位置坐标,生成一个遗传算法类
Path_short.rand_parent()# 初始化父类## 绘制初始化的路径图
fig, ax = plt.subplots()
x = data[:,0]
y = data[:,1]
ax.scatter(x, y, linewidths=0.1)for i, txt inenumerate(range(1,len(data)+1)):
ax.annotate(txt,(x[i], y[i]))
res0 = Path_short.parent[0]
x0 = x[res0]
y0 = y[res0]for i inrange(len(data)-1):
plt.quiver(x0[i], y0[i], x0[i +1]- x0[i], y0[i +1]- y0[i], color='r', width=0.005, angles='xy', scale=1,
scale_units='xy')
plt.quiver(x0[-1], y0[-1], x0[0]- x0[-1], y0[0]- y0[-1], color='r', width=0.005, angles='xy', scale=1,
scale_units='xy')
plt.show()print('初始染色体的路程: '+str(Path_short.fitness[0]))# 循环迭代遗传过程for i inrange(Path_short.maxgen):
Path_short.Select()# 选择子代
Path_short.Cross()# 交叉
Path_short.Mutation()# 变异
Path_short.Reverse()# 进化逆转
Path_short.Born()# 子代插入# 重新计算新群体的距离值for j inrange(Path_short.size_pop):
Path_short.fitness[j]= Path_short.comp_fit(Path_short.parent[j,:])
index = Path_short.fitness.argmin()if(i +1)%50==0:print('第'+str(i +1)+'步后的最短的路程: '+str(Path_short.fitness[index]))print('第'+str(i +1)+'步后的最优路径:')
Path_short.out_path(Path_short.parent[index,:])# 显示每一步的最优路径# 存储每一步的最优路径及距离
Path_short.best_fit.append(Path_short.fitness[index])
Path_short.best_path.append(Path_short.parent[index,:])return Path_short # 返回遗传算法结果类if __name__ =='__main__':
data = np.array([16.47,96.10,16.47,94.44,20.09,92.54,22.39,93.37,25.23,97.24,22.00,96.05,20.47,97.02,17.20,96.29,16.30,97.38,14.05,98.12,16.53,97.38,21.52,95.59,19.41,97.13,20.09,92.55]).reshape((14,2))
main(data)
运行结果
ok,我们来看看运行的结果: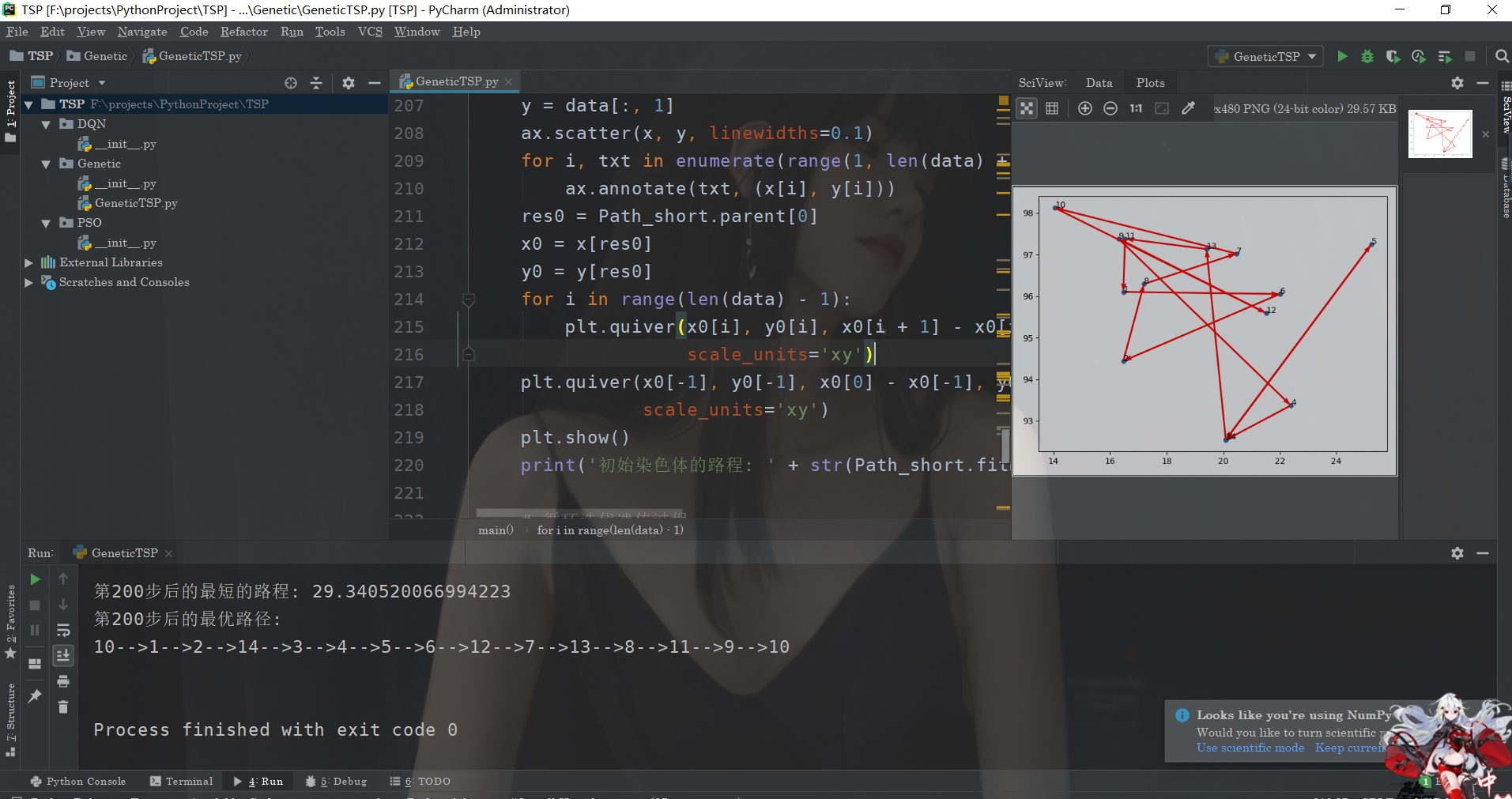
总结
这个是咱们最简单的遗传算法版本,其实后面的这个PSO版本也简单,也要用到遗传的部分。
那么DQN的部分其实也简单,当然难一点的是并行编程(懂原理的情况下)。那么这个咱们依次来点兵点将。ok,今天水了两篇文章了超常发挥了哈,没事很快我又会成为周更作者的(狗头)。
版权归原作者 Huterox 所有, 如有侵权,请联系我们删除。