1 准备阶段
部署好相关的软件和工具
BWA (Burrow-Wheeler Aligner) Version 0.7.17-r1188 解压、编译
Samtools Version: 1.16.1
解压 tar jxvf samtools-1.16.1.tar.gz
进入目录 cd samtools-1.16.1
配置 ./configure --prefix=~/biosoft/samtools-1.16.1
编译安装 make
make install
Picard 直接下载java包picard.jar
GATK gatk-4.3.0.0 下载后不用编译直接使用
上述软件均需添加到环境变量
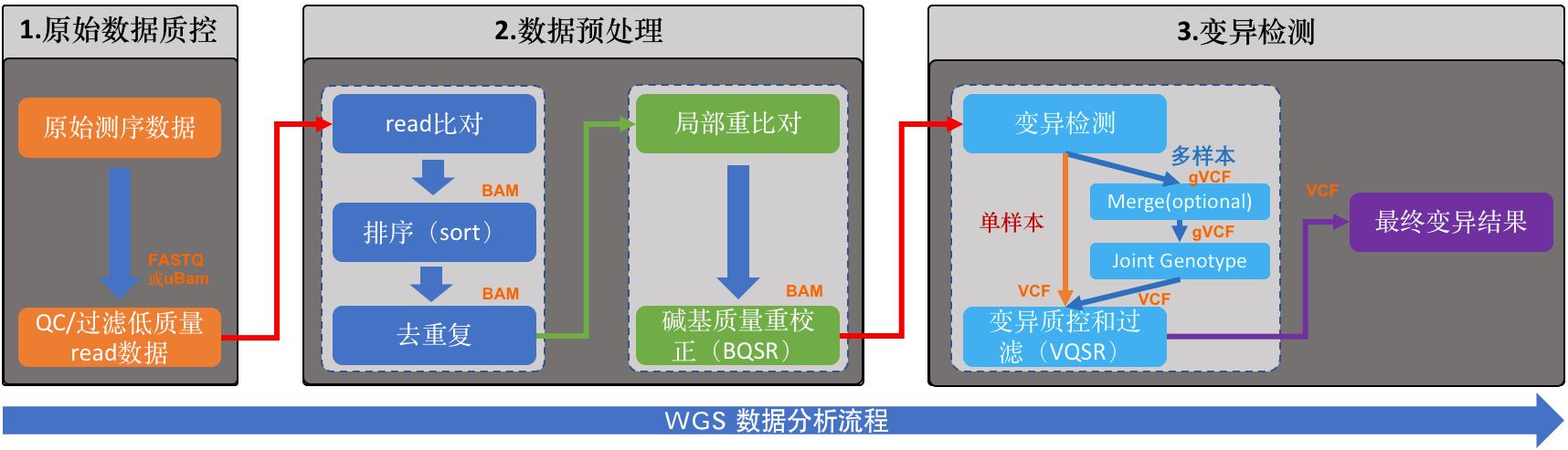
2 数据预处理
2.1 构建索引
参考基因组索引的构建
bwa index Sus_scrofa.Sscrofa11.1.dna.toplevel.fa
java -jar /home/dengsx/biosoft/picard.jar CreateSequenceDictionary \
R=/home/dengsx/publicdata/reference_genome/Sus_scrofa.Sscrofa11.1.dna.toplevel.fa \
O=/home/dengsx/publicdata/reference_genome/Sus_scrofa.Sscrofa11.1.dna.toplevel.fa.dict
samtools faidx Sus_scrofa.Sscrofa11.1.dna.toplevel.fa ##fai索引的构建
后续GATK的使用需要用到多种索引类型,要用多个软件创建
dict索引文件也可以通过gatk来获取
gatk CreateSequenceDictionary -R E.coli_K12_MG1655.fa -O E.coli_K12_MG1655.dict && echo "** dict done **"(本人没试过)
###需要注意的是.dict文件的名字前缀需要和fasta的一样,并跟它在同一个路径下,这样GATK才能够找到。
dbSNP索引的构建(2.6 BQSR之前做好就行)
java -jar /home/dengsx/biosoft/gatk-4.3.0.0/gatk-package-4.3.0.0-local.jar IndexFeatureFile --input /home/dengsx/publicdata/dbsnp/sus_scrofa.nospace.vcf > /home/dengsx/publicdata/dbsnp/sus_scrofa.nospace.vcf.index.log 2>&1
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-hErFUiEQ-1666255841603)(Snipaste_2022-10-19_21-52-02.png)]
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-nuH0BtQJ-1666255841604)(Snipaste_2022-10-19_21-53-48.png)]
这一步完成之后,我们就可以将read比对至参考基因组了
2.2 bwa比对
将比对的输出结果直接重定向到一份*.sam文件中,这类文件是BWA比对的标准输出文件,。但SAM文件是文本文件,一般整个文件都非常巨大,因此,为了有效节省磁盘空间,用samtools将它转化为BAM文件(SAM的特殊二进制格式),而且BAM会更加方便于后续的分析。
bwa mem -t 5 -R '@RG\tID:YF62_E7\tPL:UNKNOWN\tLB:library2\tSM:YF62_E7' /home/dengsx/publicdata/reference_genome/Sus_scrofa.Sscrofa11.1.dna.toplevel.fa YF62_E7_1_clean.fq.gz YF62_E7_2_clean.fq.gz | samtools view -S -b - > YF62_E7.bam
-t,线程数,我们在这里使用4个线程;-R 接的是Read Group的字符串信息,这是一个非常重要的信息,以@RG开头,它是用来将比对的read进行分组的。不同的组之间测序过程被认为是相互独立的,这个信息对于我们后续对比对数据进行错误率分析和Mark duplicate时非常重要。
在Read Group中,有如下几个信息非常重要:
- ID,这是Read Group的分组ID,一般设置为测序的lane ID(不同lane之间的测序过程认为是独立的),下机数据中我们都能看到这个信息的,一般都是包含在fastq的文件名中;
- SM,sample_name 同一个个体的sampleID必须一样,有时候我们测序的数据比较多的时候,那么可能会分成多个不同的lane分布测出来,这个时候SM名字就是可以用于区分这些样本;
- PL,指的是所用的测序平台,这个信息不要随便写!在GATK中,PL只允许被设置为:ILLUMINA,SLX,SOLEXA,SOLID,454,LS454,COMPLETE,PACBIO,IONTORRENT,CAPILLARY,HELICOS或UNKNOWN这几个信息,如果实在不知道,那么必须设置为UNKNOWN;
- LB,测序文库的名字,这个重要性稍微低一些,主要也是为了协助区分不同的group而存在。文库名字一般可以在下机的fq文件名中找到,如果上面的lane ID足够用于区分的话,也可以不用设置LB。 以上,我们就完成了read比对的步骤。接下来是排序:
2.3 merge个体
最好是在这一步就merge,后面的操作都是针对merge后的文件
samtools merge -o srr.bam srr1.bam srr2.bam
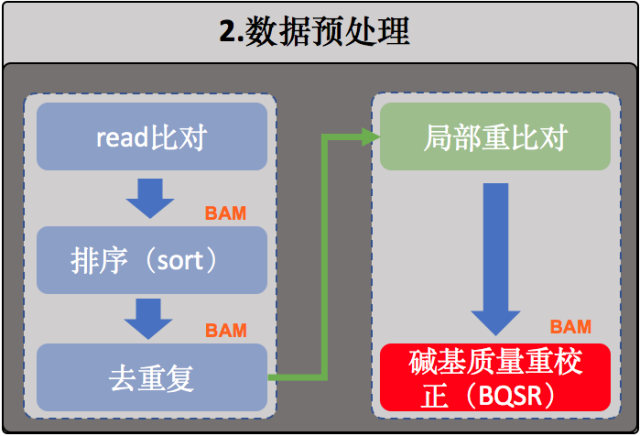
2.4 排序
samtools sort -@ 1 -m 64G -O bam -o A65.sorted.bam A65.bam
其中,-@,用于设定排序时的线程数;-m,限制排序时最大的内存消耗,-O 指定输出为bam格式;-o 是输出文件的名字。建议在做类似分析的时候在文件名字将所做的关键操作包含进去,因为这样即使过了很长时间,当你再去看这个文件的时候也能够立刻知道当时对它做了什么。
2.5 去除重复序列(或者标记重复序列)
for i in `ls *.sorted.bam`
do
java -jar /home/dengsx/biosoft/picard.jar MarkDuplicates \
REMOVE_DUPLICATES=false \
I=${input}/${i} \
O=${output}/${i%\.bam}.markup.bam \
M=${output}/${i%\.bam}.markup_metrics.txt
- 这里只把重复序列在输出的新结果中标记出来,但不删除。如果我们非要把这些序列完全删除的话可以设置参数
REMOVE_DUPLICATES=true建议使用第一种做法,只是标记出来,并留存这些序列,以便在你需要的时候还可以对其做分析。 - 这一步完成之后,我们需要为sample_name.sorted.markdup.bam创建索引文件,它的作用能够让我们可以随机访问这个文件中的任意位置,而且后面的“局部重比对”步骤也要求这个BAM文件一定要有索引,命令如下:
cd /home/dengsx/publicdata/markup_out
for markup_bam in `ls *.sorted.markup.bam`
do
samtools index ${markup_bam}
done
在重新校正碱基质量值(BQSR)之前把相同个体的bam文件merge,将同个样本的所有比对结果合并成唯一一个大的BAM文件
samtools merge -o srr.sorted.markup.bam srr1.sorted.markup.bam srr2.sorted.markup.bam
2.6 重新校正碱基质量值(BQSR)
主要是通过机器学习的方法构建测序碱基的错误率模型,然后对这些碱基的质量值进行相应的调整。
这里包含了两个步骤:
- 第一步,BaseRecalibrator,这里计算出了所有需要进行重校正的read和特征值,然后把这些信息输出为一份校准表文件(sample_name.recal_data.table)
- 第二步,ApplyBQSR ,这一步利用第一步得到的校准表文件(sample_name.recal_data.table)重新调整原来BAM文件中的碱基质量值,并使用这个新的质量值重新输出一份新的BAM文件。
数据准备
dbSNP
数据处理
- 解压
sus_scrofa.vcf.gz文件 - the dbsnp vcf file contains axiom snp array snps that offends the gatk because they have white space in the INFO field, remove these (去除空白格)
awk -F "\t" '!($8 ~ /\s/)' /home/dengsx/publicdata/dbsnp/sus_scrofa.vcf > /home/dengsx/publicdata/dbsnp/sus_scrofa.nospace.vcf
- 构建.idx索引
java -jar /home/dengsx/biosoft/gatk-4.3.0.0/gatk-package-4.3.0.0-local.jar IndexFeatureFile --input /home/dengsx/publicdata/dbsnp/sus_scrofa.nospace.vcf > /home/dengsx/publicdata/dbsnp/sus_scrofa.nospace.vcf.index.log 2>&1
BQRS重新校正碱基质量值
BQRS 第一步(BaseRecalibrator)
fasta=/home/dengsx/publicdata/reference_genome/Sus_scrofa.Sscrofa11.1.dna.toplevel.fa
input=/home/dengsx/publicdata/markup_out/
gatk --java-options "-Xmx10G -Djava.io.tmpdir=./" BaseRecalibrator \
-R ${fasta} \
-I ${input}/A65.sorted.markup.bam \
--known-sites /home/dengsx/publicdata/dbsnp/sus_scrofa.nospace.vcf \
-O A65.recal_data.table
- 这里计算出了所有需要进行重校正的read和特征值,然后把这些信息输出为一份校准表文件(sample_name.recal_data.table)
不能只用bwa构建的索引来做BQSR
需要的索引类型很多(参考上面索引的构建)
- 参考基因组的索引
.fai、.dict
java -jar /home/dengsx/biosoft/picard.jar CreateSequenceDictionary R=/home/dengsx/publicdata/reference_genome/Sus_scrofa.Sscrofa11.1.dna.toplevel.fa O=/home/dengsx/publicdata/reference_genome/Sus_scrofa.Sscrofa11.1.dna.toplevel.fa.dict
samtools faidx Sus_scrofa.Sscrofa11.1.dna.toplevel.fa
- sus_scrofa.nospace.vcf的索引
.idx - bam文件的索引
BQRS 第二步(ApplyBQSR)
fasta=/home/dengsx/publicdata/reference_genome/Sus_scrofa.Sscrofa11.1.dna.toplevel.fa
input=/home/dengsx/publicdata/markup_out/
bqsr=/home/dengsx/publicdata/BQSR/
gatk --java-options "-Xmx10G -Djava.io.tmpdir=./" ApplyBQSR \
-R ${fasta} \
-I ${input}/A65.sorted.markup.bam \
--bqsr-recal-file ${bqsr}/A65.recal_data.table \
-O A65.sorted.markdup.BQSR.bam
- 这一步利用第一步得到的校准表文件(sample_name.recal_data.table)重新调整原来BAM文件中的碱基质量值,并使用这个新的质量值重新输出一份新的BAM文件。
3 变异检测
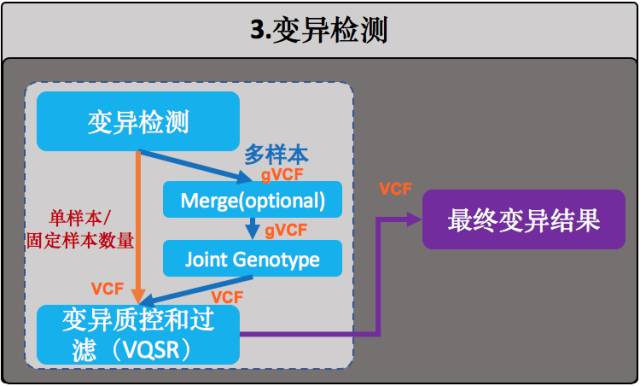
我们这里使用GATK HaplotypeCaller模块对样本中的变异进行检测,它也是目前最适合用于对二倍体基因组进行变异(SNP+Indel)检测的算法。
一般来说,在实际的WGS流程中对HaplotypeCaller的应用有两种做法,差别只在于要不要在中间生成一个gVCF:
- 第一种,直接进行HaplotypeCaller,这适合于单样本,或者那种固定样本数量的情况,也就是执行一次HaplotypeCaller之后就老死不相往来了。否则你会碰到仅仅只是增加一个样本就得重新运行这个HaplotypeCaller的坑爹情况(即,N+1难题),而这个时候算法需要重新去读取所有人的BAM文件,这将会是一个很费时间的痛苦过程;
- 第二种,每个样本先各自生成gVCF,然后再进行群体joint-genotype。这其实就是GATK团队为了解决(1)中的N+1难题而设计出来的模式。gVCF全称是genome VCF,是每个样本用于变异检测的中间文件,格式类似于VCF,它把joint-genotype过程中所需的所有信息都记录在这里面,文件无论是大小还是数据量都远远小于原来的BAM文件。这样一旦新增加样本也不需要再重新去读取所有人的BAM文件了,只需为新样本生成一份gVCF,然后重新执行这个joint-genotype就行了。
基因组上各个不同的染色体之间其实是可以理解为相互独立的(结构性变异除外),也就是说,为了提高效率我们可以按照染色体一条条来独立执行这个步骤,最后再把结果合并起来就好了,这样的话就能够节省很多的时间
input=/home/dengsx/publicdata/BQSR
output=/home/dengsx/publicdata/haplotypeCaller
reference=/home/dengsx/publicdata/reference_genome/Sus_scrofa.Sscrofa11.1.dna.toplevel.fa
cd /home/dengsx/publicdata/haplotypeCaller
ls ../BQSR/*.sorted.markdup.BQSR.bam | while read line
do
gatk --java-options "-Xmx10G -Djava.io.tmpdir=./" HaplotypeCaller \
-I $input/${line##../BQSR/} \
-R $reference \
-ERC GVCF \
-ploidy 2 \
-O $output/${line##../BQSR/}.g.vcf > /home/dengsx/publicdata/haplotypeCaller/haplotypeCaller.log 2>&1
done
wait # this is necessary because both processes need to complete for the outside call to check on logs
echo $(date) done.main.process
参考:碱基矿工
版权归原作者 dengshaoxiong 所有, 如有侵权,请联系我们删除。