小罗碎碎念
最近休息了一段时间没有更新公众号,一方面是找不到写作的手感,还有一方面是想借着这段时间给自己踩一个刹车,不要太浮躁了。
这篇推文算是一个回归之作,想要解决一些大家比较关心的问题:
- 我目前手头的数据能否支撑研究病理AI
- 从事病理AI的研究需要哪些条件
- 完成一个病理AI的项目需要多久的周期
注意,本篇推文提供的预估时间取决于以下几个方面:
- 想要解决的临床问题
- 可提供的样本数量(纯病理or多组学)
- 可提供的计算资源(课题组是否拥有服务器or成本是否支撑租借服务器)
以下内容仅代表小罗个人观点,能力有限,认知有限,若有错误,敬请谅解和批评指正!!
完成一个病理AI的项目需要多久的周期?
对于由临床医生主导的步骤,假设项目想法是从零开始构思的,但数据已经收集完毕。对于由工科主导的步骤,假设已经准备好了服务器,并且操作人员具备基础的背景知识。
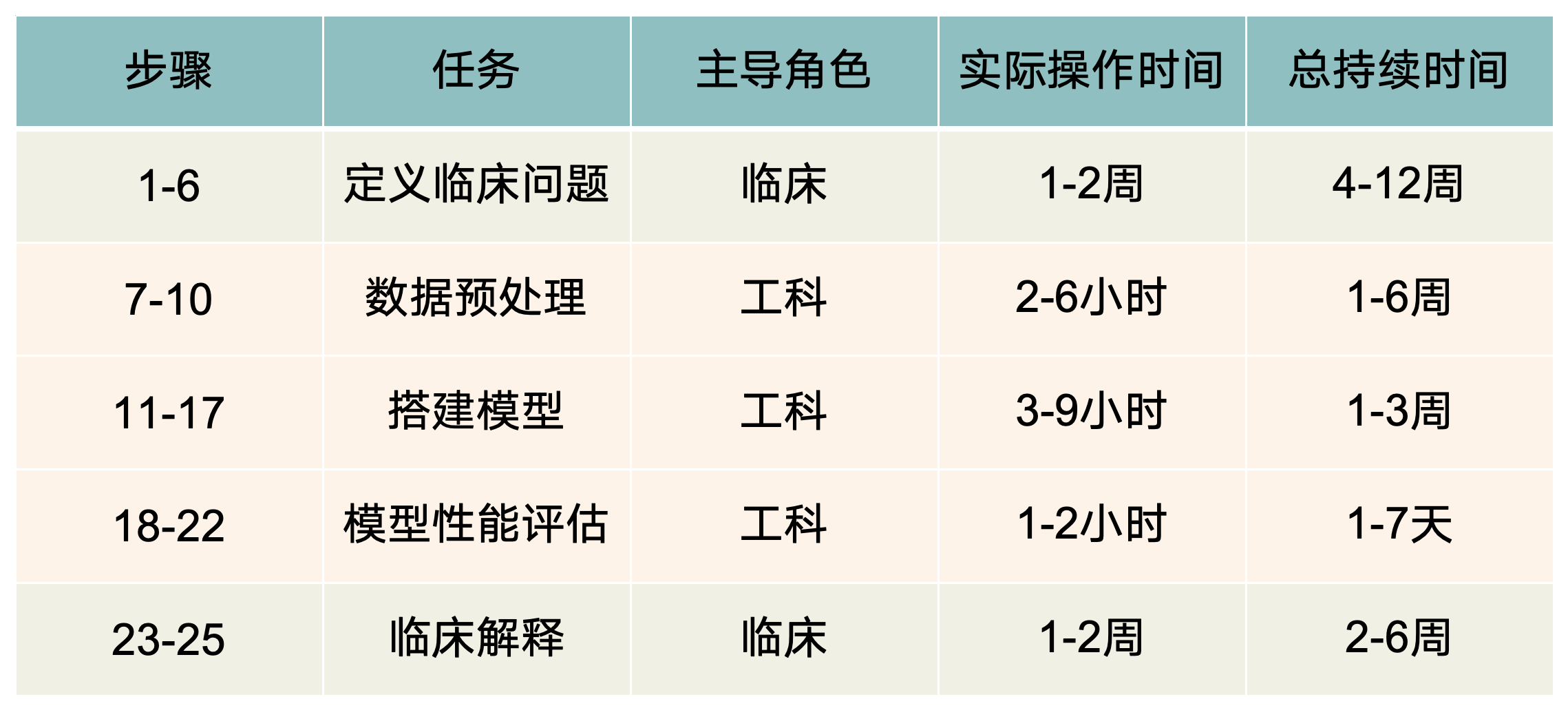
这个表格是我根据数篇顶刊总结并结合自己的实际经验得来的,最短的周期是8周(2个月),最长的周期是28周(7个月)。其实只要严格按照流程走,是可以一边做实验一边写文章的,因为设计好了模型直接运行代码即可,这段时间是可以同步完成后续试验或者用来写论文的。
如果团队有多个成员,可以轮流完成不同的任务流程,这样既可以保证每个人都能掌握所有环节的技能,也能以流水线的方式快速的完成课题。
论文投稿需要多久的周期?
最近这段没有更新推文的时间,我还在做一件事情——总结Nature Medicine发表的所有与病理AI相关的文献。
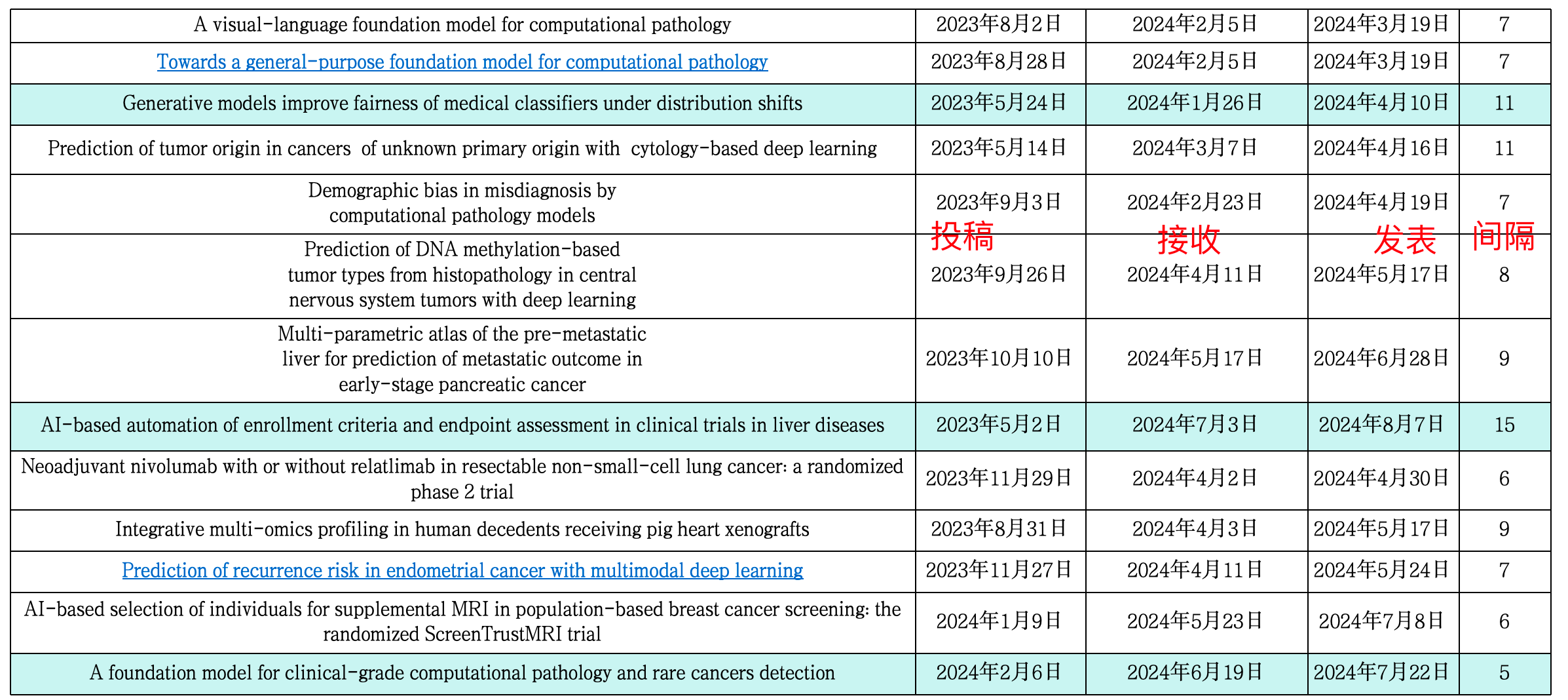
由上面这张表我们可以看出一篇文章从投稿到发表所需的时间往往在半年以上,至于从事这个研究花了多久,我们就不得而知了。
医学AI有一个很明显的特征——收集数据要很久的时间,所以我并没有把收集数据涉及的时间放进去,不然就没有办法去建立标准了。
整个项目周期有多久?
现在我们来思考一个问题——从项目立项开始,到论文发表大概需要多久?
前面提到项目完成最短需要8周,最长需要28周,那么取个中间值18周;论文投稿最快5个月,最慢15个月,那我们就按一年算;假设我们需要两个月写论文和润色,那么总共就是4.5+12+2=18.5 个月。
当然了,第一篇文章是比较慢的,因为没有积累,但是第二篇文章就快了,因为可以在这个基础上迭代,很多步骤不需要重复,这也是为啥上图13篇文章中,有3篇来自同一个课题组。除此以外,人家还顺带发了Nature和Cell。(没错,我说的就是Faisal Mahmood)

重视配置文件的设置
在正式开始介绍后续的内容时,我要额外强调一个内容——config文件的配置。
设置这个文件的初衷是为了尽可能减少用户所需的额外编程或脚本编写——用户可以在配置文件中填写所有路径、变量和设置,这些配置将用于下面步骤中交互的每个环节。
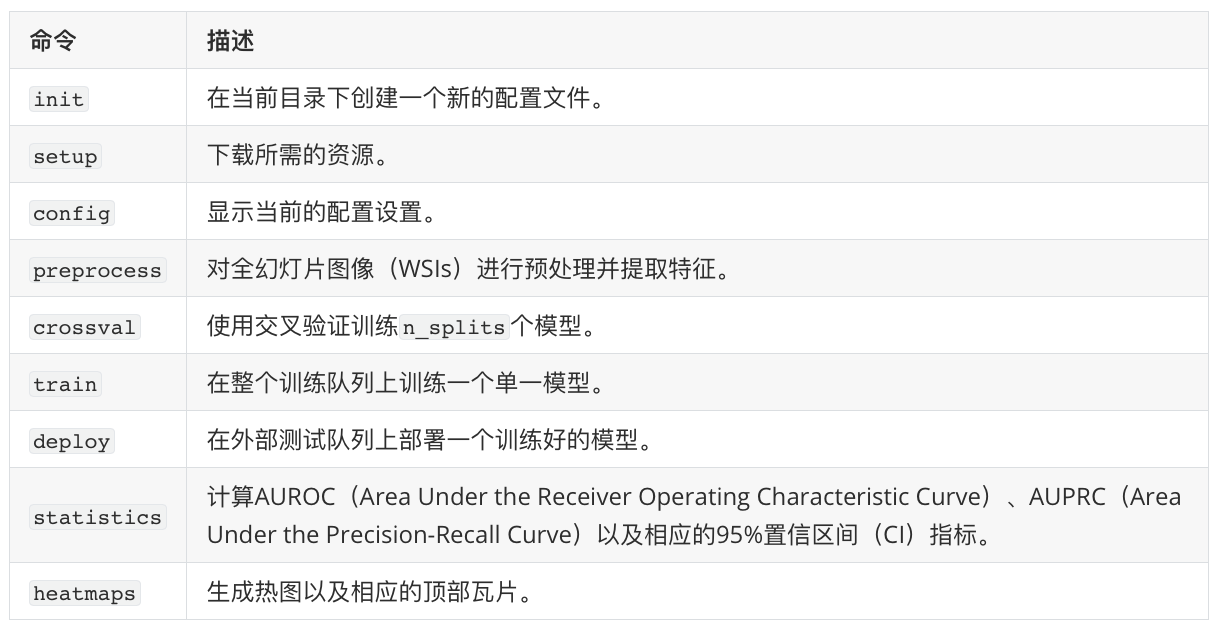
每次修改配置文件后,需保存更改,然后运行 CLI命令。
一、定义临床问题
实际操作1–2周,总计4–12周
1、定义预测目标
这一部分是最关键的,因为这一步直接决定了后续的工作有无意义。
我的推文中,往往开头就会介绍这篇文章搭建了一个什么样的模型,解决了一个什么样的临床问题。

我们可以预测复发风险,也可以预测转移风险,更常见的,我们还可以预测预后,提取新的生物标志物等等。
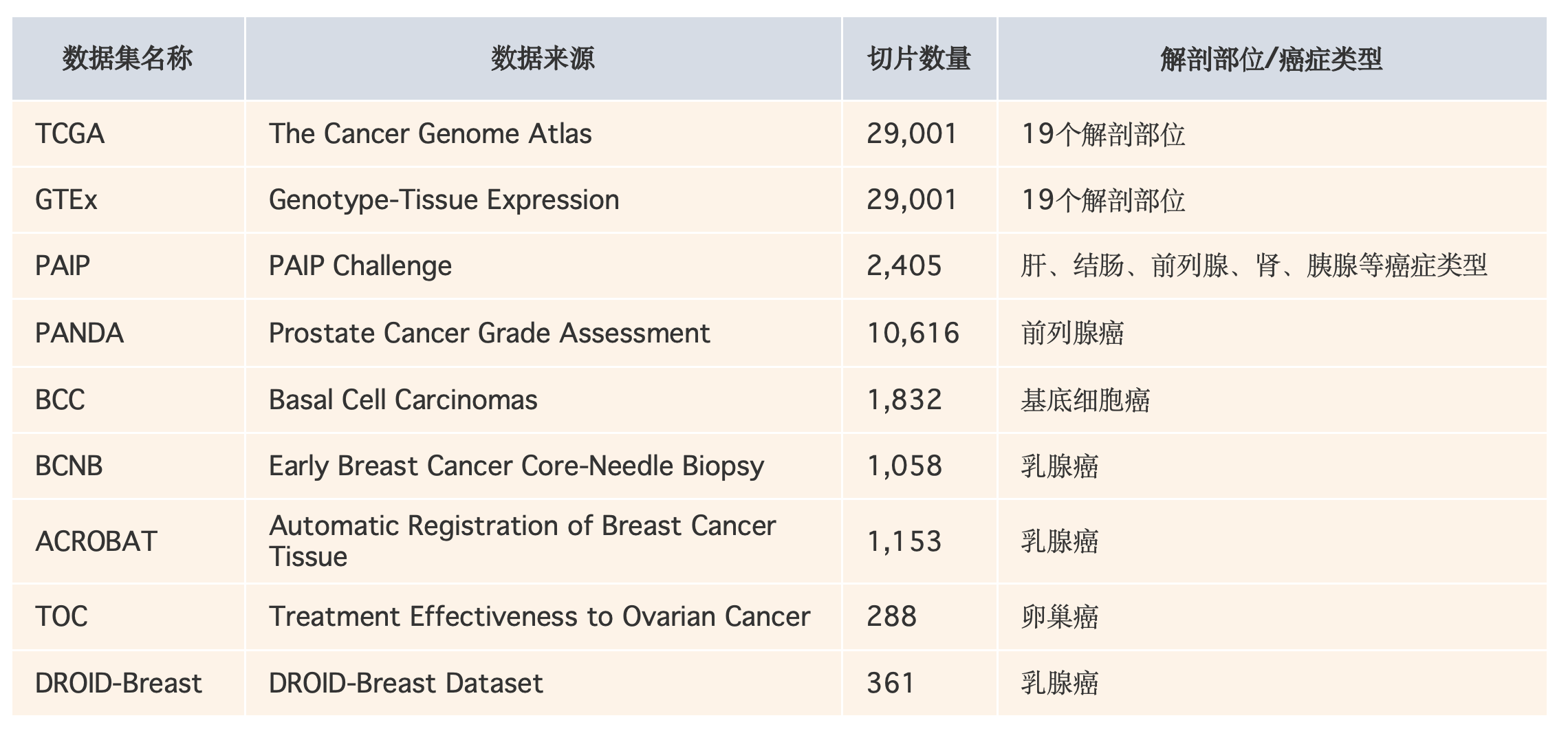
2、评估数据量
还是以HECTOR这篇文章为例,我们一开始就要梳理好这个项目会涉及多少样本。

最好是可以做一个表格,这样一目了然。
3、定义纳入和排除标准
例如下面这篇文章,是关于一项多中心、回顾性的研究——研究了多尺度病理图像纹理特征(Multi-scale pathology image texture signature,简称MPIS)对可切除肺腺癌(Lung adenocarcinoma, LUAD)预后的影响。

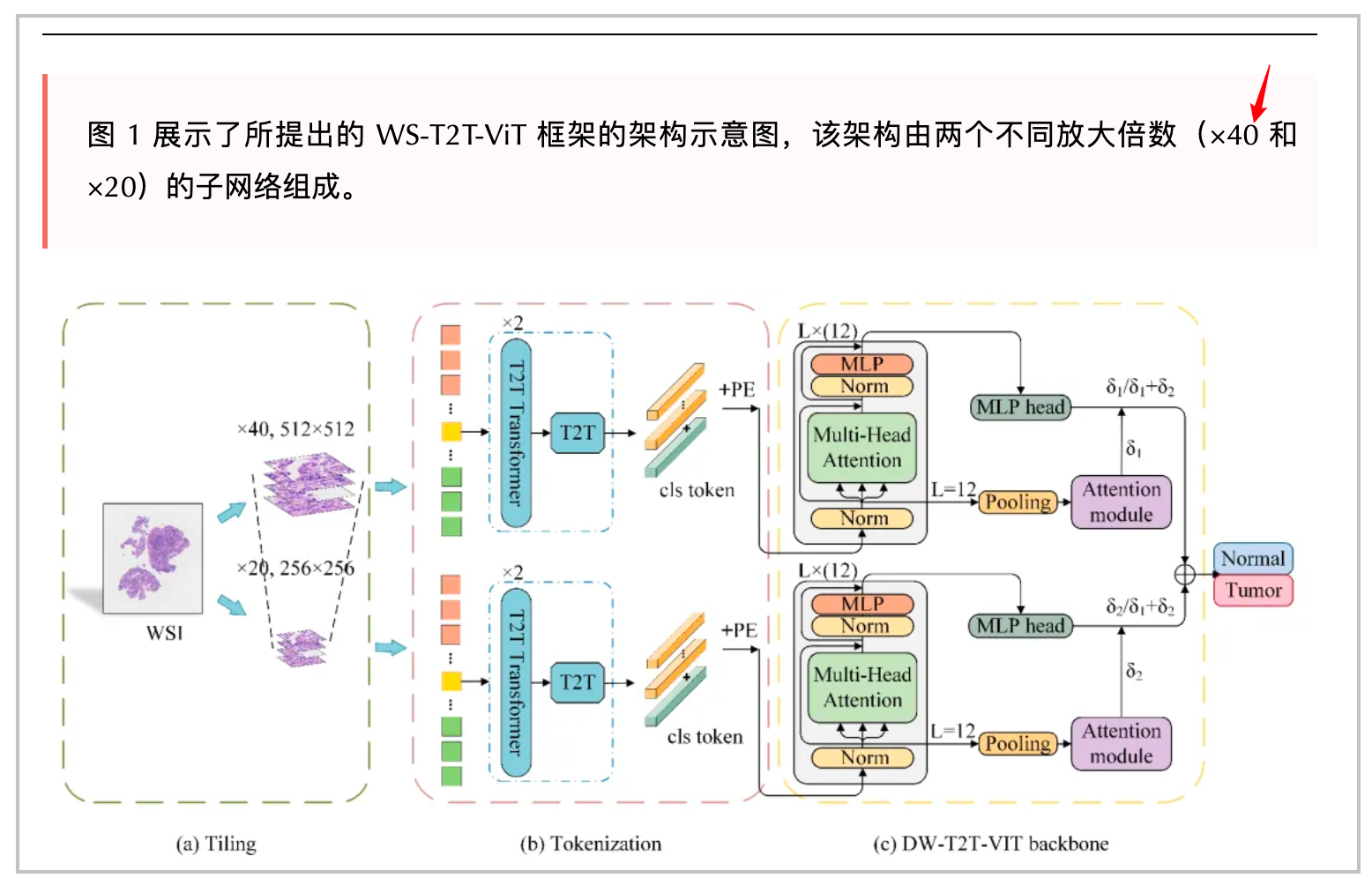
4、确定所需的切片分辨率
常见的分辨率倍数是10×、20×和40×,当然,也有5×的。目前并没有实际的证据表明,过高的分辨率,性能一定会更好。

不过现在更常用的套路是,把所有的分辨率数据都用上,模拟病理学家实际的工作流程。
5、转换数据
这一部分主要涉及的工作是染色标准化,根据目前现已发表的研究来看,默认进行染色标准化是最好的选择。不过随着大量基础模型的发表,这一部分其实已经不需要我们专门去设计了。
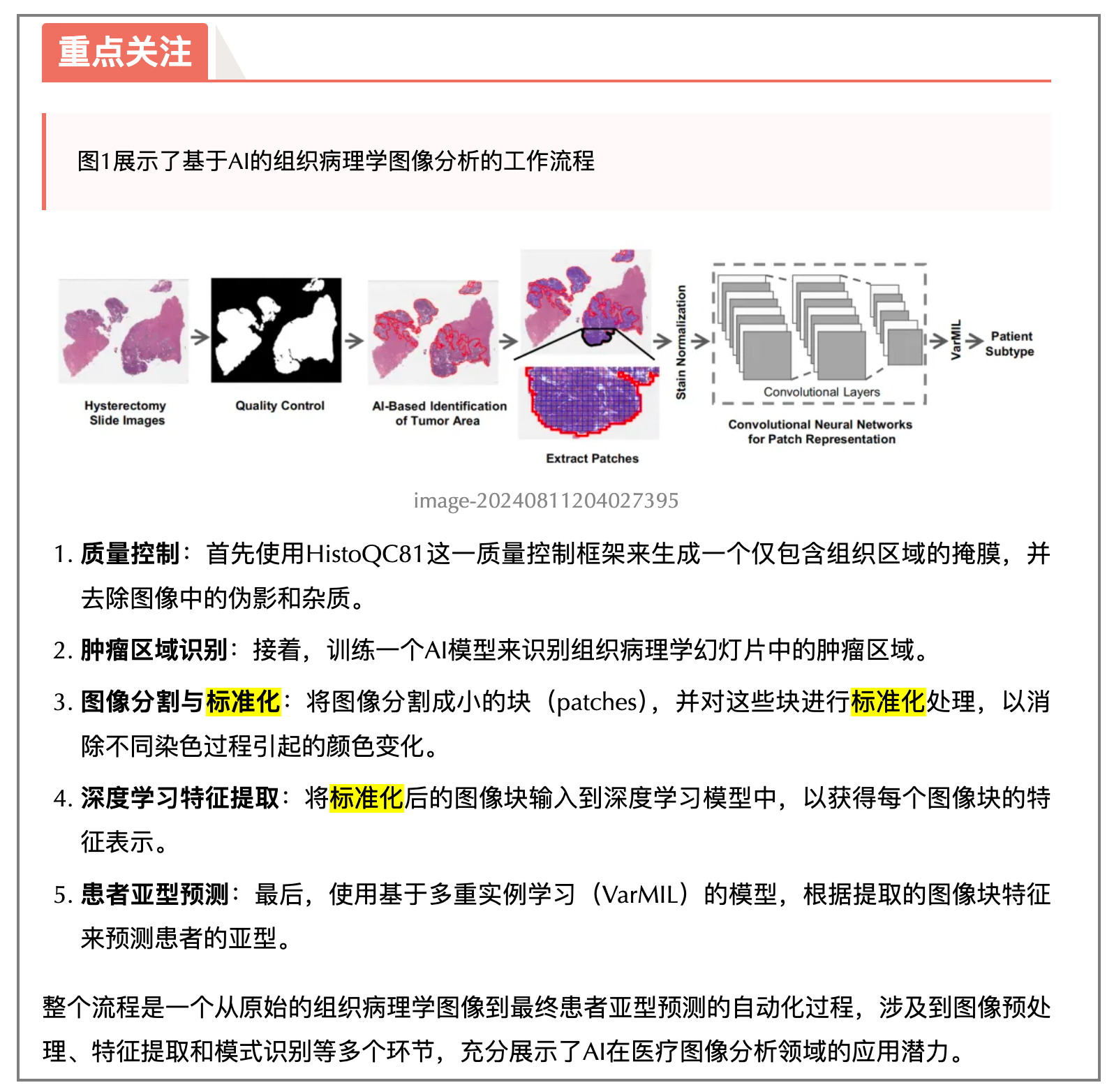
6、定义验证策略
首先,建立患者分配策略以验证步骤1中定义的假设。验证策略需要使用步骤3中定义的纳入和排除标准来确定可用于内部训练队列和外部测试队列的患者数量。
其次,定义允许测试假设的统计测试和指标。建立明确的指标标准和值范围,以及相应的统计测试,以接受或拒绝假设。

二、数据预处理
时间安排:实际操作2–6小时,总计1–6周
7、设置计算资源
这一部分与步骤4相关,我们需要选择与自己硬件条件匹配的数据参数——WSI的分辨率。
例如下图描述的这篇文章,是关于使用深度学习技术来分析组织学中的
三级淋巴结构
(Tertiary Lymphoid Structures, TLS),并预测癌症预后和免疫疗法反应的研究。
作者在描述数据集和模型的开发时做了以下陈述,表明了训练集使用的切片分辨率以及分割patch时使用的参数和patch的数量。

8、设置预处理配置
打开配置文件,并在预处理部分插入所需的参数。
参数示例输入描述
output_dir
/home/storage/output_features
保存结果特征向量的路径。
wsi_dir
/home/storage/wsi_directory
存储全切片图像(WSIs)的路径。
cache_dir
/home/storage/cache_directory
保存中间预处理产品的目录。
microns
256
瓦片的默认边缘长度,以微米为单位。用于MPP比率以确定切片的分辨率。默认的像素边缘长度为224,因为这是所用特征提取器的预期输入大小。因此,默认的MPP为256/224 ≈ 1.14,对应于约9×放大倍数。
norm
false
是否执行染色标准化。
feat_extractor
ctp
选择特征提取模型。
del_slide
false
预处理后是否删除WSIs。
cache
true
是否保存中间图像(切片、背景拒绝、标准化)。
only_feature_extraction
false
是否仅执行特征提取。仅当存在先前预处理的中间产品且被指定为仅特征提取的WSI目录时有效。
cores
8
使用的CPU核心数量。
device
cuda:0
选择用于预处理的设备,可以是GPU或CPU。
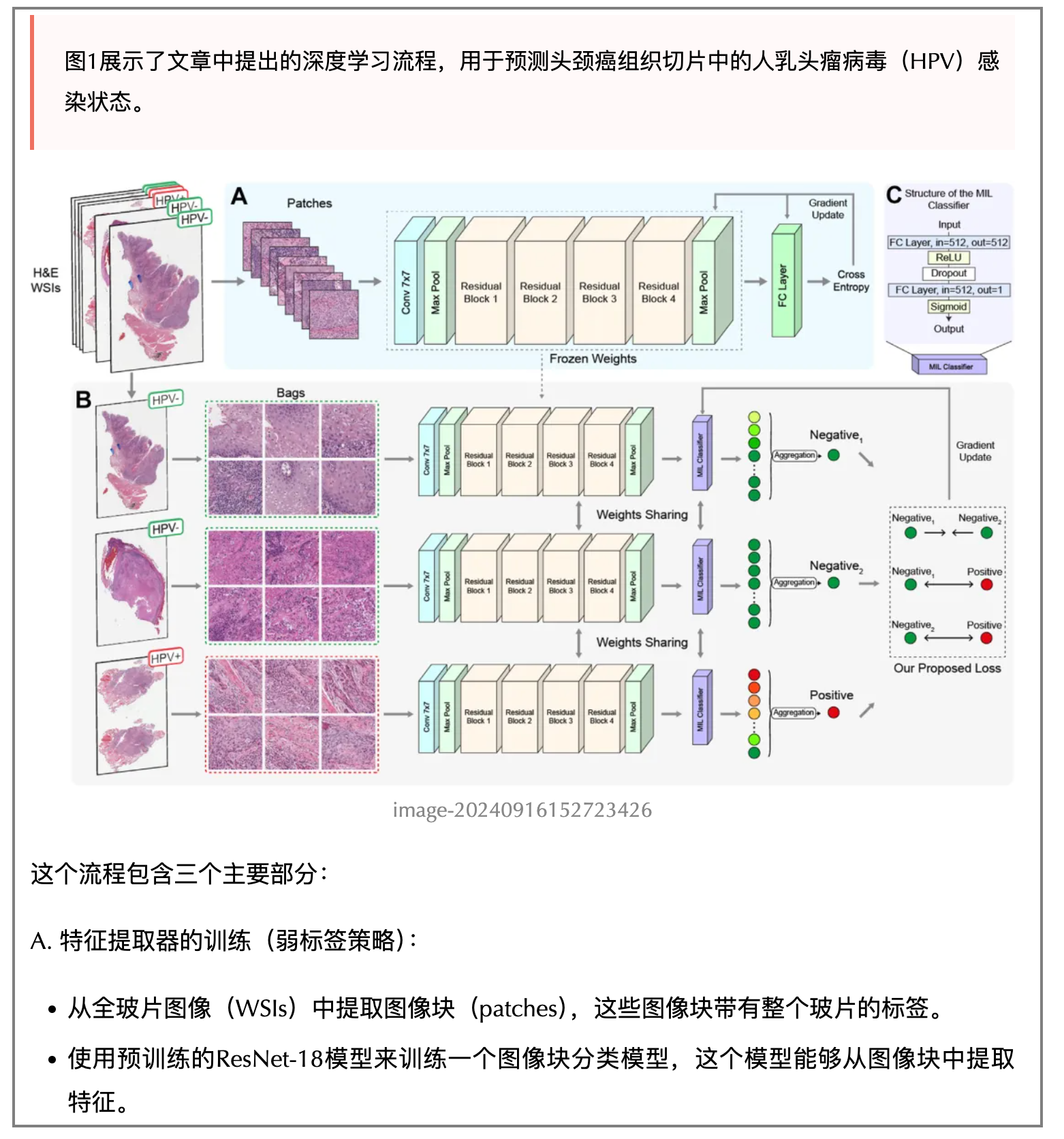
9、从WSI中提取特征
完成预处理的配置后,我们需要按照步骤7设置的分辨率和参数将WSI分割为patch,然后可以执行下列操作进行特征提取:
- 通过Canny边缘检测移除含有太少组织或模糊的瓦片
- 根据Macenko的方法对n个瓦片的H&E染色颜色分布进行标准化(也可以选择其他方法)
- 对每个patch运行特征提取器模型(如CNN、CTransPath)的推理以获取其特征向量
- 将一个WSI的所有n个patch的特征向量连接成一个大的特征矩阵
例如下图的A部分就展示了一个特征提取器的架构。
10、评估图像预处理
我们在执行图像预处理的指令以后,可以先查看命令行是否有报错提示,随后可以导出一个缩略图看看模型的效果(需要在代码中提前设置)。
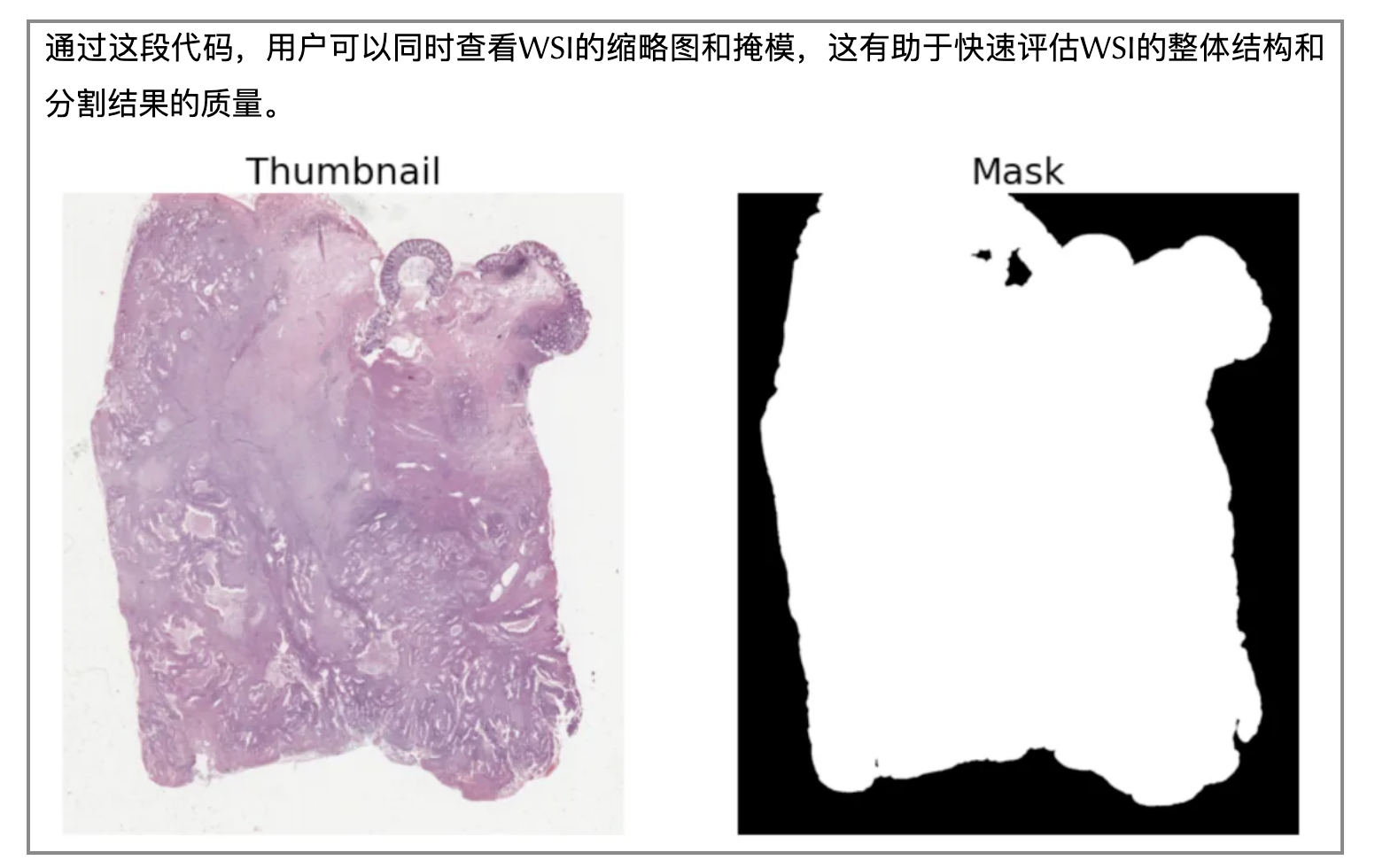
三、建模
时间安排:实际操作3–9小时,总计1–3周
11、定义切片表格
创建一个表格,将匿名患者标识符与提取的切片特征文件名关联起来。如果患者有多张切片,那么属于单个患者的所有特征向量将被连接并作为单一实例处理。
此外,该协议允许对特定切片的标签进行建模,将每个切片视为单独的实例。上述功能的使用取决于切片表格的设计——切片表格是一个文件(.xlsx或.csv格式),其中包含两列:PATIENT列和FILENAME列。
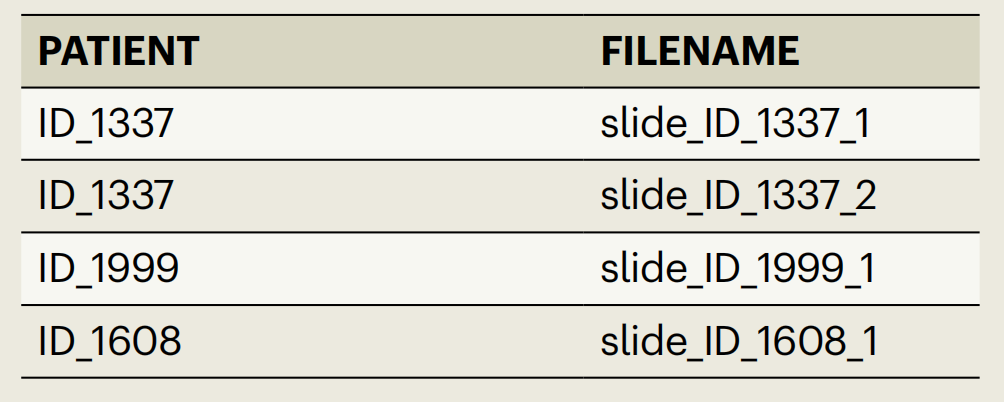
PATIENT列包含伪匿名患者标识符作为字符串,而FILENAME列包含特征矩阵名称作为不带文件扩展名(.h5)的字符串。一个伪匿名患者标识符可以通过重复该标识符并添加相应的额外特征矩阵名称,与多个WSIs相关联。
在下游分类任务中,属于单个患者的所有切片特征将在运行时连接成一个大的特征矩阵,因为所有的切片(注意,这里指已经筛选过后的)都可能包含有价值的信息。
12、定义临床表格
创建一个表格,将匿名患者标识符与生物标志物数据关联起来。
临床表格是一个文件(.xlsx或.csv格式),至少包含两列:一列是伪匿名患者标识符,名为PATIENT;另一列是用于训练模型的生物标志物数据,这是一个不带特殊字符的任意命名字符串。

临床表格可以包含其他列,只要每个列名是唯一的字符串。临床表格的绝对路径用作配置文件建模部分clini_table参数的输入。
要指定要建模的生物标志物,请提供临床表格中相应列名的字符串输入作为target_label参数。除了PATIENT列之外,临床表格中未明确作为参数提供的列名不会用于建模。建议在建模部分使用categories参数定义希望训练的类别。
临床表格中的缺失值应该是空白单元格,因为像‘NA’、‘NaN’、‘None’或‘-’这样的字符串或字符将被视为类别,除非使用categories参数明确定义类别。
13、定义数据分割
为了从WSI直接预测生物标志物而正确开发模型,需要四个主要数据组件:
- 训练数据
- 验证数据
- 测试数据
- 外部测试数据
训练和验证数据在训练期间被模型看到,然后使用未见过的测试数据进行评估。上述数据分割通常来自同一患者队列。
为了测试模型的泛化能力,其性能在未见过的外部测试集上测量。默认情况下,对队列进行五折交叉验证,将步骤12中提供的临床表格分割成五个80-20的排列,用于建模(使用训练和验证数据)和测试数据。因此,提供的队列中有64%用于训练,16%用于验证,20%用于测试,重复五次迭代,按目标变量分层(补充图1)。
5折数据分割的可视化
5折数据分割导致数据的5种80-20分割排列。换句话说,如果有5个患者,其中一个患者被留出用于测试,其余患者用于训练模型。这个过程重复5次,因此每个患者都曾在训练集中4次,并在测试集中1次。因此,使用k折数据分割进行训练将导致训练出k个模型,这些模型具有相同的架构,在训练期间分析了略微不同的患者子集。
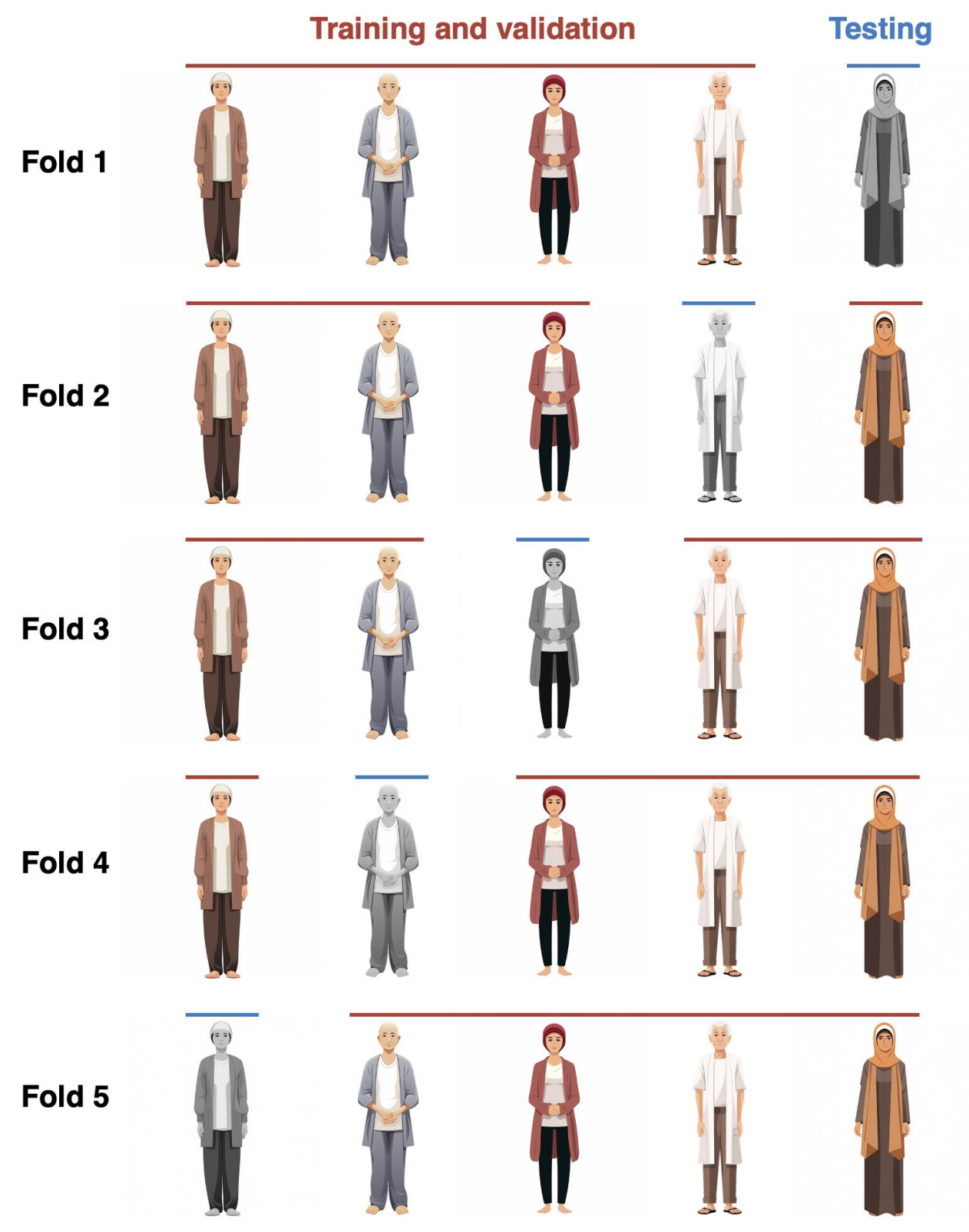
上图展示了如何将数据集分割成5个部分,每个部分都包含不同的训练和测试样本组合。在每次分割中,大约80%的数据用于训练模型,而剩余的20%用于测试。这种交叉验证方法确保了每个患者都有机会成为测试集的一部分,同时也在训练集中多次出现,从而提高了模型训练的效率和泛化能力。
通过这种方式,我们可以得到多个模型,每个模型都在略微不同的数据子集上训练,这有助于评估模型对不同数据样本的稳健性。
在患者级标签的多切片情况下,属于单个患者的所有特征向量被连接起来,并作为单个实例处理。因此,属于单个患者的切片特征只能存在于训练、验证或测试中,从而避免了训练期间的数据泄漏。
在切片特定标签的多切片情况下,拥有外部验证队列至关重要,因为同一患者的不同切片可能出现在训练、验证和测试集中,导致一种数据泄漏,从而产生过于乐观的性能估计。
五折交叉验证后,在整个队列上再进行一次训练运行,以产生单个模型。再次,数据以80-20的比例分割,但这次仅用于训练和验证,因为将使用外部测试集来衡量性能。这种数据分割机制之所以被选择,是因为它在先前研究中的计算病理学建模中一致使用。
外部测试集在模型训练期间未使用且未见,以避免数据泄漏。如果没有外部测试集可用,建议从训练队列中创建伪外部测试集,确保训练和测试队列中的患者切片和来源医院相互排斥,以减少建模期间的混杂因素。
14、设置建模配置
打开配置文件,并在建模部分插入所需的参数(表3)。建模部分的参数用于交叉验证、最终模型训练以及在CLI 命令使用时在外部队列上部署模型。在此步骤中,提供交叉验证所需的参数。
参数示例输入描述
clini_table
/home/storage/clinical_table.xlsx
包含临床参数的表格(.xlsx或.csv)的路径。包含伪匿名患者标识符的列必须命名为PATIENT。包含要预测的生物标志物的列可以命名为任何没有特殊字符的名称。
slide_table
/home/storage/slide_table.csv
链接伪匿名患者标识符到文件名的表格(.xlsx或.csv)的路径。包含伪匿名患者标识符的列必须命名为PATIENT。包含文件名的列必须命名为FILENAME,且条目中不应包含文件扩展名。
feature_dir
/home/storage/output_features/macenko_xiyuewang-ctranspath-7c998680
包含提取的特征文件的目录的路径。
output_dir
/home/storage/modeling_output
保存建模结果的路径。
target_label
isMSIH
要预测的生物标志物的名称。应与临床表格文件中出现的名称相同。
categories
[MSI-H, MSS]
可选的类别指示;否则会自动推断。包含要预测的目标标签的类别。
cat_labels
[STAGE, SEX]
可选的为多模态建模添加分类表格生物标志物。
cont_labels
[AGE]
可选的为多模态建模添加连续表格生物标志物。
n_splits
5
仅适用于交叉验证。训练队列被分割的折数。五折会产生一个5×80/20的训练/测试分割。
model_path
/home/storage/modeling_output/export.pkl
仅适用于模型部署。在外部队列上部署模型的模型路径。
deploy_feature_dir
/home/storage/output_features_external/macenko_xiyuewang-ctranspath-7c998680
仅适用于模型部署。属于要在其上部署模型的外部队列的特征的路径。
15、训练交叉验证模型
使用第14步中的配置运行带有交叉验证的建模流程,CLI命令为 crossval。
默认情况下,交叉验证训练使用第13步描述的五折数据分割。这个过程产生了五个不同的模型,它们具有相同的架构,但由于训练数据的多样性,其学习到的参数存在差异。这允许在各种数据条件下测量模型架构的性能。
16、评估交叉验证
打开配置文件,并在统计部分插入所需的参数。
参数示例输入描述
pred_csvs
-/home/storage/modeling_output/fold-0/patient-preds.csv
-/home/storage/modeling_output/fold-1/patient-preds.csv
-/home/storage/modeling_output/fold-2/patient-preds.csv
-/home/storage/modeling_output/fold-3/patient-preds.csv
-/home/storage/modeling_output/fold-4/patient-preds.csv
包含测试集上患者预测的.csv文件的路径。当在外部队列上评估完全训练的模型时,单个预测文件只需要一个路径。
target_label
isMSIH
要预测的生物标志物的名称。应与训练时的生物标志物名称相同。
true_class
MSI-H
被认为是阳性病例的预测生物标志物的类别。
output_dir
/home/storage/modeling_output
保存统计结果的路径。
使用CLI命令 statistics测量交叉验证模型的性能。接收者操作特征曲线下面积(AUROC)和精度召回曲线下面积(AUPRC)是两个计算指标,理想情况下应尽可能接近1,表明分类的质量。
同时计算95%置信区间(CI);较小的95% CI表明在不同交叉验证模型中的性能更为稳健。
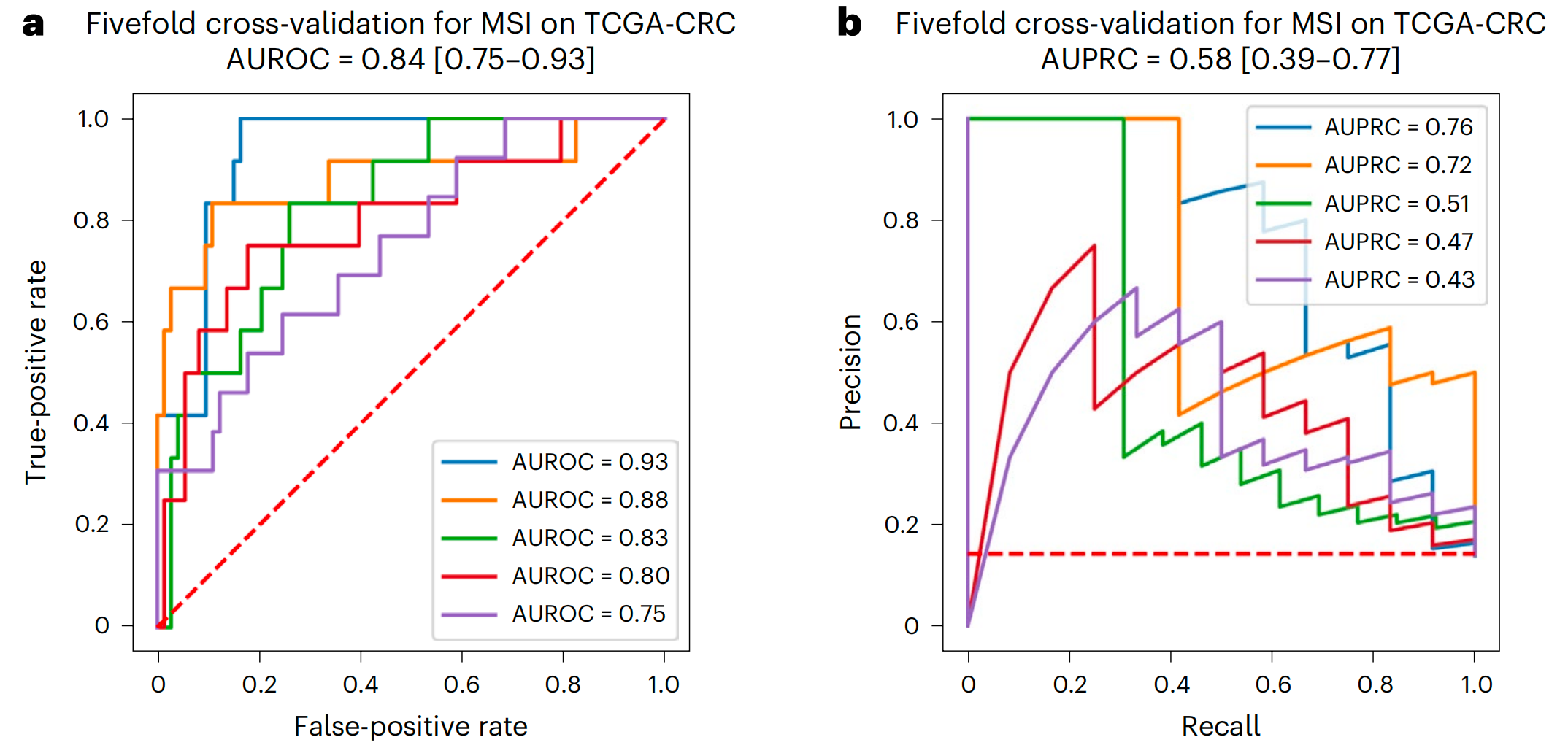
图a中的AUROC值较高,表明模型在整体上具有良好的区分能力;而图b中的AUPRC值相对较低,可能意味着模型在处理不平衡数据集时存在一定的挑战。
17、训练最终模型
打开配置文件,并在建模部分插入所需的参数(表3)。在此步骤中,提供全面训练所需的参数。建议在建模部分的输出参数中选择不同的输出目录,以避免与第15和16步中的交叉验证结果冲突。
使用CLI命令 train运行建模管道以训练最终模型。这个过程产生了一个单一模型文件(.pkl),该文件已在整个队列的100%上进行训练,因此需要外部队列来测量其预测生物标志物的性能。
四、评估
时间安排:实际操作1–2小时,总计1–7天
18、设置外部验证配置
打开配置文件,并在建模部分插入所需的参数(表3)。
在此步骤中,提供在外部队列上部署模型所需的参数。重复步骤7–10以预处理外部队列并获得用于验证最终模型的特征向量。
重复步骤11和12以获取外部队列相应的切片和临床表格。
19、部署最终模型
使用步骤18中的配置,通过CLI命令 deploy运行建模管道,将最终模型部署在外部队列上。在将模型部署到新数据时,模型不会进行训练或更新。
模型在外部队列上的部署输出是一个包含每个患者预测的单个文件。
20、评估最终模型
首先,使用包含患者预测的单个文件更新配置文件(表4)。

其次,使用CLI命令 statistics。预测结果通过案例重采样1,000次进行引导,重新计算指标,从而得到外部验证队列建模性能的95% CI(参见步骤22)。
21、生成空间可解释性热图
首先,检查步骤20中用于计算指标的患者预测文件。此处列出了每个患者的预测得分,并按损失排序;最准确的预测位于文件顶部。
其次,选择要分析空间可解释性地图的患者。建议选择预测得分极端值的患者(即得分最接近1的患者)。更极端的预测得分值表明模型捕捉到了更强的信号,这有助于人类对热图的解释。
第三,选择属于这些患者的切片名称,观察步骤18中的切片表格文件。打开配置文件,并在热图部分插入所需的参数(表5)。
使用CLI命令 heatmaps生成切片热图。这产生了一个热图,显示了对于模型切片级别预测最重要的瓦片的空间关系,增加了模型决策的可解释性。
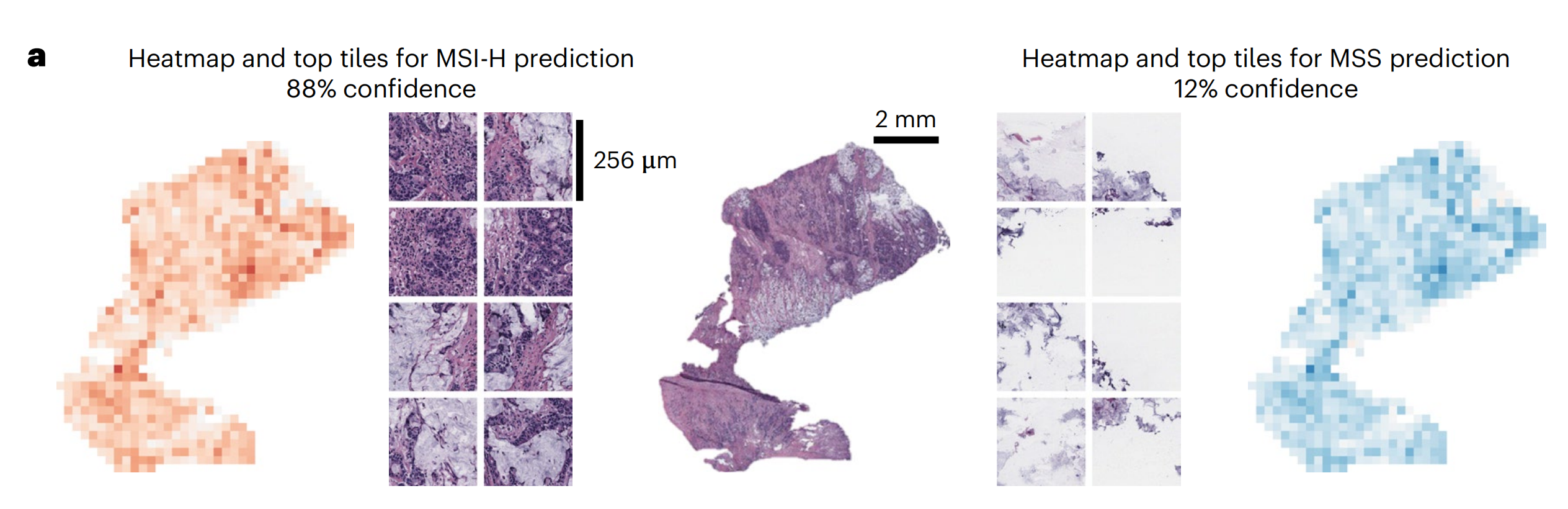
此外,用于决策制定所需的影响性瓦片数量作为带有得分和坐标的单独图像文件存储。
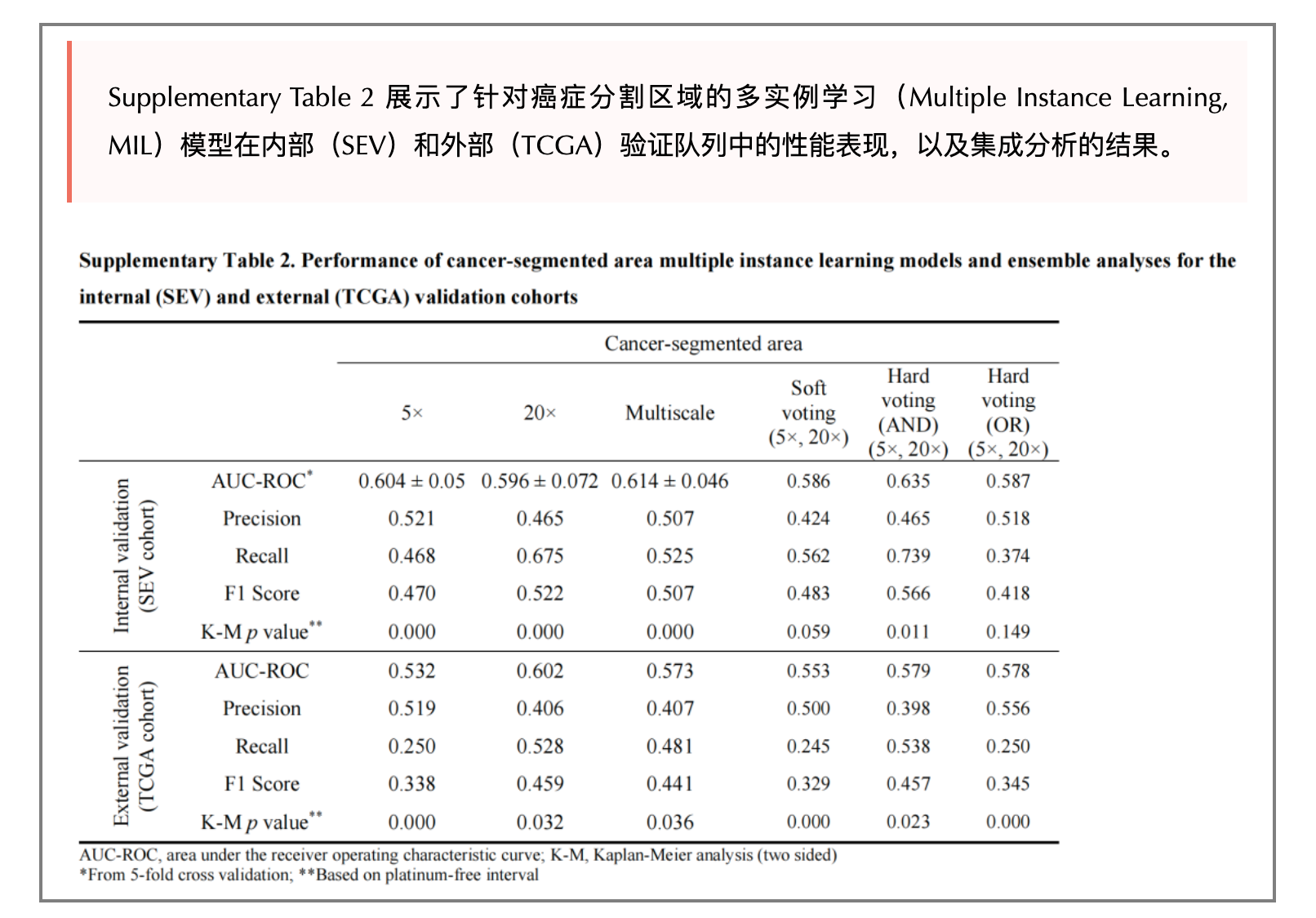
22、审查模型的性能
常见的评估指标:区域下接收者操作特征曲线(Area Under the Receiver Operating Characteristic Curve, AUC-ROC)、精确度(Precision)、召回率(Recall)、F1分数(F1 Score)和Kaplan-Meier(K-M)p值。
五、临床解释
时间安排:实际操作1–2周,总计2–6周
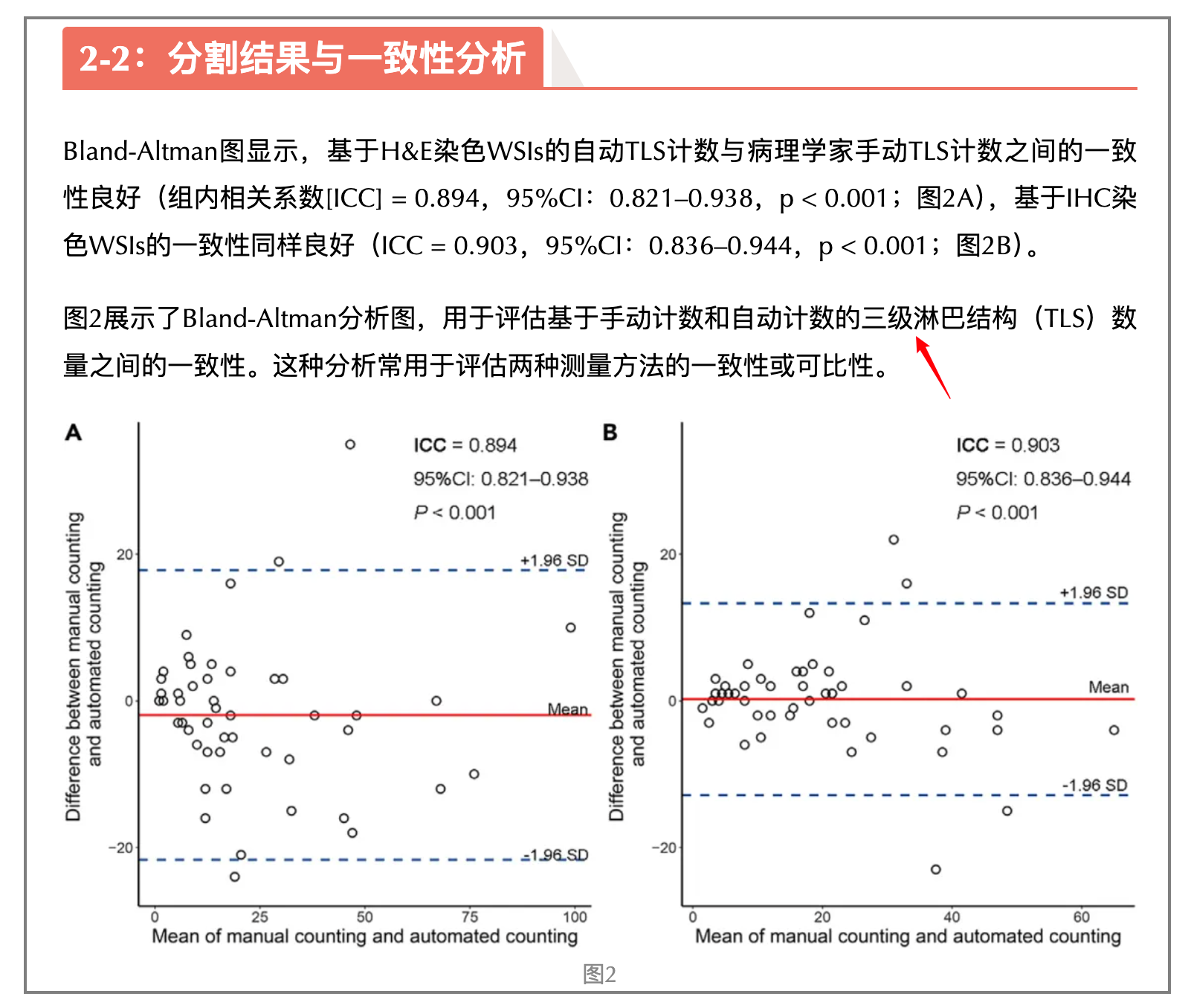
23、进行一致性分析
部分文章可能不会涉及这一部分,但是我觉得包含这一部分可以增强模型的可解释性,因为这一步就是为了寻找与已知的临床概念相符合的建模方式,或者说通过这一步我们可以间接评估模型的预测性能。
24、审查模型的临床效用
让病理学家审查步骤21中影响模型决策制定的空间可解释性热图和相应patch。将病理学家的发现与步骤23中的一致性分析结果与先前研究的文献联系起来,以发现开发的生物标志物是否与已知的生物学和医学概念相符。
25、计算预后能力
如果我们临床问题落在预后,那么我们大概率会涉及以下方面:
- 患者分层:根据我们提取的生物标志物将患者区分为高危和低危
- 使用Kaplan-Meier曲线可视化风险组的分层
- 使用对数秩和检验计算分层风险组之间不同生存率的统计显著性
- 使用最终模型对外部队列的预测标签和协变量(如肿瘤分期、年龄和性别)进行多变量Cox比例风险检验
版权归原作者 罗小罗同学 所有, 如有侵权,请联系我们删除。