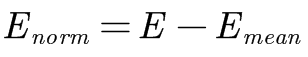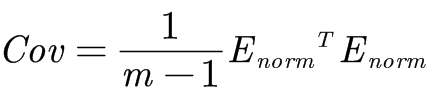目录
前言介绍:
1、PCA降维:
(1)概念解释:
PCA,全称Principal Component Analysis,即主成分分析。是一种降维方法,实现途径是提取特征的主要成分,从而在保留主要特征的情况下,将高维数据压缩到低维空间。
在经过PCA处理后得到的低维数据,其实是原本的高维特征数据在某一低维平面上的投影(只要维度较低,都可以视为平面,例如三维相对于四维空间也可以视为一个平面)。虽然降维的数据能够反映原本高维数据的大部分信息,但并不能反映原本高维空间的全部信息,因此要根据实际情况,加以鉴别使用。
(2)实现步骤:
PCA主要通过6个步骤加以实现: 1、**标准化**(将原始数据进行标准化,一般是去均值,如果特征在不同量级上,还要将矩阵除以标准差) 具体:
其中,E为原始矩阵,Emean为均值矩阵,Enorm为标准化矩阵。 2、**协方差**(计算标准化数据集的协方差矩阵) 具体:
其中,Cov为协方差矩阵,m为样本的数量,Enorm为均值矩阵。 3、**特征量**(计算协方差矩阵的特征值和特征向量) 具体: 假设实数λ、n行(原始矩阵E的列数即为n)1列的矩阵X(即n维向量)满足下式:
则λ为Cov的特征值,其中Cov为协方差矩阵。 4、**K 特征**(保留特征值最大的前K个特征(K是降维后,我们期望达到的维度)) 具体: 若有多个特征值,则保留前K个最大的特征值,以满足之后的计算需求。 5、**K 向量**(找到这K个特征值对应的特征向量) 具体: 通过步骤3中的公式得到每个特征值对应的特征向量。 6、**得降维**(将标准化数据集乘以该K个特征向量,得到降维后的结果) 具体: 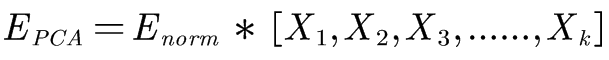 其中,Epca为最后要求得的PCA降维矩阵,Enorm为标准化矩阵,X1、X2、X3、...、Xk为对K个特征值对应的特征向量。
(3)优劣相关:
**优点**: 1.PCA降维之后的各个主成分之间相互正交,可**消除**原始数据之间**相互影响的因素**。 2.PCA降维的计算过程并不复杂,因此实现起来**较简单容易**。 3.在**保留大部分主要信息**的前提下,起到了降维,**简便化计算**效果。
**缺点**: 1.特征主成分的定义具有**模糊性**,**解释性差**。 2.PCA降维选取令原数据在新坐标轴上方差最大的主成分的标准,使得一些方差小的特征较易丢失,**有损失重要信息的可能性**。
2、DBSCAN聚类:
(1)概念解释:
密度聚类亦称“基于密度的聚类”(Density-Based Clustering),此类算法假设聚类结构能通过样本分布的**紧密程度**确定。通常情形下,密度聚类算法从样本密度的角度来考察样本之间的**可连续性**,并基于可连接样本不断**扩展聚类簇**以获得最终的聚类结果。 **DBSCAN**(Density-Based Spatial Clustering of Applications with Noise)就是这样一种聚类算法,该算法基于一组**“领域”**(neighborhood)参数(ε,MinPts)来刻画样本分布的**紧密程度**。
(2)算法原理:
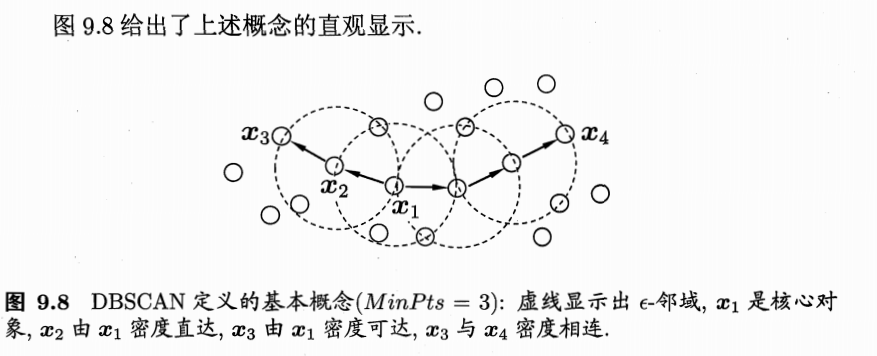
 给定数据集D={x1,x2,...,xm},定义下面这几个概念:
理解了相关概念之后,下面给出算法实现的**伪代码**:
(3)优劣相关:
优点: 1、能够识别**任意形状**的样本。 2、该算法将具有足够密度的区域划分为簇,并在**具有噪声的空间**数据库中发现任意形状的簇。 3、**无需指定**簇个数,而是由算法自主发现。
缺点: 1、**需要指定**最少点个数(MinPts)与半径(ε)。(但其实相对其他聚类算法来说,已经具有较大的自由性。) 2、最少点个数与半径对算法的**影响较大**,一般需多次调试。
代码实现:
0、数据准备:
在这里,我们使用**sklearn库的鸢尾花iris数据集**(sklearn.datasets.load_iris)作为测试数据样本。iris数据集包含**150**个样本,每个样本包含**四个属性特征**(花萼长度、花萼宽度、花瓣长度、花瓣宽度)和一个**类别标签**(分别用0、1、2表示山鸢尾、变色鸢尾和维吉尼亚鸢尾)。
首先,我们要**安装sklearn库**。安装此库,还是通过pip install命令,但是并不是pip install sklearn,而是**pip install scikit-learn**。正如我们调用opencv是import cv2,而安装却是通过pip install opencv一样。
pip install scikit-learn
然后,获取数据集,其中**x**为鸢尾花的**特征**数据集(数据类型为**数组numpy.adarray**),**y**为鸢尾花的**标签**数据集(数据类型为**数组numpy.adarray**) 。
from sklearn.datasets import load_iris
x = load_iris().data
y = load_iris().target
1、PCA降维:
import numpy as np
def PCA_DimRed(dataMat,topNfeat): #PCA_DimRed--PCA dimension reduction,PCA降维
meanVals = np.mean(dataMat, axis=0)
meanRemoved = dataMat - meanVals # 标准化(去均值)
covMat = np.cov(meanRemoved, rowvar=False)
eigVals, eigVets = np.linalg.eig(np.mat(covMat)) # 计算矩阵的特征值和特征向量
eigValInd = np.argsort(eigVals) # 将特征值从小到大排序,返回的是特征值对应的数组里的下标
eigValInd = eigValInd[:-(topNfeat + 1):-1] # 保留最大的前K个特征值
redEigVects = eigVets[:, eigValInd] # 对应的特征向量
lowDDatMat = meanRemoved * redEigVects # 将数据转换到低维新空间
# reconMat = (lowDDatMat * redEigVects.T) + meanVals # 还原原始数据
return lowDDatMat
2、DBSCAN聚类:
import numpy as np
import random
import copy
def DBSCAN_cluster(mat,eps,min_Pts): #进行DBSCAN聚类,优点在于不用指定簇数量,而且适用于多种形状类型的簇
k = -1
neighbor_list = [] # 用来保存每个数据的邻域
omega_list = [] # 核心对象集合
gama = set([x for x in range(len(mat))]) # 初始时将所有点标记为未访问
cluster = [-1 for _ in range(len(mat))] # 聚类
for i in range(len(mat)):
neighbor_list.append(find_neighbor(mat, i, eps))
if len(neighbor_list[-1]) >= min_Pts:
omega_list.append(i) # 将样本加入核心对象集合
omega_list = set(omega_list) # 转化为集合便于操作
while len(omega_list) > 0:
gama_old = copy.deepcopy(gama)
j = random.choice(list(omega_list)) # 随机选取一个核心对象
k = k + 1
Q = list()
Q.append(j)
gama.remove(j)
while len(Q) > 0:
q = Q[0]
Q.remove(q)
if len(neighbor_list[q]) >= min_Pts:
delta = neighbor_list[q] & gama
deltalist = list(delta)
for i in range(len(delta)):
Q.append(deltalist[i])
gama = gama - delta
Ck = gama_old - gama
Cklist = list(Ck)
for i in range(len(Ck)):
cluster[Cklist[i]] = k
omega_list = omega_list - Ck
return cluster
3、代码汇总:
from sklearn.datasets import load_iris
import numpy as np
import random
import copy
import matplotlib.pyplot as plt
def PCA_DimRed(dataMat,topNfeat): #PCA_DimRed--PCA dimension reduction,PCA降维
meanVals = np.mean(dataMat, axis=0)
meanRemoved = dataMat - meanVals # 标准化(去均值)
covMat = np.cov(meanRemoved, rowvar=False)
eigVals, eigVets = np.linalg.eig(np.mat(covMat)) # 计算矩阵的特征值和特征向量
eigValInd = np.argsort(eigVals) # 将特征值从小到大排序,返回的是特征值对应的数组里的下标
eigValInd = eigValInd[:-(topNfeat + 1):-1] # 保留最大的前K个特征值
redEigVects = eigVets[:, eigValInd] # 对应的特征向量
lowDDatMat = meanRemoved * redEigVects # 将数据转换到低维新空间
# reconMat = (lowDDatMat * redEigVects.T) + meanVals # 还原原始数据
return lowDDatMat
def find_neighbor(data,pos,eps): #寻找相邻点函数
N = list()
temp = np.sum((data-data[pos])**2, axis=1)**0.5
N = np.argwhere(temp <= eps).flatten().tolist()
return set(N)
def DBSCAN_cluster(data,eps,min_Pts): #进行DBSCAN聚类,优点在于不用指定簇数量,而且适用于多种形状类型的簇,如果使用K均值聚类的话,对于这次实验的数据(条状簇)无法得到较好的分类结果
k = -1
neighbor_list = [] # 用来保存每个数据的邻域
omega_list = [] # 核心对象集合
gama = set([x for x in range(len(data))]) # 初始时将所有点标记为未访问
cluster = [-1 for _ in range(len(data))] # 聚类
for i in range(len(data)):
neighbor_list.append(find_neighbor(data, i, eps))
if len(neighbor_list[-1]) >= min_Pts:
omega_list.append(i) # 将样本加入核心对象集合
omega_list = set(omega_list) # 转化为集合便于操作
while len(omega_list) > 0:
gama_old = copy.deepcopy(gama)
j = random.choice(list(omega_list)) # 随机选取一个核心对象
k = k + 1
Q = list()
Q.append(j)
gama.remove(j)
while len(Q) > 0:
q = Q[0]
Q.remove(q)
if len(neighbor_list[q]) >= min_Pts:
delta = neighbor_list[q] & gama
deltalist = list(delta)
for i in range(len(delta)):
Q.append(deltalist[i])
gama = gama - delta
Ck = gama_old - gama
Cklist = list(Ck)
for i in range(len(Ck)):
cluster[Cklist[i]] = k
omega_list = omega_list - Ck
return cluster
if __name__ == "__main__":
#1、准备数据
x = load_iris().data
y = load_iris().target
#2、PCA降维
pro_data = PCA_DimRed(x,2)
#3、DBSCAN聚类(此步中要保证数据集类型为数组,以配合find_neighbor函数)
pro_array = np.array(pro_data)
thecluster = DBSCAN_cluster(pro_array,eps=0.8,min_Pts=30)
#4、展示降维效果:
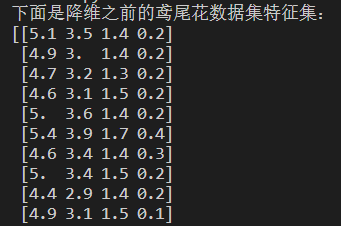
print("下面是降维之前的鸢尾花数据集特征集:")
print(x)
print("下面是降维之后的鸢尾花数据集特征集:")
print(pro_data)
#5、展示聚类效果:
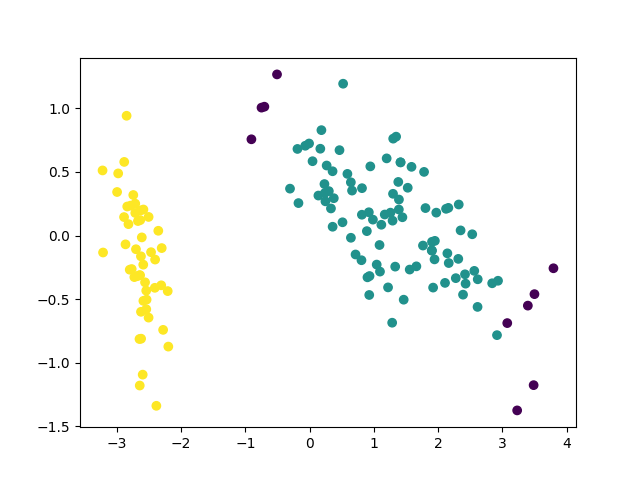
plt.figure()
plt.scatter(pro_array[:, 0], pro_array[:, 1], c=thecluster)
plt.show()
实现效果:
1、降维效果:
降维之前的鸢尾花数据集特征集:
降维之后的鸢尾花数据集特征集:
2、聚类效果:
可以看出来,DBSCAN聚类方法并不能很准确地根据PCA降维后的鸢尾花特征集对鸢尾花样本进行聚类,原因是变色鸢尾与维吉尼亚鸢尾的样本特征较近,两者更类似于同属于一个密度空间,因而导致了该实验的不准确性。
但是,其实也可以看出,山鸢尾与其他两种鸢尾能够进行较好的区别 ,说明该方法仍适用于不同类别样本间差距较大的聚类情形。
写在最后:
本篇文章主要介绍了PCA降维、DBSCAN聚类这两个机器学习操作的基本原理,以及两者结合的用于实际数据处理的方法。
可能基于PCA降维的DBSCAN聚类的方法不是很适用于sklearn库中的鸢尾花数据集,但是该方法既具有处理高维数据的能力,也能够处理各种形状的簇,说明其作为一套较为完整的聚类方法,仍然具有较为广阔的应用场景。
希望大家能够积极应用这个方法,使得其拥有更多的应用可能性。谢谢各位!
参考书籍:
周志华.机器学习[M].北京:清华大学出版社,2016.01
参考文章:
六种常见聚类算法:http://t.csdn.cn/Urhn9
Python PCA(主成分分析法)降维的两种实现:http://t.csdn.cn/NlAeU
DBSCAN聚类算法Python实现:http://t.csdn.cn/lkFhF
PCA降维原理 操作步骤与优缺点:http://t.csdn.cn/QiEJM
好了以上就是所有的内容,希望大家多多关注,点赞,收藏,这对我有很大的帮助。谢谢大家了!

好了,这里是Kamen Black 君。祝国康家安,大家下次再见喽!!!溜溜球~~

版权归原作者 Kamen Black君 所有, 如有侵权,请联系我们删除。