前言
背景:项目需要爬取来自MCE制药公司网页展示的药物数据
网页示例:Mitophagy激活剂、基因 | MCE
难点:数据不太好取+我懒得复习request+beautifulSoup
需求分析
遍历下图展示的网页中超链接对应的具体产品页面
如图共6页,每页约展示20个产物(紫色字体包含超链接)
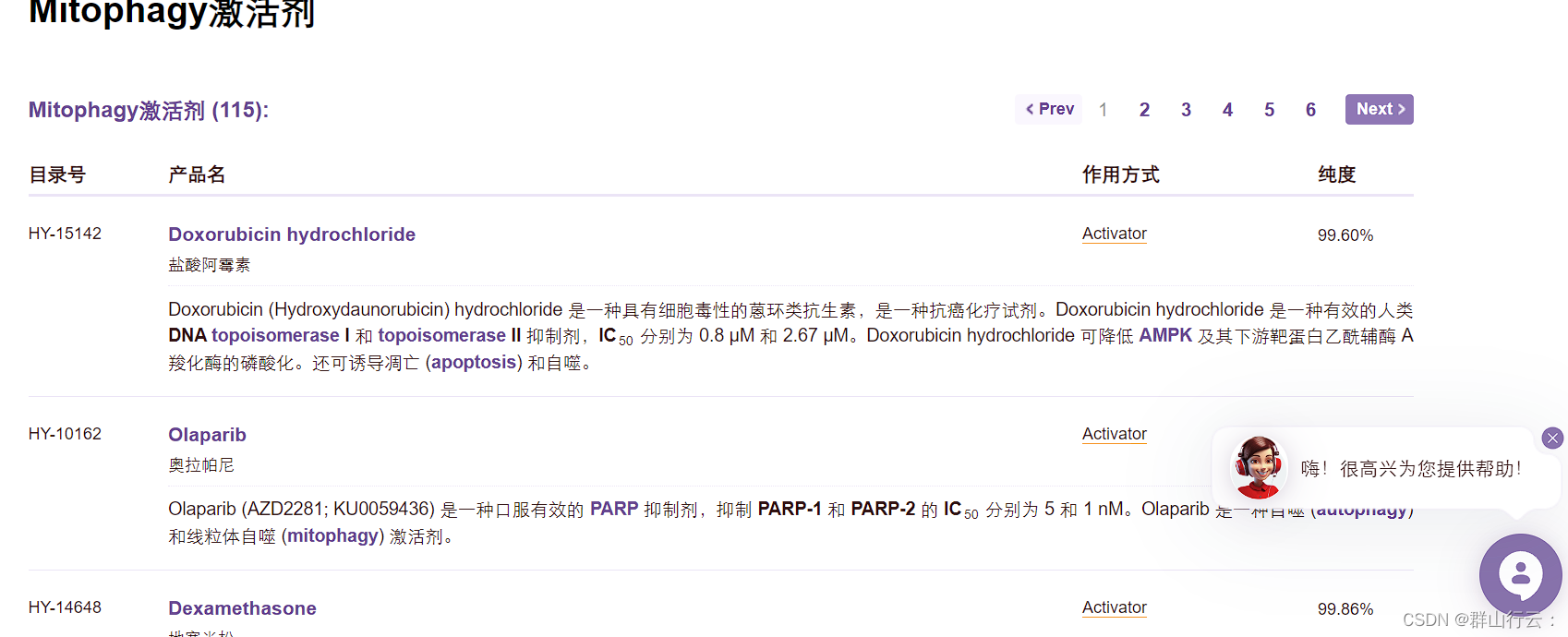
点击紫色字体后会打开新标签页,如下图所示
真正需要采集的是其中的药物学名、描述和图片等数据

框架
比beautifulSoup更好用的DrissionPage库,你值得拥有
DrissionPage 是一个基于 python 的网页自动化工具。
它既能控制浏览器,也能收发数据包,还能把两者合而为一。
可兼顾浏览器自动化的便利性和 requests 的高效率。
它功能强大,内置无数人性化设计和便捷功能。
它的语法简洁而优雅,代码量少,对新手友好。
👆来自DrissionPage官网,不是我写的,但我完全认同
DrissionPage中定义了三种对象(第三种对象实际上为前两种的结合,故不作介绍):
SessionPage
实现收发数据包,即不需要控制浏览器的情况下,调用此对象的方法可以快速的抓取网站数据
如果每个药物网页的链接有规律可循,则可使用此对象,不操控浏览器就能抓取数据
然而,该网站中的所有药物网页均以学名来命名,没有规律或区间,如下图

每个药物的页面只能通过图一这样的列表展示页来逐一访问,故使用下方的ChromiumPage对象
ChromiumPage
该对象实现对浏览器的控制,并且可以非常方便的控制网站中的标签页。
元素定位和获取
上述两种对象都可以通过相对简单的语法规则定位并获取HTML元素。
如匹配HTML中的id标签,只需要"#"符号
# 在页面中查找id属性为one的元素
ele1 = page.ele('#one')
# 在ele1元素内查找id属性包含ne文本的元素
ele2 = ele1.ele('#:ne')
而匹配HTML中的class标签则是'.'符号
# 查找class属性为p_cls的元素
ele2 = ele1.ele('.p_cls')
# 查找class属性'_cls'文本开头的元素
ele2 = ele1.ele('.^_cls')
对于其他标签,可以通过'tag'进行匹配,同时可以采用'@'符号匹配属性,或通过'@@'符号匹配多种属性(逻辑为'与',类似&&)
# 定位div元素
ele2 = ele1.ele('tag:div')
# 定位class属性为p_cls的p元素
ele2 = ele1.ele('tag:p@class=p_cls')
# 定位文本为"第二行"的p元素
ele2 = ele1.ele('tag:p@text()=第二行')
# 查找name属性为row1且class属性包含cls文本的元素
ele2 = ele1.ele('@@name=row1@@class:cls')
在取得了某项元素后,该框架也提供了便捷的属性访问方法。
例如对于HTML代码
<div id="div1" class="divs">Hello World!
<p>行元素</p>
</div>
通过.text可以返回元素内所有文本组合成的字符串(不包含任何标签内的文字)
print(ele.text)
运行结果为
Hello World!
行元素
而对于含有链接的HTML代码如
<a href='http://www.baidu.com'>百度</a>
使用.link可以获取其中的链接,包括href和src
print(a_ele.link)
运行结果为
http://www.baidu.com
更多方法和参数请查阅DrissionPage官网
实战
在快速学习过上述基础后,就可以进入实战,主要放上代码和注释
首先正常导入库,并设定参数
考虑到部分药物网页可能缺少某个元素,提前设置好填充值
# 导入必要的库
from DrissionPage import ChromiumPage
from DrissionPage.common import Settings
# 创建页面对象
page = ChromiumPage()
# 如果元素找不到怎么办?先设置不报错
Settings.raise_when_ele_not_found = False
# 然后设置一个默认值进行填充
page.set.NoneElement_value('')
使用 page.get,打开图1中的列表展示页面,这里仅展示爬取第一页的内容
page.get(f'https://www.medchemexpress.cn/Targets/Mitophagy/effect/activator.html?page=1')
若需要爬取多页,则使用循环并修改page即可
for i in range(1, n):
page.get(f'https://www.example.com?page={i}')
接下来开始定位元素,使用浏览器的审查元素功能

可以看出所有药物网页均被包裹在如上图所示的'id=sub_ctg_list_target'中
而每个具体的网页链接则被包裹在标签内

使用框架的ele方法(返回整个块,然后对块使用eles方法(返回块内所有标签组成的列表)
然后对获取到的药物列表进行遍历
# 获取产品列表
list = page.ele('#sub_ctg_list_target')
links = list.eles('tag:a')
for link in links:
然而这种采集方法会采集到一些额外的超链接,如下图所示详情页面中有时会介绍药物对应的受体,而受体同样包含超链接

同样由上图右侧,可以注意到所有受体对应的链接均位于网站的'/Targets'目录下,故采用框架提供的文本匹配方法来筛选掉受体超链接,和网页中用于阻止浏览器默认超链接的'javascript:(0);'
然后使用tab对象操作标签页,new_tab()方法建立新标签页,get方法使标签页转向我们刚刚获取并初步筛选过的超链接。link.link即获取link对象
if link.link != 'javascript:(0);' and ('Targets' not in link.link):
tab = page.new_tab()
tab.get(link.link)
同时注意到列表中的部分药物除英文名外还有中文名称,并且中文名称包含重复的超链接
为了防止重复打开标签页和采集,观察两种名称的HTML元素
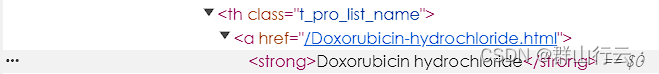
 可以发现两种名称的父元素不同,因此使用框架中的parent方法进行二次筛选
可以发现两种名称的父元素不同,因此使用框架中的parent方法进行二次筛选
# 判断link的父元素,如果是th说明是英文对应超链接,p则为中文超链接。使用.tag获取
tag = link.parent(1).tag
# 部分药剂有中文和英文名字,两个名字都包含相同超链接,造成重复采集;
# 只选取英文超链接
if tag == 'th':
接下来就开始正式定位和采集了
观察具体药物的网页,以几张不同的元素为例
简单的文本元素

上图是药物名称的HTML代码,可以看到name元素被包裹在<id>中,使用'#'符号匹配即可
# .text方法获取内部文本
name = tab.ele('#head_pro_name').text
表格包裹的元素
这种稍微麻烦一些


可以看到左侧为
# 先搜索tr结构,然后搜索指定的td列
formula = tab.ele('tag:tr@@text():生物活性').ele('.details_info_td').text
代码有点长,其实就是双重ele,首先使用'tag'匹配该HTML页面中的所有
然后使用第二次ele,从
图片元素


项目还需要采集网页中的图片,并且将图片也爬取到本地保存。我们分两步来,获取图片,控制浏览器下载图片。
观察HTML代码可知图片的链接被包裹在<id>中,使用如下代码匹配
image_source = tab.ele('#pro_structure_img')
但匹配得到的其实是整个标签,而我们需要的只是其中的src部分。故使用框架提供的attr()方法获取src,并且通过此方法获取的src是包括网站前缀的完整链接,不需要手动补全
url = image_source.attr('src')
获取图片url后,框架也提供了便捷的下载方法,可以控制标签页对象使用浏览器的下载方式。这里注意到网页提供的图片都是.gif后缀,并且命名不统一,我们可以手动去修改这些参数
# 定义保存路径
savepath = r'E:\exampleImage'
# 调用标签页对象的下载方法
# 修改后缀名,并重命名文件
res = tab.download(url, savepath, suffix='png', rename=f'{flag}.' + name)
数据保存和网站协议
数据的存储使用pandas提供的DataFrame对象即可,图片使用WPS一件插入即可
raw_data = {'Name': name, 'Formula': formula...etc}
df = pd.concat([df, pd.DataFrame([raw_data])], ignore_index=True)
网站带有爬虫协议,本项目认真阅读并遵守了协议内容。
后记
使用该框架大大提高了本项目爬虫部分的效率,并且相当简单易用。但还有一些值得改进和思考的地方,例如缺少某项数据,需要使用预填充值的网页,其爬取速度要远低于数据齐全的网页。具体来说,数据齐全网页打开后2~3秒左右自动关闭,而数据缺少网页需要至少10秒时间。单纯的数据填充不应该产生这么大的影响,还需要继续学习框架寻找原因。
版权归原作者 群山行云: 所有, 如有侵权,请联系我们删除。