python 利用Bio.Blast.Applications 完成类似命令行的本地blast
biopython中常用的blast,包括其说明书(链接)中都没有介绍如何建立本地数据库。
然而,由于我们在工作中可能会需要进行本地blast,并且利用本地建立的数据库作为使用。要从程序中跳出去,要求用户自己在cmd命令行中输入blast的语句,会显得程序十分不完善。
因此,博主查阅了很多方法,最终找到了一个最有效的调用库的方法。
本文将包含两种类型的思路(虽然只有第二个思路走通了,但是博主认为第一种方法更加灵活应变,今后也会努力寻找能走通的方式,一次完善本篇文章):
1.使用os,直接写入命令行语句【失败】
2.调用方法【成功】
1.import os
这种方法博主参考了一下几篇博客:
python执行cmd命令用Python从FASTA生成Blast数据库python command line 包_python – 使用NcbiblastxCommandline自定义blast d…Python中执行系统命令的四种方法windows 下 python 连续执行 cmd 命令(多行)
主要尝试包括了:
i.逐行输入cmd命令
ii.统一写为一大段命令
iii.使用os.system
iv.使用os.popen
尝试代码如下:
import os
'''一大段命令一起执行【失败】'''
mingling ='E: & \ cd testdb & \ makeblastdb -in not_hit_file.fasta -dbtype nucl & \ blastn -query need_blast.fasta -db not_hit_file.fasta -out need_blast.txt -outfmt 6''''尝试不切换文件夹直接使用ncbi安装目录下的bin目录进行执行【失败】'''
mingling ='C:/Program Files/NCBI/blast-2.11.0+/bin/blast_formatter.exe -in E:/testdb/not_hit_file.fasta -dbtype nucl'
以上方法执行完毕之后没有文件夹中没有任何反应。期待大佬提点。
2.from Bio.Blast.Applications import NcbimakeblastdbCommandline
这部分博主参考了以下文章:
将blast等工具的命令行写入到biopython的代码脚本中怎么用python实现序列比对_序列比对在biopython中的处理利用Biopython在线或者在本地环境运行blast
这几篇文章中都各自涉及了博主需要的一部分,并且第一篇文章在import时打错字了。但是根据本地blast的命令,可以猜到本来需要import什么库。
首先本地blast建库,引用方式如下:
from Bio.Blast.Applications import NcbimakeblastdbCommandline
然后,博主要blast的是核酸序列因此引用方式如下:
from Bio.Blast.Applications import NcbiblastnCommandline
如果是想要使用其他blast,如blastp,blastx,根据博主阅读的几篇文章(以及瞎改的经验),应该是把博主引用中blast后面的字母改一下就行了hhh。
之后就是执行命令的代码:
makedb = NcbimakeblastdbCommandline(dbtype ='nucl',input_file ='E:/testdb/not_hit_file.fasta',out ='E:/testdb/not_hit_file.fasta')
stdout,stderr = makedb()
blastn = NcbiblastnCommandline(query ='E:/testdb/need_blast.fasta', db ='E:/testdb/not_hit_file.fasta',outfmt =6, out ='E:/testdb/need_blast.txt')
stdout,stderr = blastn()
然后就成功得到比对结果啦!
对于命令中传入的参数,博主还没有找到这个库的说明书网页,但是基本上可以根据本地blast命令行中的参数进行尝试,如果失败了,就删改以下试试,比如使用首字母或使用常用简称等。如果米娜桑找到了说明书网页,如果可以评论区给博主那就太好啦~~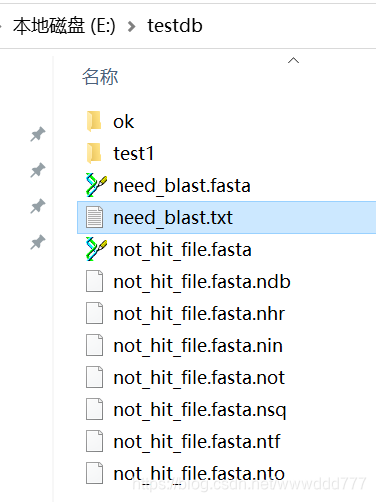
版权归原作者 wwwddd666 所有, 如有侵权,请联系我们删除。