cryoSPARC是当前冷冻电镜单颗粒重构的主流软件之一,它的界面交互十分友好。本文采用一套铁蛋白apoferritin的数据作为testdata。我们服务器路径在/data/testdata/apoferritin下。别的朋友有需要的可以通过网盘下载,放在评论区。本人也是新手,文章有不对的或者补充的地方欢迎指出,大家一起讨论进步。
这是cryoSPARC v4.0的主界面,cryoSPARC软件是通过Web网页来访问的。
点击最左边第二个按钮可以显示所有的project
** 第一步:新建Project**
点击上方绿色的New Project按钮可以新建Projet。
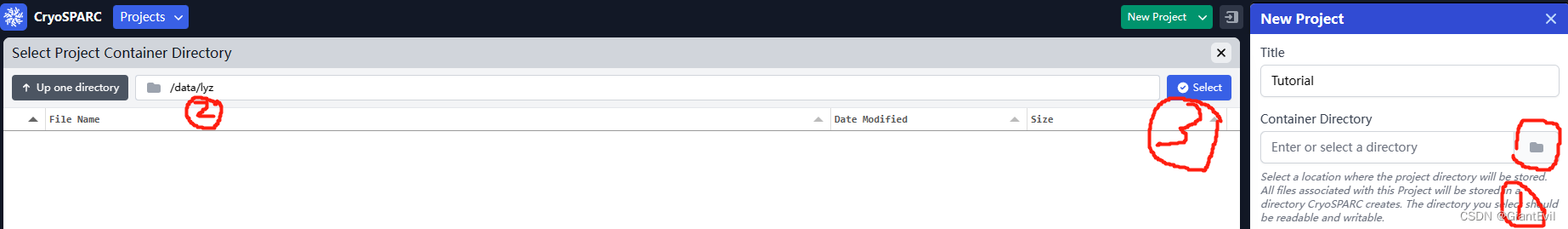
Title输入Project的名称,点击Container Directory右边的文件夹图标选择保存的路径并点击Select确认。下方点击Create新建。
注意:它会在选择的文件夹内创建一个“CS-xxx”的文件夹,所有的运算结果都在这个文件夹下。但是和relion不同,它可以允许访问外部文件夹的文件或者数据。最好别在服务器的各种路径下到处生成Project文件夹,一不小心就被管理员清掉了,在自己的文件夹下新建,做好管理和规划。
第二步:创建Workspace
创建Project之后会自动要求你先创建一个Workspace,输入个自己看得懂的名字比如W1。不同worksparce之间是可以互相使用计算的结果的,所以创建Workspace方便分类和查找自己的计算。
新的Workspace会显示如下,因为是空的,所以它给你推荐了一些可能需要的Job类型,一般是Import导入数据。右上角Detail显示Workspace或者Job的信息,点击Builder可以新建所有类型的Job。

** 第三步:导入数据**
冷冻电镜收集的原始图片一般是以Mutiframes Movies形式(比如SeriesEM软件收集的Tiff格式,EPU软件收集的eer格式)。
如果在收集的时候就做过MotionCorretion,或者在别的软件里比如relion、MotionCor2等做过MotionCorretion,那么是文件就是mrc格式的Micrograph,直接选择Import Micrograph就行。
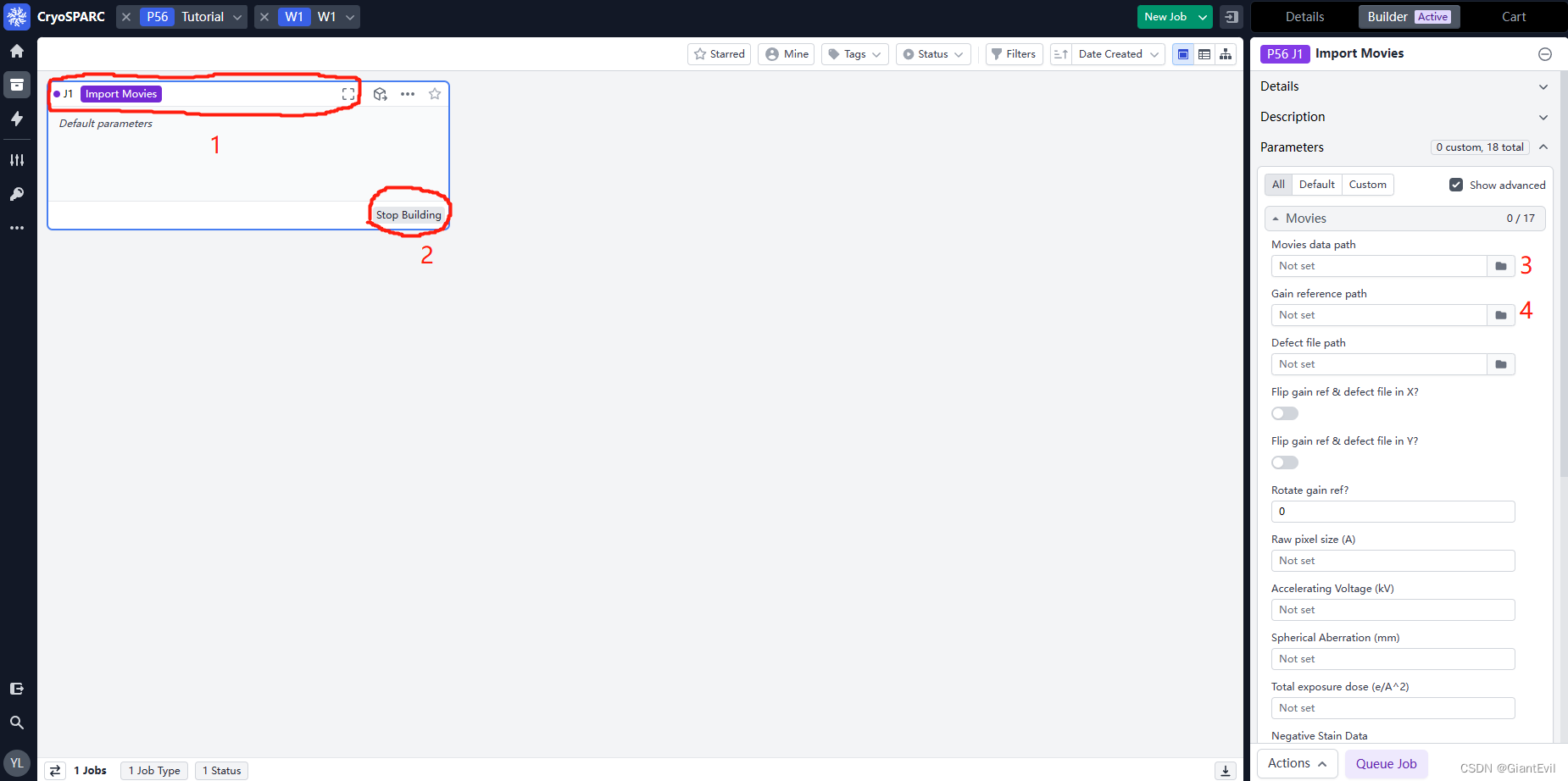
点击Import Movies。
1、点击这个区域可以放大Job看详细信息
2、如果想要暂停一下去做别的事,可以点击Stop Building。同时,这个图标会变成紫色的Build,再点一下就可以继续编辑。
3、输入Movies的路径
4、输入gain文件(gain.mrc)
这里如果收集回来的gain是.dm4格式的,用eman2软件的e2proc2d.py程序转化格式。
module load eman2/2.31
cd /path/to/gain
e2proc2d.py gain.dm4 gain.mrc
点击Movie data path右边的文件夹,找到自己的原始数据位置,选择其中一张照片,然后在路径里改成*.tiff或者*.eer(*代表该文件夹下全部文件,为了防止一起导入当前路径下有gain或者其它文件,写*.tiff保证导入的都是以tiff后缀的Movies)。点击Select。这时候再点击Movie data path右边的文件夹图标,我们会发现当前目录下全部Movies都被选中了。

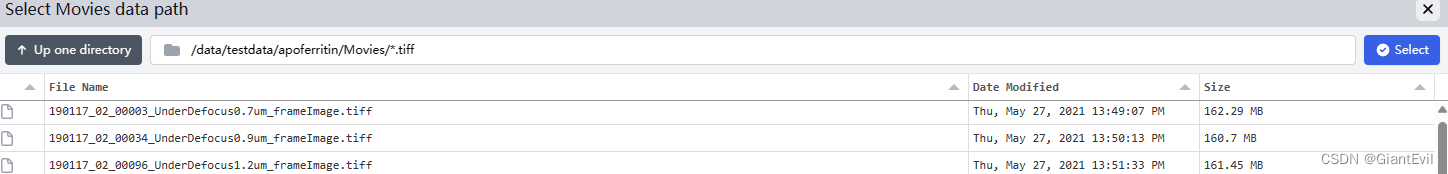
同样的,点击Gain reference path右边的文件夹图标,选中gain.mrc

然后将电镜收集参数填完(这些参数收集数据的时候记下来)。
Rotate gain ref? 一般gain flip和rotate都不用管,这套tutorial的数据需要旋转90度。输入1代表选择一次90度。
Raw pixel size (A) 像素大小
Accelerating Voltage (kV) 加速电压
Spherical Aberration (mm) 球差系数
Total exposure dose (e/A^2) 总电子剂量
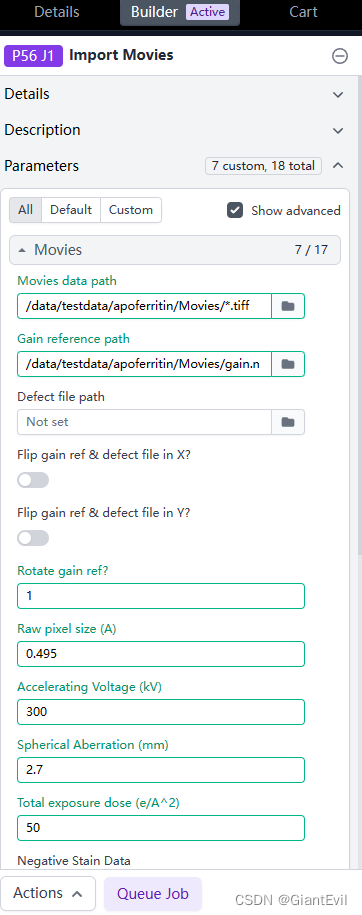
可以选择CPU的个数,这个只是导入文件,一般默认4个够用了。Skip Header Check可以选择性地勾选。默认不勾的话会检查每一张图片的Header看看有无异常。如果赶时间或者确认你的图片没啥问题,可以跳过。点击最下方Queue Job然后Queue。
可以看到这就是我们的原始图像。

第四步 漂移校正(MotionCorrection)
点击Builder,点击Patch Motion Correction。(或者使用Zhang et al开发的MotionCor2)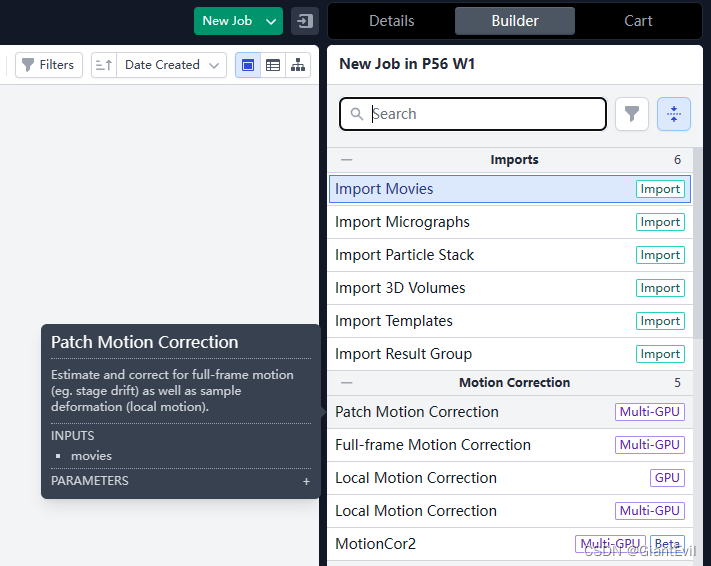
然后点击J1-Import Movies左上角,这时显示J1的详细信息,右边是J2的Builder信息。点住J1右侧Output Groups输出项中的Import Movies,此时右侧的Input框会变绿,拖住Import Movies并放入Input。即用J1的Output作为J2的Input。这种操作在cryoSPARC中十分常用,下文不再重复。
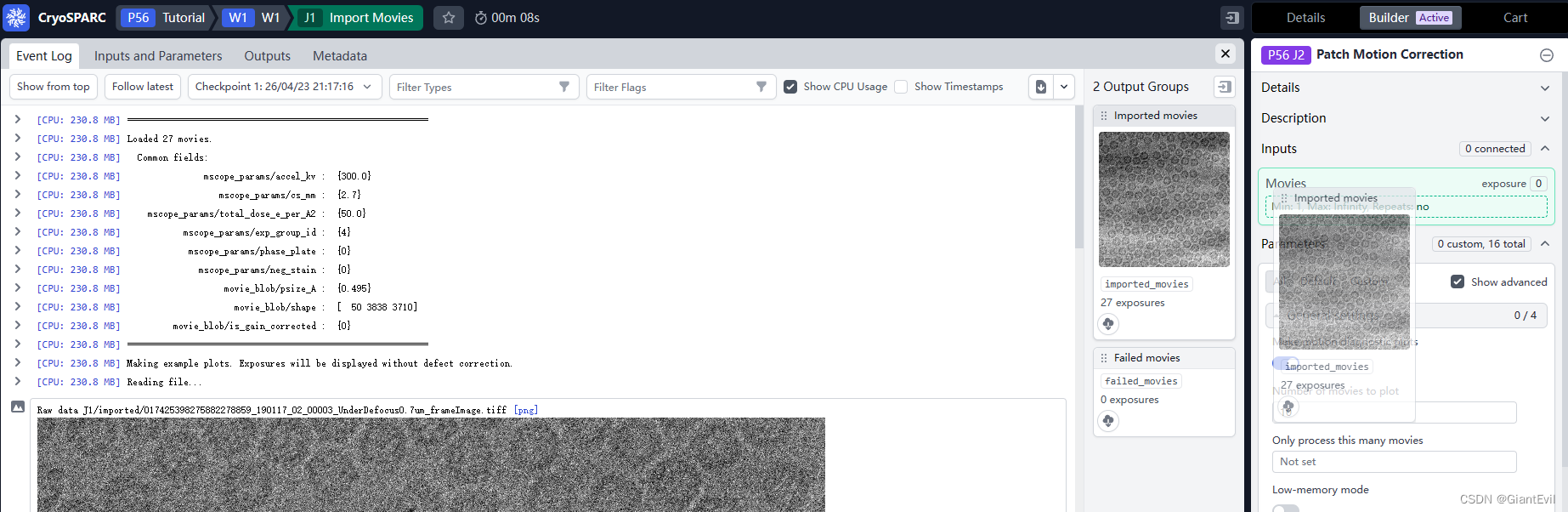
在Parameters中将Output F-crop factor选为1/2。这个为bin2。由于数据是以Superresolution模式收集的,但是实际上并没有多好的效果,只是增大计算量,因此bin2后可以加快运行速度。
点击Queue Job,选择CPU、GPU。选择GPU前在shell用nvidia-smi命令查看是否有人占用GPU,不要一起用一块GPU,容易内存溢出报错等等。点击Queue运行。
nvidia-smi

第五步 计算CTF(CTF estimation)
CTF叫做衬度传递函数(contrast transfer function)是电镜拍摄图片的特征,照片要作ctf修正。
点击右侧Builder,点击创建Patch CTF Estimation或者引用CTFFIND4算法的CTF Estimation。
类似于上一步,点开MotionCorrection的结果(J2),将其Output-Micrograph作为Input拖入Patch CTF Estimation的Inputs框内。参数默认就行,点击Queue,选择CPU/GPU后运行。
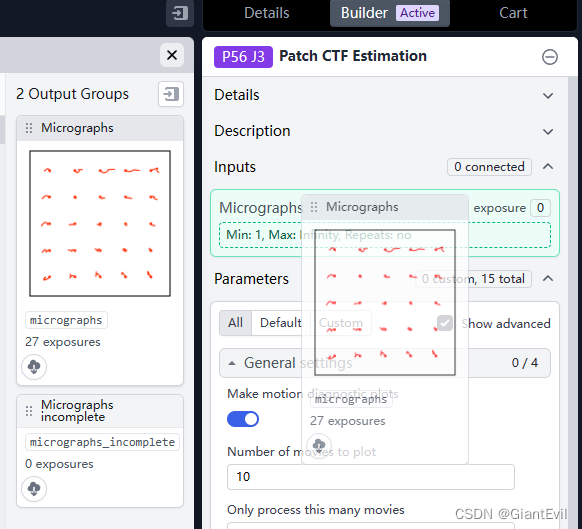
第六步 筛选图片
在数据收集过程中可能会有一些冰污染严重、像散较高等等问题的照片,可以通过Manually Curate Exposures Job来进行人工筛选剔除。
注意,任何有着红色Interactive的标签的Job意味着需要交互,否则Job不会自动完成。
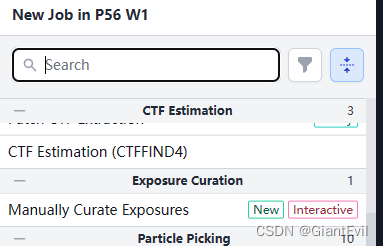
将CTF estimated的Micrographs作为Input Exposures。Paticles可以不管,它写着Min:0就是允许没有Input。点击Queue。
上面提到的Interactive标签,Queue后就会显示成这样“waiting”的粉红色。点击J4打开。
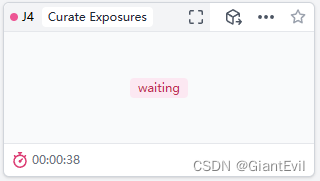
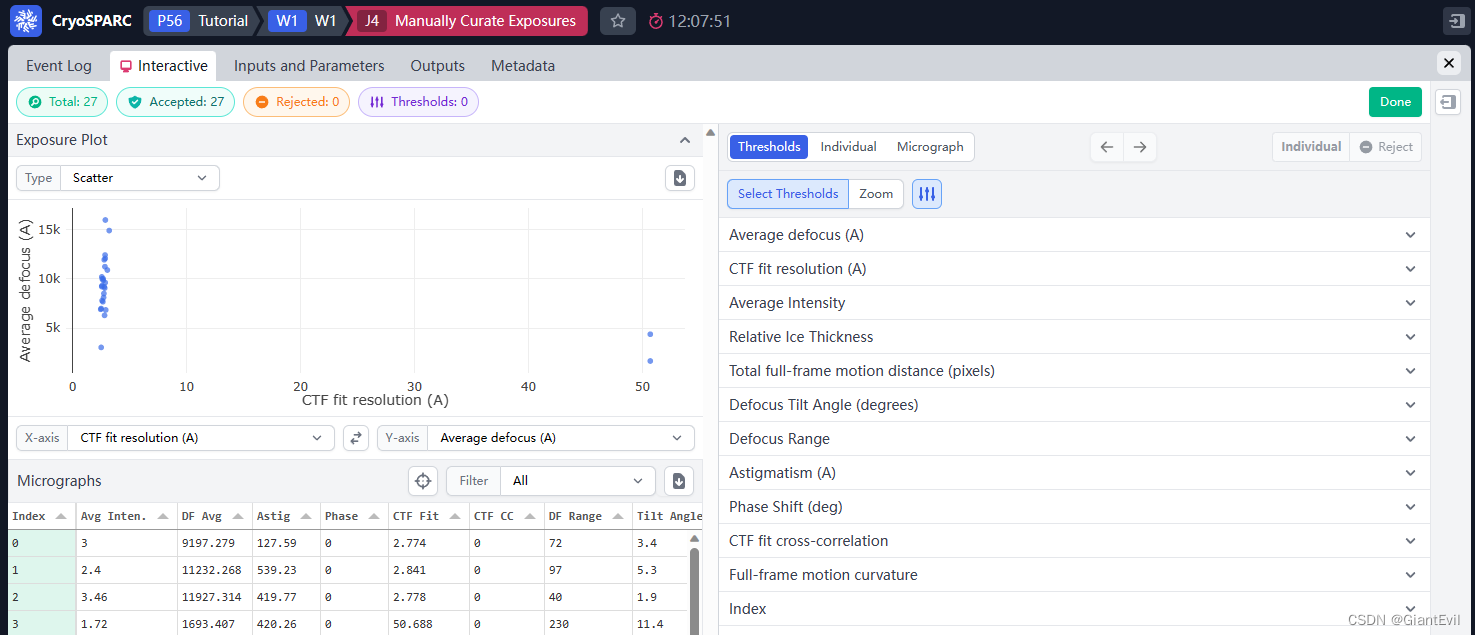
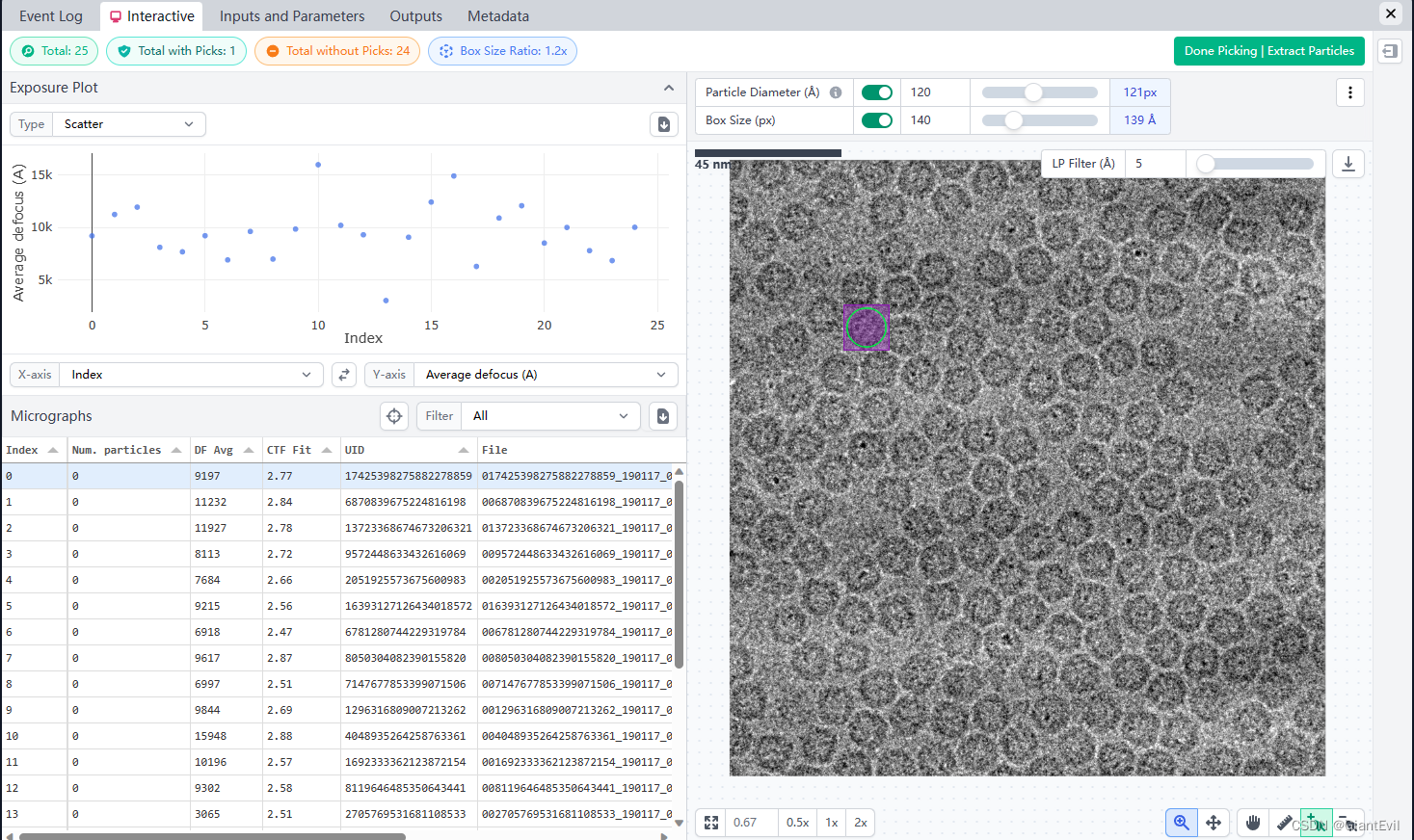
打开后会显示这样的交互界面。右边有三个选项卡Thresholds-Individual-Micrograph。Thresholds下可以通过调整阈值来批量选择照片。Individual会显示单张照片的参数,Micrograph会显示照片,这两个都需要选择某张照片来显示。
一般电镜照片都是数千上万张,所以大部分时候会用Thresholds快速筛选明显有问题的照片。比如这里把在CTF fit resolution里看到两个明显离群的照片,值异常大。鼠标放到上面显示照片就能看出明显有问题。
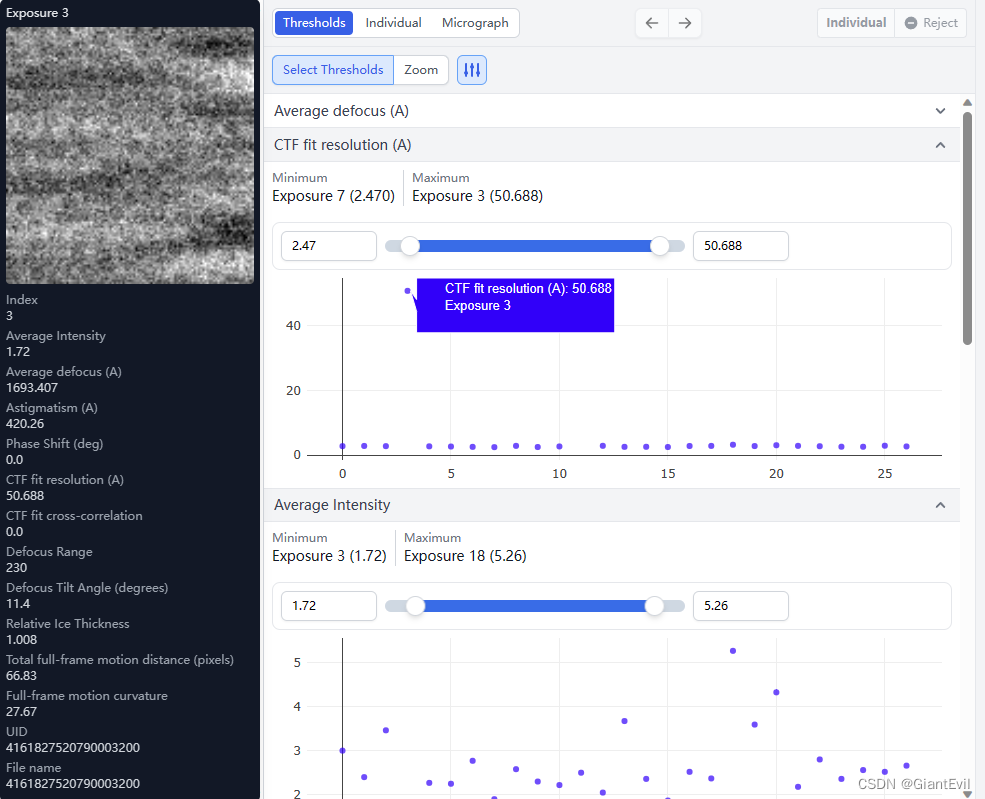
一种方法是在左侧选中这张照片(3号),右上角可以Reject舍弃掉。
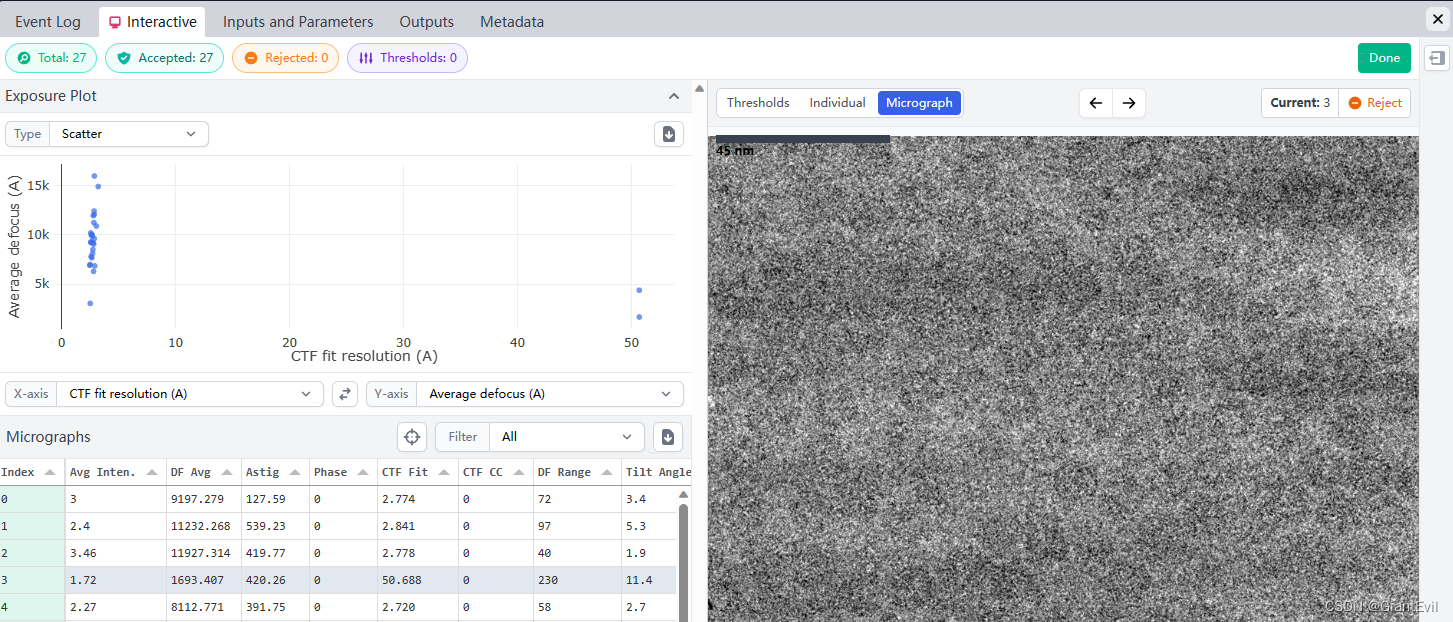
另一种方法是在Thresholds里选中CTF fit resolution,设置阈值的范围在2A-6A然后点击Set Thresholds,这样两张异常的照片就都Excluded了。
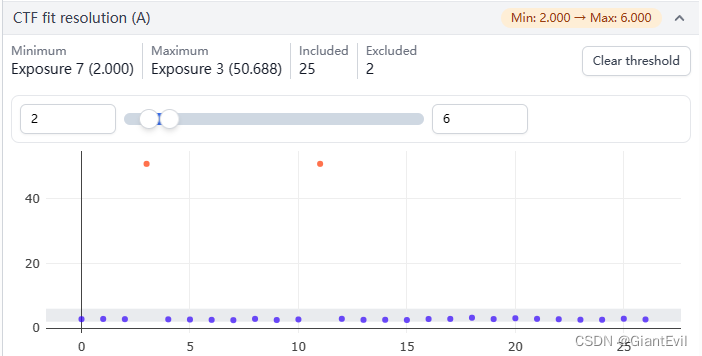
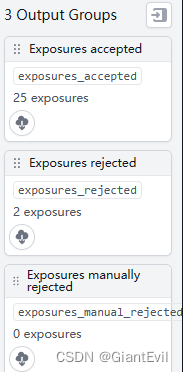
点击右上角Done完成。右侧Output就可以看到25张照片accepted,2张rejected。
第七步 挑选颗粒(Particle picking)
挑选颗粒是选出颗粒的坐标,一般常用的有Manual Picker手动挑选,Blob Picker通过给定颗粒大小自动挑选,Template Picker以给定二维模板来挑选颗粒。颗粒的挑加上二维分类在一些具有挑战性的蛋白或者照片中是困难且需要技巧的,往往这一步决定了后续三维重构、分类、细化的质量和结果。cryoSPARC中还有一些别的颗粒挑选的方法,比如通过深度学习训练等新的方法,可以探索。
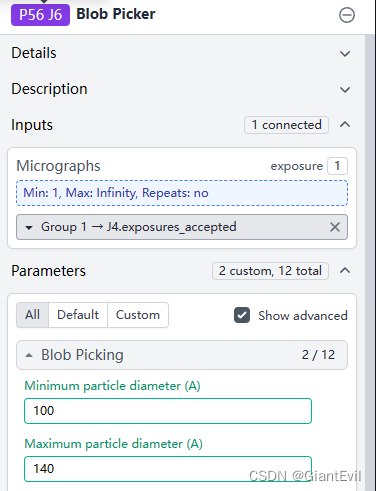
Blob Picker非常简单,直接给颗粒大小就自动挑选。选择Builder-Blob Picker。将Curate Exposure里accepted作为Input。并且给出颗粒的大小范围。你的颗粒不规则就要考虑到不同方向的投影可能大小不同。Queue Job选择CPU/GPU。
尝试一下Manual Picker。在自动挑选不太理想的情况下,我们自己是最熟悉自己的颗粒形状的,往往需要通过我们的经验来先挑选一部分高质量的颗粒,以此作为模板来进行Template Picker。选中Manual Picker,把上一步Curate Exposure的accepted作为输入。Queue Job。
鼠标左键点击可以挑选颗粒,右下角可以把左键功能改为删减颗粒。上方显示Particle Diameter是绿色的圈大小,BoxSize是紫色的框大小,这里只是作为后续Job参数的参考以及看起来更舒服,不会造成什么影响。这是铁蛋白的数据,铁蛋白直径约为120A。
可以在左侧选不同照片进行颗粒挑选。根据需求选择几十上百个颗粒,完成后点击右上角Done即可。
第八步 颗粒提取(Paricle extraction)
新建Job,Builder-Extract From Micographs。有GPU可以用GPU或者不用就用CPU。
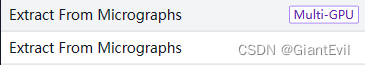
ParticlePicking的Job里有两个Output:Micrograph和Particle。分别作为Extract From Micographs的Input Micrograph和Particle。当然Input Micrograph要是想选择Curated exposures也行,ParticlePicking里的Output Micrograph只是它的一个子集。
然后修改Extraction box size。这个参数代表提取颗粒的box大小,一般需要比你的颗粒大20%-100%。注意单位,用你的pixel size参数来进行pix和A之间的转化。Queue Job。
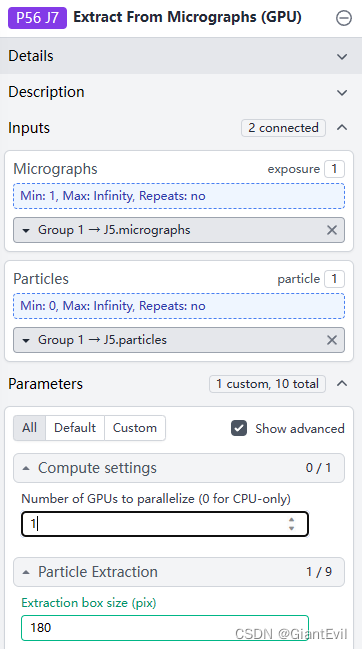
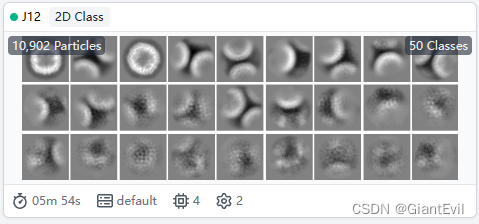
第九步 二维分类(2D Classification)
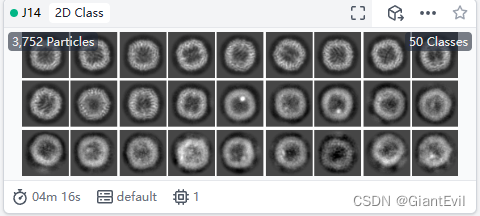
在Particle Extraction之后,每一个颗粒都被抠出来当成一个单独的micrograph。选中Builder-2D Classification,将Particle Extraction的Output-Particle Extract作为Input输入。Number of 2D classes是要分的类数目。默认是50。如果颗粒太少它会自动减少类,这里象征性地改成20。如果颗粒多的话可以增加分类的数目来获得更好的效果,但是也会增加计算时间。Queue Job。

默认迭代次数是20轮,一般足以应付大部分数据至收敛了。
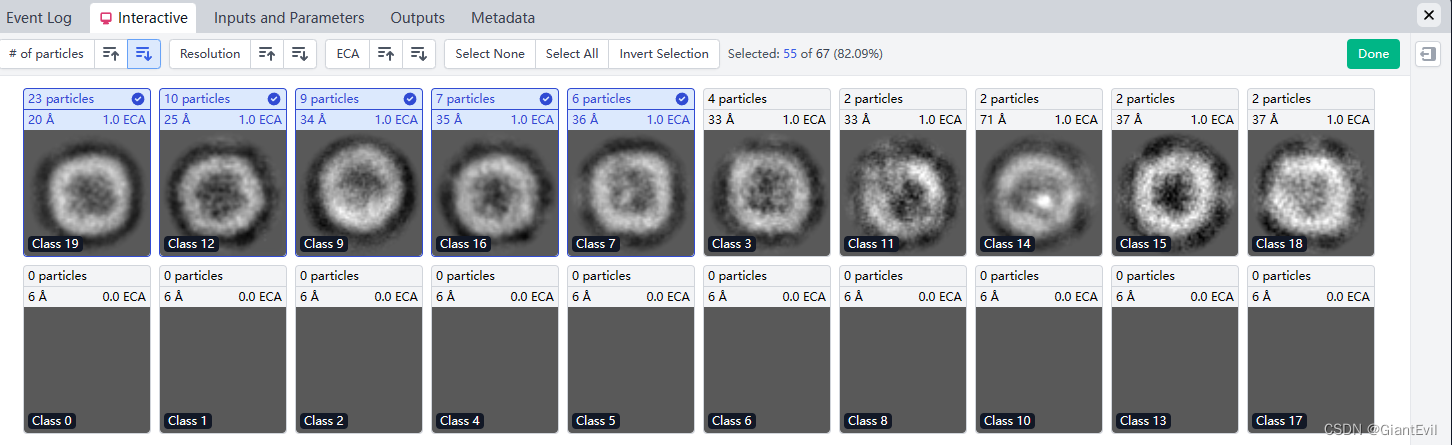
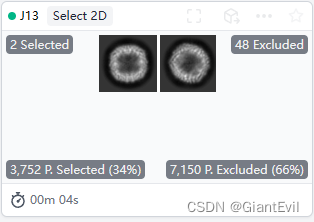
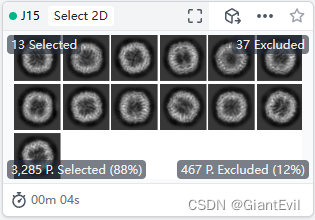
Builder-Select 2D Classes,将2D Classification的两个Output作为Input输入。Queue Job。这也是个Interactive Job。
选择自己想要的二维平均,点击Done。
第十步 用模板挑选颗粒
我们有了二维的模板,就可以以此进行Template Picker了。点击Builder-Template Picker。将Select 2D Job里Output-Templated Selected作为Template Input,将Curate Exposure-accepted作为Micrograph Input。然后输入一个Particle Diameter。Queue Job。
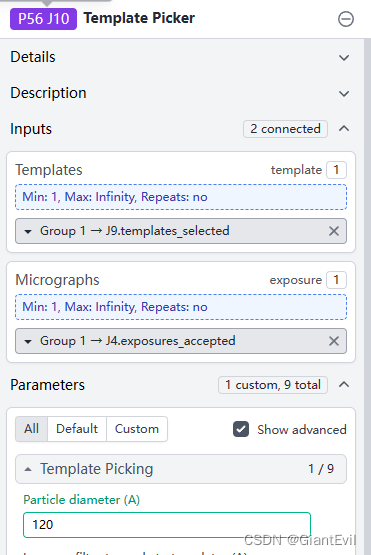
然后和之前步骤类似,Extract Particles,2D Classification,Select 2D Classes。将用模板挑的颗粒进行二维分类。
在实际的数据处理过程中,往往一轮二维分类无法达到好的效果。挑选好的二维类,然后再进行二维分类。往复循环直到达到满意的效果。
除了这种手动挑选颗粒生成2D template,还有另一种方法生成模板。Builder-Import Volume可以导入已有的3D map。Builder-Create Template可以将3D Volume进行2D投影,从而生成2D Template。
第十一步 生成3D模型(Ab-initio)
Ab-initio可以根据颗粒从零开始生成三维模型,后续再用这个模型可以用来三维分类或者细化。Builder-Ab-Initio Reconstruction。将Select 2D Classes作为Particle stacks输入。Number of Ab-Initio classes可以选择想要分的类,在早期版本的cryoSPARC中还没有类似relion的3D Classification,这是三维分类的方法之一,只是精度稍低,可以分出构象差别较大的类,也可以用来剔除二维分类中没有剔除的杂颗粒。
这里分个2类。铁蛋白是高度对称的球体,我们给个对称性O。



这一步可能耗时较长,且Ab-Initio只支持单卡GPU,分的类越多耗时越久。
第十二步 三维分类(3D Classification)
v4.0以前的cryoSPARC还是用的Heterogeneous refinement来进行颗粒三维分类,需要有初始不同三维模型。v4.0之后有了新的3D Classification的Job,类似于relion里的3D Classification。
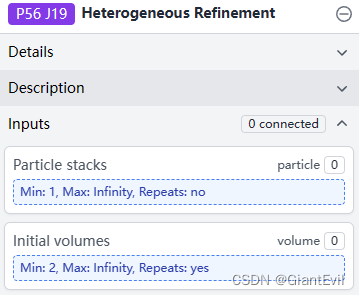
Heterogeneous Refinement点击后输入Particle和Volume,至少输入两个Volume。
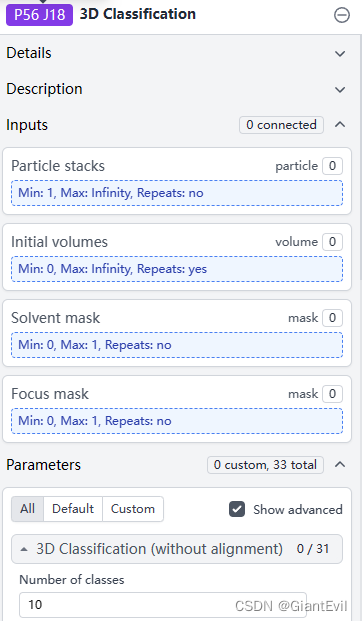
3D Classification点击后必需输入Particle stack,Volume、Mask等都是可选的。
第十三步 细化map(3D refinement)
无论是三维分类还是Heterogeneous refinement,都有限制最高分辨率。3D refinement能够将分辨率进一步提升,如果数据质量足够好,是可以到达3A以内近原子分辨率的。
常用的有两个Job,一个是经典的Homogeneous refinement,一个是Non-uniform refinement,号称是可以有更好的细化效果,尤其是对膜蛋白。具体的参考cryoSPARC的官方文档和论文,感兴趣的去研究下算法。

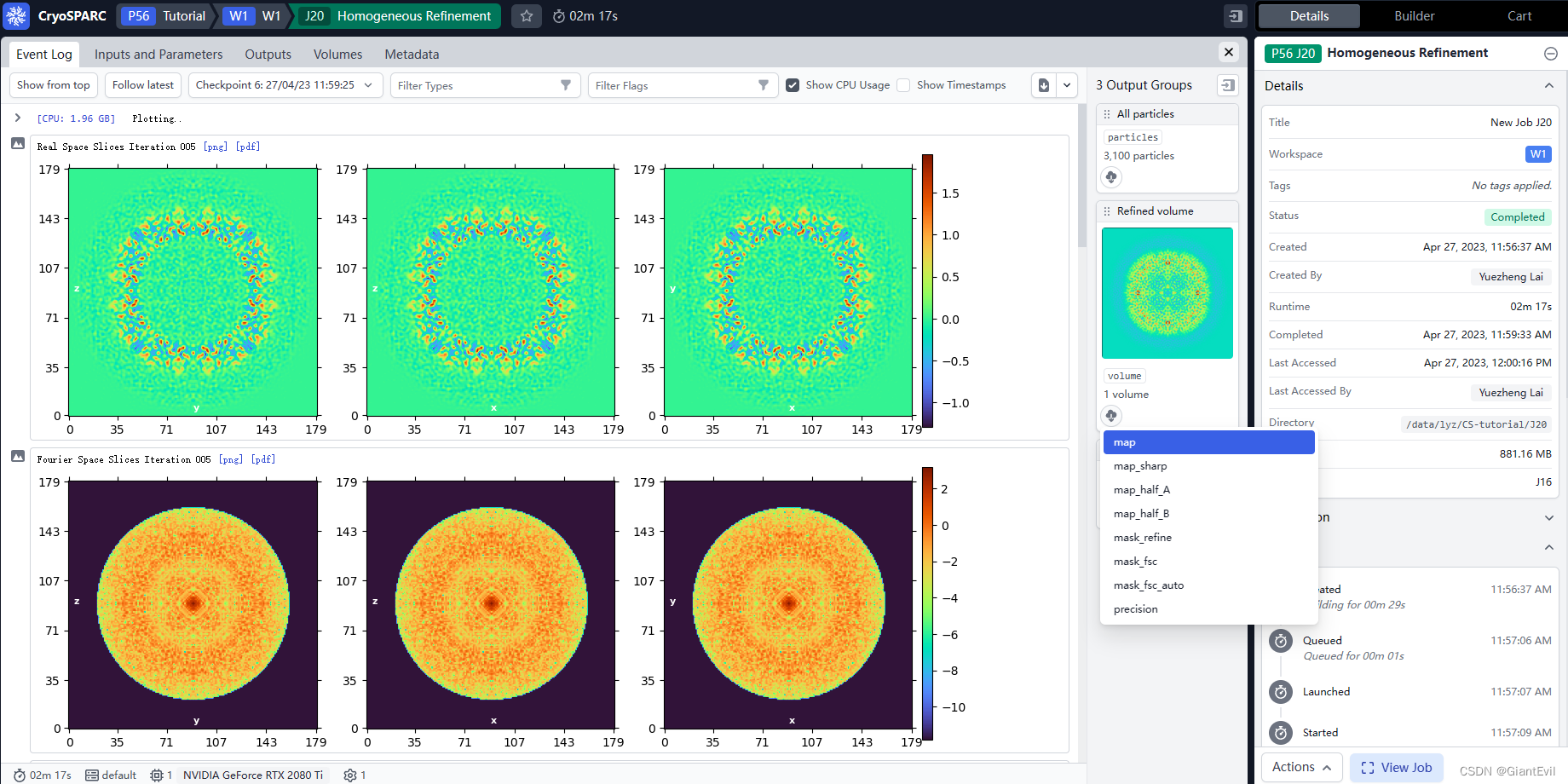
这里选择Homogeneous refinement,一般而言速度会比Non-uniform refinement快一点。选择三维分类获得的Particle stack颗粒集和对应的Volume map作为输入。同样给个O对称性。
由于铁蛋白本身是个标准蛋白,又给了高对称性,所以很容易就到了2.5A的高分辨率。
点击Output里一个云和一个箭头的图标可以下载结果。
map就是获得的密度图
map_sharp是sharpen后的map,在job的详细信息Guinier Plot里可以看到map sharpen BFactor
map_halfA/B是half map。软件会把颗粒随机分成两部分计算,看它们拟合度来判断分辨率,就是详细信息里的GSFSC(Golden Standard Fourier Shell Correlation) 。常见的有用0.143或者0.5作为阈值衡量分辨率。现在上传pdb数据库已经必需上传halfmap了。
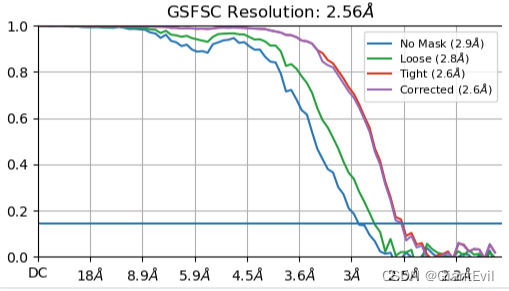
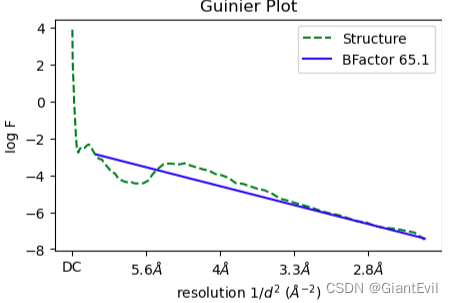
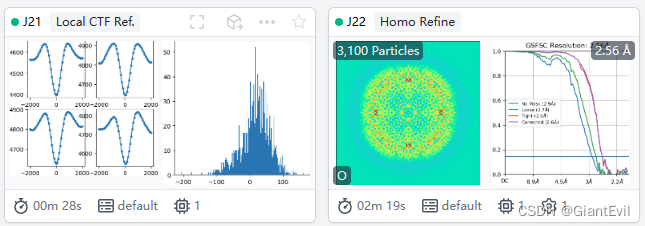
** 第十四步 局部CTF细化(Local CTF Refinement)**
Local CTF Refinement会对每一个颗粒的局部CTF做一个校正,使得结果更加精准,有可能进一步提高分辨率。将Refinement的Particle和Volume输入Local CTF Refinement的Job。然后将Local CTF Refinement输出的校正过的Particle和前面的Volume再做一次Refinement。这个并没有提高分辨率,我们这里的分辨率可能以及收敛到极限了。
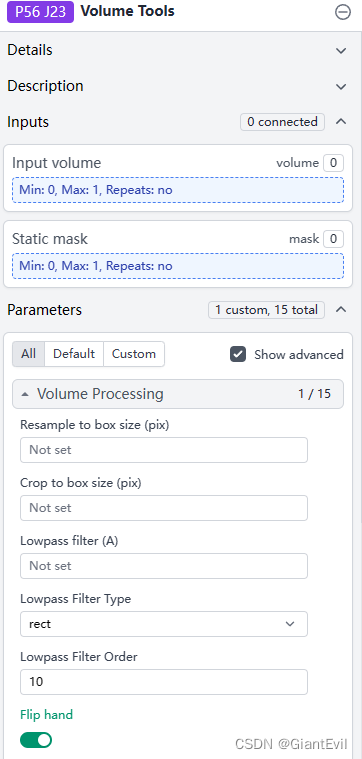
第十五步 一些其它Jobs
在运算过程中,可能会出现手性翻转的现象,Builder-Volume Tools的Job选Flip hands可以将Volume的手性翻转。
发表文章中可能需要3DFSC histogram的柱形图,就是下面这个图,Builder-ThreeDFSC的job可以生成。

发表文章中可能需要Local resolution的图片,Builder-Local resolution estimation的Job可以生成Local resolution文件(选择那个叫做map_locres的下载),在ChimeraX等软件中可以展示。
下载后同时打开map和map_locres。上方工具栏Tools-Volume Data-Surface Color。选择by volume data value。Color surface选择map,using map选择locres。颜色输入对应分辨率。点击color。具体的ChimeraX软件操作详见ChimeraX的操作指南,这里不细说。


版权归原作者 GiantEvil 所有, 如有侵权,请联系我们删除。