图像分割是医学图像分析中最重要的任务之一,在许多临床应用中往往是第一步也是最关键的一步。在脑MRI分析中,图像分割通常用于测量和可视化解剖结构,分析大脑变化,描绘病理区域以及手术计划和图像引导干预,分割是大多数形态学分析的先决条件。
本文我们将介绍如何使用QuickNAT对人脑的图像进行分割。使用MONAI, PyTorch和用于数据可视化和计算的常见Python库,如NumPy, TorchIO和matplotlib。
本文将主要设计以下几个方面:
- 设置数据集和探索数据
- 处理和准备数据集适当的模型训练
- 创建一个训练循环
- 评估模型并分析结果
完整的代码会在本文最后提供。
设置数据目录
使用MONAI的第一步是设置MONAI_DATA_DIRECTORY环境变量指定目录,如果未指定将使用临时目录。
directory=os.environ.get("MONAI_DATA_DIRECTORY")
root_dir=tempfile.mkdtemp() ifdirectoryisNoneelsedirectory
print(root_dir)
设置数据集
将CNN模型扩展到大脑分割的主要挑战之一是人工注释的训练数据的有限性。作者引入了一种新的训练策略,利用没有手动标签的大型数据集和有手动标签的小型数据集。
首先,使用现有的软件工具(例如FreeSurfer)从大型未标记数据集中获得自动生成的分割,然后使用这些工具对网络进行预训练。在第二步中,使用更小的手动注释数据[2]对网络进行微调。
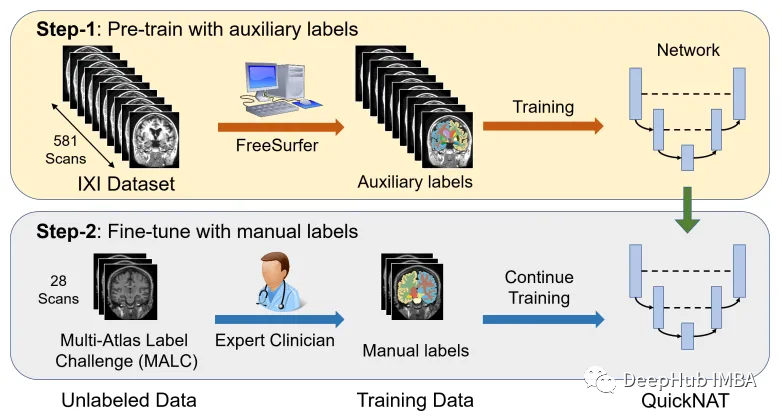
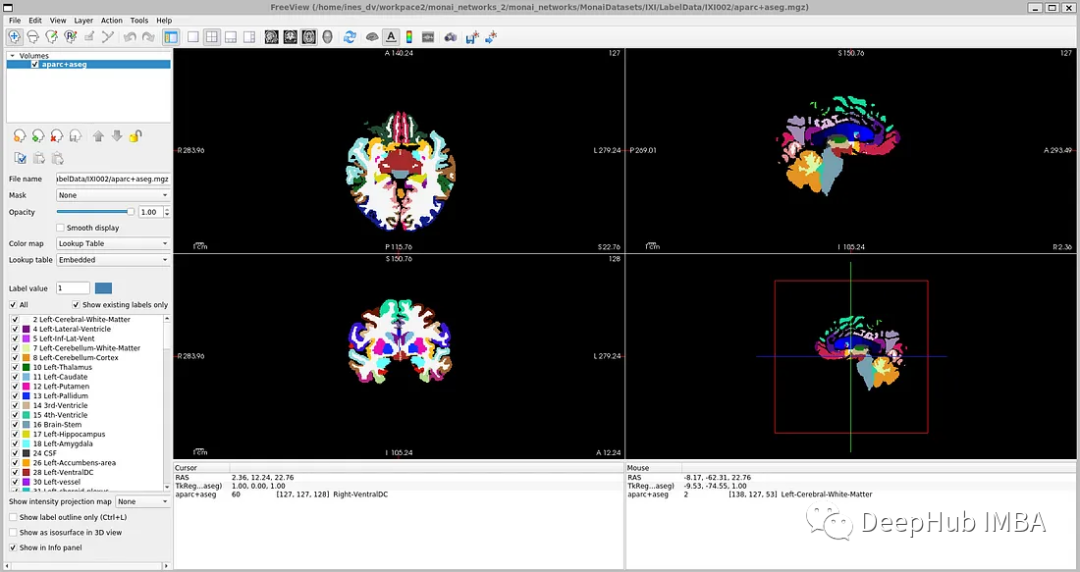
IXI数据集由581个健康受试者的未标记MRI T1扫描组成。这些数据是从伦敦3家不同的医院收集来的。使用该数据集的主要缺点是标签不是公开可用的,因此为了遵循与研究论文中相同的方法,本文将使用FreeSurfer为这些MRI T1扫描生成分割。
FreeSurfer是一个用于分析和可视化结构的软件包。下载和安装说明可以在这里找到。可以直接使用了“recon-all”命令来执行所有皮层重建过程。
尽管FreeSurfer是一个非常有用的工具,可以利用大量未标记的数据,并以监督的方式训练网络,但是扫描生成这些标签需要长达5个小时,所以我们这里直接使用OASIS数据集来训练模型,OASIS数据集是一个较小的数据集,具有公开可用的手动注释。
OASIS是一个向科学界免费提供大脑神经成像数据集的项目。OASIS-1是由39个受试者的横断面组成的数据集,获取方式如下:
resource="https://download.nrg.wustl.edu/data/oasis_cross-sectional_disc1.tar.gz"
md5="c83e216ef8654a7cc9e2a30a4cdbe0cc"
compressed_file=os.path.join(root_dir, "oasis_cross-sectional_disc1.tar.gz")
data_dir=os.path.join(root_dir, "Oasis_Data")
ifnotos.path.exists(data_dir):
download_and_extract(resource, compressed_file, data_dir, md5)
数据探索
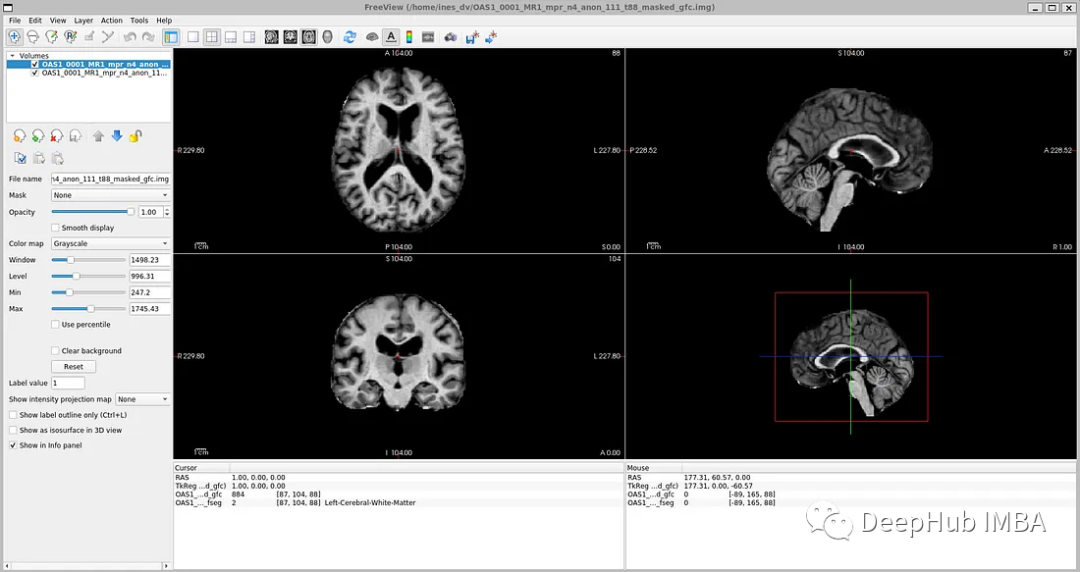
如果你打开' oasis_crosssectional_disc1 .tar.gz ',你会发现每个主题都有不同的文件夹。例如,对于主题OAS1_0001_MR1,是这样的:
镜像数据文件路径:disc1\OAS1_0001_MR1\PROCESSED\MPRAGE\T88_111\ oas1_0001_mr1_mpr_n4_anon_111_t88_masked_ggc .img
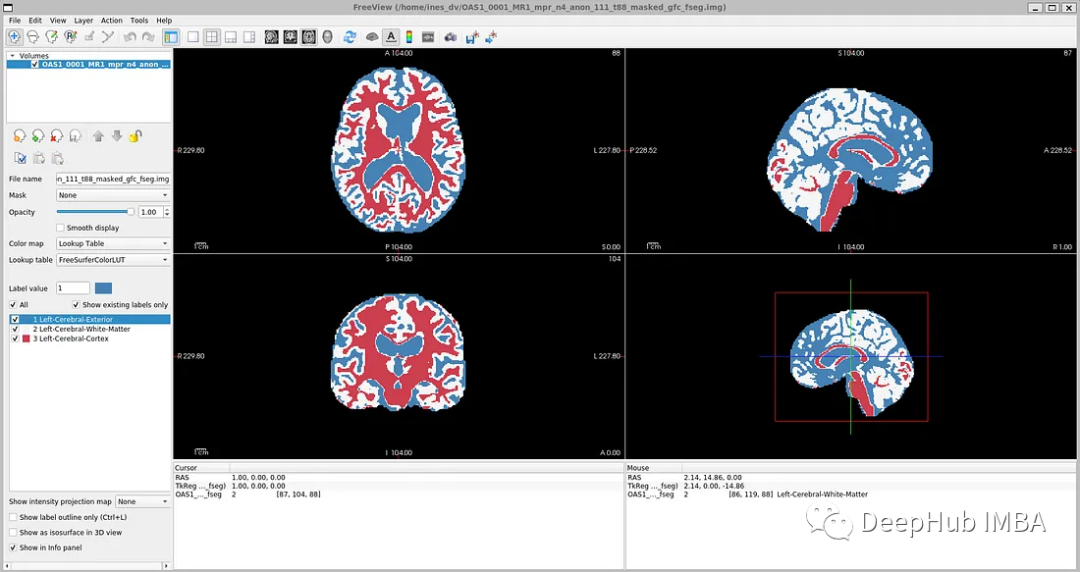
标签文件:disc1\OAS1_0001_MR1\FSL_SEG\OAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc_fseg.img
数据加载和预处理
下载数据集并将其提取到临时目录后,需要对其进行重构,我们希望我们的目录看起来像这样:
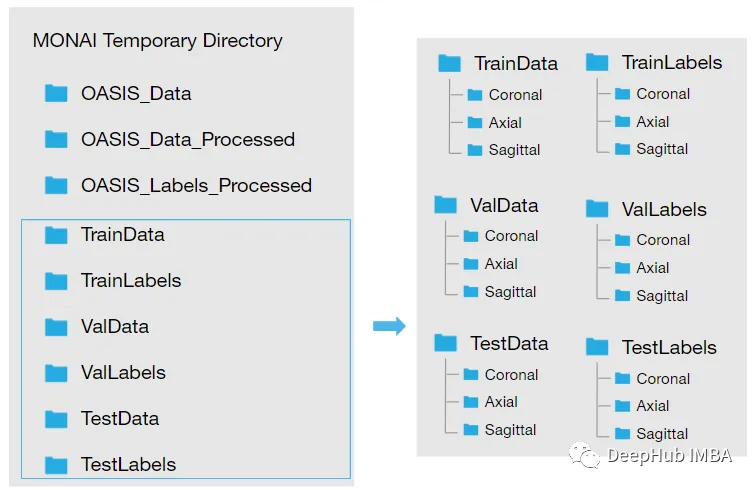
所以需要按照下面的步骤加载数据:
将。img文件转换为。nii文件并保存到新文件夹中:创建两个新文件夹。Oasis_Data_Processed包括每个受试者的处理过的MRI T1扫描,Oasis_Labels_Processed包括相应的标签。
new_path_data=root_dir+'/Oasis_Data_Processed/'
ifnotos.path.exists(new_path_data):
os.makedirs(new_path_data)
new_path_labels=root_dir+'/Oasis_Labels_Processed/'
ifnotos.path.exists(new_path_labels):
os.makedirs(new_path_labels)
然后就是对其进行操作:
foriin [xforxinrange(1, 43) ifx!=8andx!=24andx!=36]:
ifi<7ori==9:
filename=root_dir+'/Oasis_Data/disc1/OAS1_000'+str(i) +'_MR1/PROCESSED/MPRAGE/T88_111/OAS1_000'+str(i) +'_MR1_mpr_n4_anon_111_t88_masked_gfc.img'
elifi==7:
filename=root_dir+'/Oasis_Data/disc1/OAS1_000'+str(i) +'_MR1/PROCESSED/MPRAGE/T88_111/OAS1_000'+str(i) +'_MR1_mpr_n3_anon_111_t88_masked_gfc.img'
elifi==15ori==16ori==20ori==24ori==26ori==34ori==38ori==39:
filename=root_dir+'/Oasis_Data/disc1/OAS1_00'+str(i) +'_MR1/PROCESSED/MPRAGE/T88_111/OAS1_00'+str(i) +'_MR1_mpr_n3_anon_111_t88_masked_gfc.img'
else:
filename=root_dir+'/Oasis_Data/disc1/OAS1_00'+str(i) +'_MR1/PROCESSED/MPRAGE/T88_111/OAS1_00'+str(i) +'_MR1_mpr_n4_anon_111_t88_masked_gfc.img'
img=nib.load(filename)
nib.save(img, filename.replace('.img', '.nii'))
i=i+1
具体代码就不再粘贴了,有兴趣的看看最后的完整代码。下一步就是读取图像和标签文件名
image_files=sorted(glob(os.path.join(root_dir+'/Oasis_Data_Processed', '*.nii')))
label_files=sorted(glob(os.path.join(root_dir+'/Oasis_Labels_Processed', '*.nii')))
files= [{'image': image_name, 'label': label_name} forimage_name, label_nameinzip(image_files, label_files)]
为了可视化带有相应标签的图像,可以使用TorchIO,这是一个Python库,用于深度学习中多维医学图像的加载、预处理、增强和采样。
image_filename=root_dir+'/Oasis_Data_Processed/OAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc.nii'
label_filename=root_dir+'/Oasis_Labels_Processed/OAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc_fseg.nii'
subject=torchio.Subject(image=torchio.ScalarImage(image_filename), label=torchio.LabelMap(label_filename))
subject.plot()
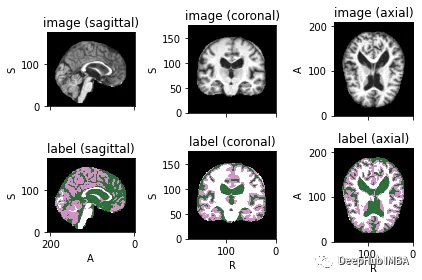
下面就是将数据分成3部分——训练、验证和测试。将数据分成三个不同的类别的目的是建立一个可靠的机器学习模型,避免过拟合。
我们将整个数据集分成三个部分:
Train: 80%,Validation: 10%,Test: 10%
train_inds, val_inds, test_inds=partition_dataset(data=np.arange(len(files)), ratios= [8, 1, 1], shuffle=True)
train= [files[i] foriinsorted(train_inds)]
val= [files[i] foriinsorted(val_inds)]
test= [files[i] foriinsorted(test_inds)]
print(f"Training count: {len(train)}, Validation count: {len(val)}, Test count: {len(test)}")
因为模型需要的是二维切片,所以将每个切片保存在不同的文件夹中,如下图所示。这两个代码单元将训练集的每个MRI体积的切片保存为“.png”格式。
Savecoronalslicesfortrainingimages
dir=root_dir+'/TrainData'
os.makedirs(os.path.join(dir, "Coronal"))
path=root_dir+'/TrainData/Coronal/'
forfileinsorted(glob(os.path.join(root_dir+'/TrainData', '*.nii'))):
image=torchio.ScalarImage(file)
data=image.data
filename=os.path.basename(file)
filename=os.path.splitext(filename)
foriinrange(0, 208):
slice=data[0, :, i]
array=slice.numpy()
data_dir=root_dir+'/TrainData/Coronal/'+filename[0] +'_slice'+str(i) +'.png'
plt.imsave(fname=data_dir, arr=array, format='png', cmap=plt.cm.gray)
同理,下面是保存标签:
dir=root_dir+'/TrainLabels'
os.makedirs(os.path.join(dir, "Coronal"))
path=root_dir+'/TrainLabels/Coronal/'
forfileinsorted(glob(os.path.join(root_dir+'/TrainLabels', '*.nii'))):
label=torchio.LabelMap(file)
data=label.data
filename=os.path.basename(file)
filename=os.path.splitext(filename)
foriinrange(0, 208):
slice=data[0, :, i]
array=slice.numpy()
data_dir=root_dir+'/TrainLabels/Coronal/'+filename[0] +'_slice'+str(i) +'.png'
plt.imsave(fname=data_dir, arr=array, format='png')
为训练和验证定义图像的变换处理
在本例中,我们将使用Dictionary Transforms,其中数据是Python字典。
train_images_coronal= []
forfileinsorted(glob(os.path.join(root_dir+'/TrainData/Coronal', '*.png'))):
train_images_coronal.append(file)
train_images_coronal=natsort.natsorted(train_images_coronal)
train_labels_coronal= []
forfileinsorted(glob(os.path.join(root_dir+'/TrainLabels/Coronal', '*.png'))):
train_labels_coronal.append(file)
train_labels_coronal=natsort.natsorted(train_labels_coronal)
val_images_coronal= []
forfileinsorted(glob(os.path.join(root_dir+'/ValData/Coronal', '*.png'))):
val_images_coronal.append(file)
val_images_coronal=natsort.natsorted(val_images_coronal)
val_labels_coronal= []
forfileinsorted(glob(os.path.join(root_dir+'/ValLabels/Coronal', '*.png'))):
val_labels_coronal.append(file)
val_labels_coronal=natsort.natsorted(val_labels_coronal)
train_files_coronal= [{'image': image_name, 'label': label_name} forimage_name, label_nameinzip(train_images_coronal, train_labels_coronal)]
val_files_coronal= [{'image': image_name, 'label': label_name} forimage_name, label_nameinzip(val_images_coronal, val_labels_coronal)]
现在我们将应用以下变换:
LoadImaged:加载图像数据和元数据。我们使用' PILReader '来加载图像和标签文件。ensure_channel_first设置为True,将图像数组形状转换为通道优先。
Rotate90d:我们将图像和标签旋转90度,因为当我们下载它们时,它们方向是不正确的。
ToTensord:将输入的图像和标签转换为张量。
NormalizeIntensityd:对输入进行规范化。
train_transforms=Compose(
[
LoadImaged(keys= ['image', 'label'], reader=PILReader(converter=lambdaimage: image.convert("L")), ensure_channel_first=True),
Rotate90d(keys= ['image', 'label'], k=2),
ToTensord(keys= ['image', 'label']),
NormalizeIntensityd(keys= ['image'])
]
)
val_transforms=Compose(
[
LoadImaged(keys= ['image', 'label'], reader=PILReader(converter=lambdaimage: image.convert("L")), ensure_channel_first=True),
Rotate90d(keys= ['image', 'label'], k=2),
ToTensord(keys= ['image', 'label']),
NormalizeIntensityd(keys= ['image'])
]
)
MaskColorMap将我们定义了一个新的转换,将相应的像素值以一种格式映射为多个标签。这种转换在语义分割中是必不可少的,因为我们必须为每个可能的类别提供二元特征。One-Hot Encoding将对应于原始类别的每个样本的特征赋值为1。
因为OASIS-1数据集只有3个大脑结构标签,对于更详细的分割,理想的情况是像他们在研究论文中那样对28个皮质结构进行注释。在OASIS-1下载说明中,可以找到使用FreeSurfer获得的更多大脑结构的标签。
所以本文将分割更多的神经解剖结构。我们要将模型的参数num_classes修改为相应的标签数量,以便模型的输出是具有N个通道的特征映射,等于num_classes。
为了简化本教程,我们将使用以下标签,比OASIS-1但是要比FreeSurfer的少:
- Label 0: Background
- Label 1: LeftCerebralExterior
- Label 2: LeftWhiteMatter
- Label 3: LeftCerebralCortex
所以MaskColorMap的代码如下:
class MaskColorMap(Enum):
Background = (30)
LeftCerebralExterior = (91)
LeftWhiteMatter = (137)
LeftCerebralCortex = (215)
数据集和数据加载
数据集和数据加载器从存储中提取数据,并将其分批发送给训练循环。这里我们使用monai.data.Dataset加载之前定义的训练和验证字典,并对输入数据应用相应的转换。dataloader用于将数据集加载到内存中。我们将为训练和验证以及每个视图定义一个数据集和数据加载器。
为了方便演示,我们使用通过使用torch.utils.data.Subset,在指定的索引处创建一个子集,只是用部分数据训练加快演示速度。
train_dataset_coronal=Dataset(data=train_files_coronal, transform=train_transforms)
train_loader_coronal=DataLoader(train_dataset_coronal, batch_size=1, shuffle=True)
val_dataset_coronal=Dataset(data=val_files_coronal, transform=val_transforms)
val_loader_coronal=DataLoader(val_dataset_coronal, batch_size=1, shuffle=False)
# We will use a subset of the dataset
subset_train=list(range(90, len(train_dataset_coronal), 120))
train_dataset_coronal_subset=torch.utils.data.Subset(train_dataset_coronal, subset_train)
train_loader_coronal_subset=DataLoader(train_dataset_coronal_subset, batch_size=1, shuffle=True)
subset_val=list(range(90, len(val_dataset_coronal), 50))
val_dataset_coronal_subset=torch.utils.data.Subset(val_dataset_coronal, subset_val)
val_loader_coronal_subset=DataLoader(val_dataset_coronal_subset, batch_size=1, shuffle=False)
定义模型
给定一组MRI脑扫描I = {I1,…In}及其对应的分割S = {S1,…Sn},我们想要学习一个函数fseg: I -> S。我们将这个函数表示为F-CNN模型,称为QuickNAT:
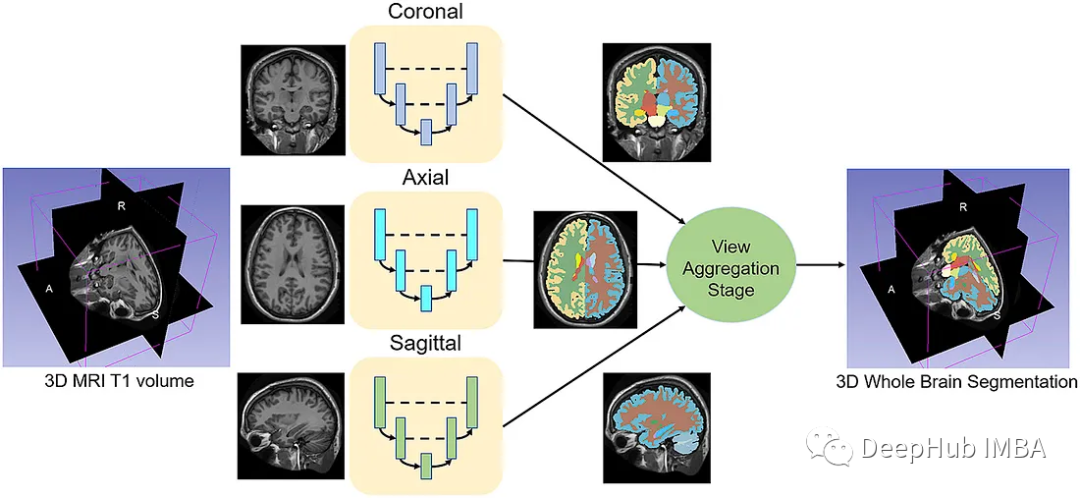
QuickNAT由三个二维f - cnn组成,分别在coronal, axial, sagittal视图上操作,然后通过聚合步骤推断最终的分割结果,该分割结果由三个网络的概率图组合而成。每个F-CNN都有一个编码器/解码器架构,其中有4个编码器和4个解码器,并由瓶颈层分隔。最后一层是带有softmax的分类器块。该架构还包括每个编码器/解码器块内的残差链接。
classQuickNat(nn.Module):
"""
A PyTorch implementation of QuickNAT
"""
def__init__(self, params):
"""
:param params: {'num_channels':1,
'num_filters':64,
'kernel_h':5,
'kernel_w':5,
'stride_conv':1,
'pool':2,
'stride_pool':2,
'num_classes':28
'se_block': False,
'drop_out':0.2}
"""
super(QuickNat, self).__init__()
# from monai.networks.blocks import squeeze_and_excitation as se
# self.cSE = ChannelSELayer(num_channels, reduction_ratio)
# self.encode1 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE)
# params["num_channels"] = params["num_filters"]
# self.encode2 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.encode3 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.encode4 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.bottleneck = sm.DenseBlock(params, se_block_type=se.SELayer.CSSE)
# params["num_channels"] = params["num_filters"] * 2
# self.decode1 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.decode2 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.decode3 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.decode4 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.encode1 = EncoderBlock(params, se_block_type=se.ChannelSELayer)
self.encode1=EncoderBlock(params, se_block_type=se.SELayer.CSSE)
params["num_channels"] =params["num_filters"]
self.encode2=EncoderBlock(params, se_block_type=se.SELayer.CSSE)
self.encode3=EncoderBlock(params, se_block_type=se.SELayer.CSSE)
self.encode4=EncoderBlock(params, se_block_type=se.SELayer.CSSE)
self.bottleneck=DenseBlock(params, se_block_type=se.SELayer.CSSE)
params["num_channels"] =params["num_filters"] *2
self.decode1=DecoderBlock(params, se_block_type=se.SELayer.CSSE)
self.decode2=DecoderBlock(params, se_block_type=se.SELayer.CSSE)
self.decode3=DecoderBlock(params, se_block_type=se.SELayer.CSSE)
self.decode4=DecoderBlock(params, se_block_type=se.SELayer.CSSE)
params["num_channels"] =params["num_filters"]
self.classifier=ClassifierBlock(params)
defforward(self, input):
"""
:param input: X
:return: probabiliy map
"""
e1, out1, ind1=self.encode1.forward(input)
e2, out2, ind2=self.encode2.forward(e1)
e3, out3, ind3=self.encode3.forward(e2)
e4, out4, ind4=self.encode4.forward(e3)
bn=self.bottleneck.forward(e4)
d4=self.decode4.forward(bn, out4, ind4)
d3=self.decode1.forward(d4, out3, ind3)
d2=self.decode2.forward(d3, out2, ind2)
d1=self.decode3.forward(d2, out1, ind1)
prob=self.classifier.forward(d1)
returnprob
defenable_test_dropout(self):
"""
Enables test time drop out for uncertainity
:return:
"""
attr_dict=self.__dict__["_modules"]
foriinrange(1, 5):
encode_block, decode_block= (
attr_dict["encode"+str(i)],
attr_dict["decode"+str(i)],
)
encode_block.drop_out=encode_block.drop_out.apply(nn.Module.train)
decode_block.drop_out=decode_block.drop_out.apply(nn.Module.train)
@property
defis_cuda(self):
"""
Check if model parameters are allocated on the GPU.
"""
returnnext(self.parameters()).is_cuda
defsave(self, path):
"""
Save model with its parameters to the given path. Conventionally the
path should end with '*.model'.
Inputs:
- path: path string
"""
print("Saving model... %s"%path)
torch.save(self.state_dict(), path)
defpredict(self, X, device=0, enable_dropout=False):
"""
Predicts the output after the model is trained.
Inputs:
- X: Volume to be predicted
"""
self.eval()
print("tensor size before transformation", X.shape)
iftype(X) isnp.ndarray:
# X = torch.tensor(X, requires_grad=False).type(torch.FloatTensor)
X= (
torch.tensor(X, requires_grad=False)
.type(torch.FloatTensor)
.cuda(device, non_blocking=True)
)
eliftype(X) istorch.TensorandnotX.is_cuda:
X=X.type(torch.FloatTensor).cuda(device, non_blocking=True)
print("tensor size ", X.shape)
ifenable_dropout:
self.enable_test_dropout()
withtorch.no_grad():
out=self.forward(X)
max_val, idx=torch.max(out, 1)
idx=idx.data.cpu().numpy()
prediction=np.squeeze(idx)
print("prediction shape", prediction.shape)
delX, out, idx, max_val
returnprediction
损失函数
神经网络的训练需要一个损失函数来计算模型误差。训练的目标是最小化预测输出和目标输出之间的损失。我们的模型使用Dice Loss 和Weighted Logistic Loss的联合损失函数进行优化,其中权重补偿数据中的高类不平衡,并鼓励正确分割解剖边界。
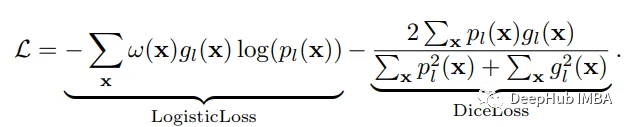
优化器
优化算法允许我们继续更新模型的参数并最小化损失函数的值,我们设置了以下的超参数:
学习率:初始设置为0.1,10次后降低1阶。这可以通过学习率调度器来实现。
权重衰减:0.0001。
批量大小:1。
动量:设置为0.95的高值,以补偿由于小批量大小而产生的噪声梯度。
训练网络
现在可以训练模型了。对于QuickNAT需要在3个(coronal, axial, sagittal)2d切片上训练3个模型。然后再聚合步骤中组合三个模型的概率生成最终结果,但是本文中只演示在coronal视图的2D切片上训练一个F-CNN模型,因为其他两个与之类似。
num_epochs=20
start_epoch=1
val_interval=1
train_loss_epoch_values= []
val_loss_epoch_values= []
best_ds_mean=-1
best_ds_mean_epoch=-1
ds_mean_train_values= []
ds_mean_val_values= []
# ds_LCE_values = []
# ds_LWM_values = []
# ds_LCC_values = []
print("START TRAINING. : model name = ", "quicknat")
forepochinrange(start_epoch, num_epochs):
print("==== Epoch ["+str(epoch) +" / "+str(num_epochs)+"] DONE ====")
checkpoint_name=CHECKPOINT_DIR+"/checkpoint_epoch_"+str(epoch) +"."+CHECKPOINT_EXTENSION
print(checkpoint_name)
state= {
"epoch": epoch,
"arch": "quicknat",
"state_dict": model_coronal.state_dict(),
"optimizer": optimizer.state_dict(),
"scheduler": scheduler.state_dict(),
}
save_checkpoint(state=state, filename=checkpoint_name)
print("\n==== Epoch [ %d / %d ] START ===="% (epoch, num_epochs))
steps_per_epoch=len(train_dataset_coronal_subset) /train_loader_coronal_subset.batch_size
model_coronal.train()
train_loss_epoch=0
val_loss_epoch=0
step=0
predictions_train= []
labels_train= []
predictions_val= []
labels_val= []
fori_batch, sample_batchedinenumerate(train_loader_coronal_subset):
inputs=sample_batched['image'].type(torch.FloatTensor)
labels=sample_batched['label'].type(torch.LongTensor)
# print(f"Train Input Shape: {inputs.shape}")
labels=labels.squeeze(1)
_img_channels, _img_height, _img_width=labels.shape
encoded_label=np.zeros((_img_height, _img_width, 1)).astype(int)
forj, clsinenumerate(MaskColorMap):
encoded_label[np.all(labels==cls.value, axis=0)] =j
labels=encoded_label
labels=torch.from_numpy(labels)
labels=torch.permute(labels, (2, 1, 0))
# print(f"Train Label Shape: {labels.shape}")
# plt.title("Train Label")
# plt.imshow(labels[0, :, :])
# plt.show()
optimizer.zero_grad()
outputs=model_coronal(inputs)
loss=loss_function(outputs, labels)
loss.backward()
optimizer.step()
scheduler.step()
withtorch.no_grad():
_, batch_output=torch.max(outputs, dim=1)
# print(f"Train Prediction Shape: {batch_output.shape}")
# plt.title("Train Prediction")
# plt.imshow(batch_output[0, :, :])
# plt.show()
predictions_train.append(batch_output.cpu())
labels_train.append(labels.cpu())
train_loss_epoch+=loss.item()
print(f"{step}/{len(train_dataset_coronal_subset) //train_loader_coronal_subset.batch_size}, Training_loss: {loss.item():.4f}")
step+=1
predictions_train_arr, labels_train_arr=torch.cat(predictions_train), torch.cat(labels_train)
# print(predictions_train_arr.shape)
dice_metric(predictions_train_arr, labels_train_arr)
ds_mean_train=dice_metric.aggregate().item()
ds_mean_train_values.append(ds_mean_train)
dice_metric.reset()
train_loss_epoch/=step
train_loss_epoch_values.append(train_loss_epoch)
print(f"Epoch {epoch+1} Train Average Loss: {train_loss_epoch:.4f}")
if (epoch+1) %val_interval==0:
model_coronal.eval()
step=0
withtorch.no_grad():
fori_batch, sample_batchedinenumerate(val_loader_coronal_subset):
inputs=sample_batched['image'].type(torch.FloatTensor)
labels=sample_batched['label'].type(torch.LongTensor)
# print(f"Val Input Shape: {inputs.shape}")
labels=labels.squeeze(1)
integer_encoded_labels= []
_img_channels, _img_height, _img_width=labels.shape
encoded_label=np.zeros((_img_height, _img_width, 1)).astype(int)
forj, clsinenumerate(MaskColorMap):
encoded_label[np.all(labels==cls.value, axis=0)] =j
labels=encoded_label
labels=torch.from_numpy(labels)
labels=torch.permute(labels, (2, 1, 0))
# print(f"Val Label Shape: {labels.shape}")
# plt.title("Val Label")
# plt.imshow(labels[0, :, :])
# plt.show()
val_outputs=model_coronal(inputs)
val_loss=loss_function(val_outputs, labels)
predicted=torch.argmax(val_outputs, dim=1)
# print(f"Val Prediction Shape: {predicted.shape}")
# plt.title("Val Prediction")
# plt.imshow(predicted[0, :, :])
# plt.show()
predictions_val.append(predicted)
labels_val.append(labels)
val_loss_epoch+=val_loss.item()
print(f"{step}/{len(val_dataset_coronal_subset) //val_loader_coronal_subset.batch_size}, Validation_loss: {val_loss.item():.4f}")
step+=1
predictions_val_arr, labels_val_arr=torch.cat(predictions_val), torch.cat(labels_val)
dice_metric(predictions_val_arr, labels_val_arr)
# dice_metric_batch(predictions_val_arr, labels_val_arr)
ds_mean_val=dice_metric.aggregate().item()
ds_mean_val_values.append(ds_mean_val)
# ds_mean_val_batch = dice_metric_batch.aggregate()
# ds_LCE = ds_mean_val_batch[0].item()
# ds_LCE_values.append(ds_LCE)
# ds_LWM = ds_mean_val_batch[1].item()
# ds_LWM_values.append(ds_LWM)
# ds_LCC = ds_mean_val_batch[2].item()
# ds_LCC_values.append(ds_LCC)
dice_metric.reset()
# dice_metric_batch.reset()
ifds_mean_val>best_ds_mean:
best_ds_mean=ds_mean_val
best_ds_mean_epoch=epoch+1
torch.save(model_coronal.state_dict(), os.path.join(BESTMODEL_DIR, "best_metric_model_coronal.pth"))
print("Saved new best metric model coronal")
print(
f"Current Epoch: {epoch+1} Current Mean Dice score is: {ds_mean_val:.4f}"
f"\nBest Mean Dice score: {best_ds_mean:.4f} "
# f"\nMean Dice score Left Cerebral Exterior: {ds_LCE:.4f} Mean Dice score Left White Matter: {ds_LWM:.4f} Mean Dice score Left Cerebral Cortex: {ds_LCC:.4f} "
f"at Epoch: {best_ds_mean_epoch}"
)
val_loss_epoch/=step
val_loss_epoch_values.append(val_loss_epoch)
print(f"Epoch {epoch+1} Average Validation Loss: {val_loss_epoch:.4f}")
print("FINISH.")
代码也是传统的Pytorch的训练步骤,就不详细解释了
绘制损失和精度曲线
训练曲线表示模型的学习情况,验证曲线表示模型泛化到未见实例的情况。我们使用matplotlib来绘制图形。还可以使用TensorBoard,它使理解和调试深度学习程序变得更容易,并且是实时的。
epoch=range(1, num_epochs+1)
# Plot Loss Curves
plt.figure(figsize=(18, 6))
plt.subplot(1, 3, 1)
plt.plot(epoch, train_loss_epoch_values, label='Training Loss')
plt.plot(epoch, val_loss_epoch_values, label='Validation Loss')
plt.title('Training and Validation Loss')
plt.xlabel('Epoch')
plt.legend()
plt.figure()
plt.show()
# Plot Train Dice Coefficient Curve
plt.figure(figsize=(18, 6))
plt.subplot(1, 3, 2)
x= [(i+1) foriinrange(len(ds_mean_train_values))]
plt.plot(x, ds_mean_train_values, 'blue', label='Train Mean Dice Score')
plt.title("Training Mean Dice Coefficient")
plt.xlabel('Epoch')
plt.ylabel('Mean Dice Score')
plt.show()
# Plot Validation Dice Coefficient Curve
plt.figure(figsize=(18, 6))
plt.subplot(1, 3, 3)
x= [(i+1) foriinrange(len(ds_mean_val_values))]
plt.plot(x, ds_mean_val_values, 'orange', label='Validation Mean Dice Score')
plt.title("Validation Mean Dice Coefficient")
plt.xlabel('Epoch')
plt.ylabel('Mean Dice Score')
plt.show()
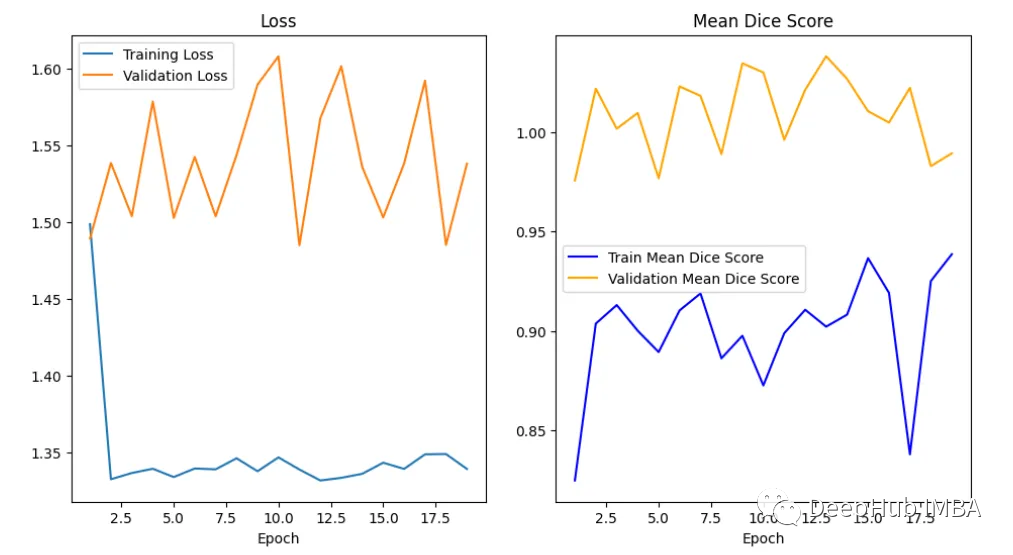
在曲线中,我们可以看到模型是过拟合的,因为验证损失上升而训练损失下降。这是深度学习算法中一个常见的陷阱,其中模型最终会记住训练数据,而无法对未见过的数据进行泛化。
避免过度拟合的技巧:
- 用更多的数据进行训练:更大的数据集可以减少过拟合。
- 数据增强:如果我们不能收集更多的数据,我们可以应用数据增强来人为地增加数据集的大小。
- 添加正则化:正则化是一种限制我们的网络学习过于复杂的模型的技术,因此可能会过度拟合。
评估网络
我们如何度量模型的性能?一个成功的预测是一个最大限度地扩大预测和真实之间的重叠。
这一目标的两个相关但不同的指标是Dice和Intersection / Union (IoU)系数,后者也被称为Jaccard系数。两个指标都在0(无重叠)和1(完全重叠)之间。
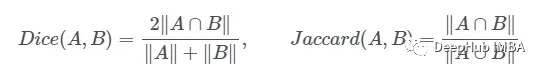
这两种指标都可以用于类似的情况,但是区别在于Dice Score倾向于平均表现,而IoU则帮助你理解最坏情况下的表现。
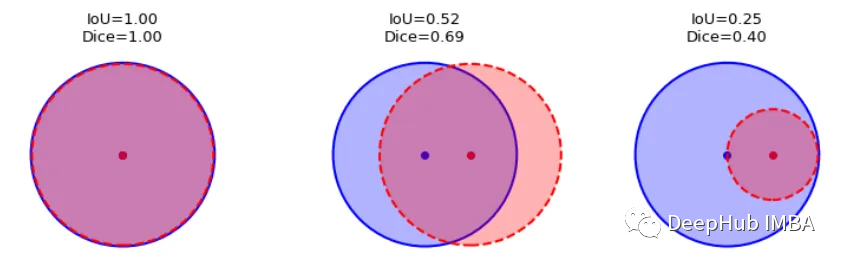
我们可以逐个类地检查度量标准,或者取所有类的平均值。这里将使用monai.metrics.DiceMetric来计算分数。一个更通用的方法是使用torchmetrics,但是因为这里使用了monai框架,所以就直接使用它内置的函数了。
我们可以看到Dice得分曲线的行为相当不寻常。主要是因为验证平均Dice得分高于1,这是不可能的,因为这个度量是在0和1之间。我们无法确定这种行为的主要原因,但我们建议在多类问题中为每个类单独提供度量计算,并始终提供可视化示例以进行可视化评估。
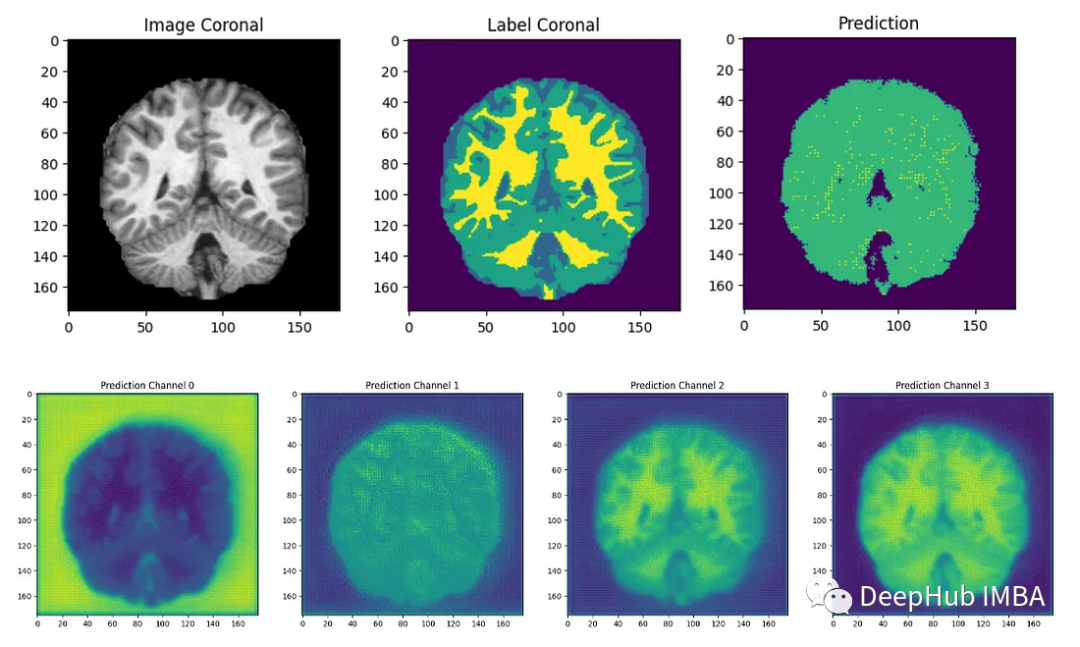
结果分析
最后我们要看看模型是如何推广到未知数据的这个模型预测的几乎所有东西都是左脑白质,一些像素是左脑皮层。尽管它的预测似乎是正确的,但仍有很大的改进空间,因为我们的模型太小了,可以选择更深的模型获得更好的效果。
总结
在本文中,我们介绍了如何训练QuickNAT来完成具有挑战性的大脑分割任务。我们尽可能遵循作者在他们的研究论文中解释的学习策略,这是本教程为了方便演示只在最简单的步骤上进行了演示,文本的完整代码:
https://github.com/inesdv26/Brain-Segmentation
作者:Ines del Val