除了随机过采样,SMOTE及其变体之外,还有许多方法可以对不平衡数据进行过采样。 在使用scikit-learn的make_classification默认设置生成的分类数据集中,使用交叉操作生成的样本在最相关的指标上胜过SMOTE和随机过采样。
本篇文章的目录如下
- 介绍
- 数据准备
- 随机过采样和SMOTE
- 交叉过采样
- 绩效指标评估
- 结论
介绍
我们中的许多人都会遇到处于使用不平衡数据集的预测模型的情况。
最流行的处理失衡的方法包括:
- 增加未被充分标记的的分类的权重
- 过采样技术
- 欠采样技术
- 过度采样和欠采样的组合
- 调整成本函数
这篇文章将讨论过采样技术,我们将特别研究依赖于在特征空间内插值的SMOTE变体(borderline SMOTE, ADASYN等)如何生成较少的新合成数据。
过度抽样的方法太多了。我们使用简单的单点、两点和均匀交叉操作对合成数据进行过采样,并将评价结果与随机过采样进行比较。一般情况下,将过采样和欠采样结合使用会更好,但是在本演示中,我们为了说明只使用过采样。
数据集准备
我们使用scikit-learn的make_classification函数来创建一个不平衡的数据集,该数据集包含两个类别中的5000个数据点(二进制分类)。目标为0的机会为95%,目标为1的机会为5%。
from sklearn.datasets import make_classification
import seaborn as sns
X, y = make_classification(
n_samples=5000, n_classes=2,
weights=[0.95, 0.05], flip_y=0
)
sns.countplot(y)
plt.show()

默认情况下,会创建20个特征,下面是X数组中的示例条目。

make_classification中的其余设置为默认设置,下面我们将数据分为训练和测试数据集。
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y)
随机和SMOTE过采样
现在,让我们准备函数以生成数据集,其中可以使用随机过采样和SMOTE对少数类(目标= 1)进行过采样。
from imblearn.over_sampling import SMOTE, RandomOverSampler
def oversample_random(X, y, rows_1, random_state):
'''Accepts X and y arrays along with the number of
required positively labeled samples (rows_1). Returns
randomly oversampled positively labeled data.
'''
X_random, y_random = RandomOverSampler(
sampling_strategy={1: rows_1},
random_state=random_state
).fit_resample(X_train, y_train)
return X_random, y_random
def oversample_smote(X, y, rows_1, k_neighbors, random_state):
'''Accepts X and y arrays along with the number of
required positively labeled samples (rows_1) and number
of nearest neighbors to consider in the SMOTE algorithm.
Returns SMOTE oversampled positively labeled data.
'''
X_smote, y_smote = SMOTE(
sampling_strategy={1: rows_1},
k_neighbors=k_neighbors,
random_state=random_state
).fit_resample(X, y)
return X_smote, y_smote
请注意,为了方便进行比较我们使用普通的SMOTE而不是边界SMOTE,ADASYN,SVM-SMOTE等。在下一部分中使用交叉操作生成样本时,我们不会考虑是在边界附近生成样本还是被认为有噪声等。
如果您不熟悉随机过采样和SMOTE,则在网上有很多资源,但是这里有个简短的回顾:
随机过采样涉及从我们尝试过采样的少数类中随机选择数据点,然后将它们作为重复项再次添加回数据集。

随机过采样的插图,较大的气泡代表随机选择用于过采样的数据点,它们在数据集中显示为重复项
SMOTE涉及从少数类中查看样本的最近邻居,并在该样本与从其最近邻居中随机选择的另一个样本之间插入特征值。

SMOTE,红色数据点是插值后综合生成的数据
交叉算子
交叉运算在遗传算法中被广泛使用,它是由有性生殖中发生的遗传物质的交叉驱动的。
该操作相对简单,其中“染色体”中的信息由两个“父母”贡献以生成“孩子”。在我们的用例中,染色体中的信息仅是特征值。

通常以位阵列表示信息以获得更好的性能。
例如:在我们的数据集中,我们有20个特征和5000个样本。在单点交叉操作中,我们可以选择两个“父级”,例如样本#20和样本#1500,并选择一个随机的交叉点,例如第十特征。然后,我们生成一个新的“子级”,即新数据点,该新数据点的第一个父级(样本#20)具有特征1–9,第二个父级(样本#1500)具有特征10–20。

我们将考虑3种交叉操作:
- single-point 单点
- two-point 两点
- uniform 均匀
单点交叉操作是上面所示的示例,其中交叉点之前的要素由一个父级提供,而交叉点之后的要素由另一父级提供。
在两点交叉操作中,父级1贡献第一个交叉点之前的子数据点的特征值,然后父级2贡献其特征值直到第二个交叉点,然后贡献在第二个交叉点之后返回父级1。

通过均匀交叉操作,两个父级中的任何一个都可以为20个要素中的任何一个对子数据点的要素值做出贡献。

下面是我们用来生成交叉样本的函数。还有一个附加参数knn,它过滤掉所有生成的样本,这些样本的最近邻居的目标是0而不是1。默认情况下,此选项设置为False。
最后,请注意,父母的选择完全是随机的,而不是基于遗传算法中常见的“fitness”。
def oversample_crossover(X, y, rows_1, mode="single", knn=False, random_state=1):
'''Oversampled positively labeled data using a crossover
operation.
Args:
X: Array of explanatory variables to oversample data from
y: Array of labels associated with X
rows_1: Number of positively labled rows required (original + oversampled)
mode: Choice between single-point ("single"), two-point ("two"), and
uniform ("uniform") crossover operation
knn: If set to True, drops oversampled data whose nearest neighbor is not
positively labeled.
random_state: random state to pass to ensure reproducability of results
Returns:
X_crossover: Array of explanatory variables associated with the new
oversampled data (includes original + new data)
y_crossover: Labels associated with X_crossover
'''
np.random.seed(random_state)
# Potential because if the knn parameter is set to True,
# those samples need to be checked if their nearest neighbor
# has a label of 1
potential_samples = []
X_positive = X[y==1]
no_rows = X_positive.shape[0]
no_cols = X_positive.shape[1]
# assume % of 1s is at least 3%, this is relevant if knn=True
for i in range(int(rows_1/0.03)):
parent_1 = np.random.randint(0, no_rows)
parent_2 = np.random.randint(0, no_rows)
if mode=="single":
cross_point = np.random.randint(1, no_cols)
mask = np.array([1 if col_no < cross_point else 0
for col_no in range(no_cols)])
elif mode=="two":
cross_point_1 = np.random.randint(1, no_cols-1)
cross_point_2 = np.random.randint(cross_point_1, no_cols-1)
mask = np.array([
1 if col_no < cross_point_1 or col_no > cross_point_2
else 0 for col_no in range(no_cols)])
elif mode=="uniform":
mask = np.random.randint(0, 2, no_cols)
else:
raise ValueError("Accebtable options for mode: single, two, uniform")
potential_samples.append(
(X_positive[parent_1] * mask)
+ (X_positive[parent_2] * (1-mask))
)
if knn == False:
X_crossover = potential_samples
else:
scaler = MinMaxScaler().fit(X)
X_scaled = scaler.transform(X)
potential_samples_scaled = scaler.transform(potential_samples)
model = KNeighborsClassifier(n_neighbors=1)
model.fit(X_scaled, y)
knn_filter = (model.predict_proba(
potential_samples_scaled)[:, 1] > 0
)
X_crossover = np.array(potential_samples)[
knn_filter]
required_rows = rows_1 - (y==1).sum()
X_crossover = np.vstack([X, X_crossover[:required_rows]])
y_crossover = np.hstack([
y, np.ones(required_rows)])
return X_crossover, y_crossover
效果评估
最后,我们遍历30个随机状态,比较随机森林分类器在原始数据集上的性能以及11种过采样方法,
- 随机过采样
- SMOTE-1个邻居
- SMOTE-3个邻居
- SMOTE-5个邻居
- SMOTE-10个邻居
- 单点交叉
- 带KNN滤波器的单点交叉
- 两点交叉
- 带KNN滤波器的两点交叉
- 均匀交叉
- 带有KNN滤波器的均匀分频
我们还将研究7个分类指标:
- ROC AUC-ROC曲线下的面积
- PR AUC —精确召回曲线下的面积
- 平衡的准确性-这也等同于两个标签的平均召回率
- 最大F1 —使用最佳概率阈值可获得的最大F1分数
- 召回
- 精确
- F1
以下是代码和结果…
metrics_dict = {"ROC AUC": [],
"PR AUC": [],
"Balanced Accuracy": [],
"Max F1": [],
"Recall": [],
"Precision": [],
"F1": []
}
results_original = deepcopy(metric_dict)
results_random = deepcopy(metric_dict)
results_smote_1 = deepcopy(metric_dict)
results_smote_3 = deepcopy(metric_dict)
results_smote_5 = deepcopy(metric_dict)
results_smote_10 = deepcopy(metric_dict)
results_singlex = deepcopy(metric_dict)
results_singlex_knn = deepcopy(metric_dict)
results_twox = deepcopy(metric_dict)
results_twox_knn = deepcopy(metric_dict)
results_uniformx = deepcopy(metric_dict)
results_uniformx_knn = deepcopy(metric_dict)
for i in range(30):
clf = RandomForestClassifier(random_state=i)
X_random, y_random = oversample_random(X_train, y_train, N_ROWS_1, i)
X_smote_1, y_smote_1 = oversample_smote(X_train, y_train, N_ROWS_1, 1, i)
X_smote_3, y_smote_3 = oversample_smote(X_train, y_train, N_ROWS_1, 3, i)
X_smote_5, y_smote_5 = oversample_smote(X_train, y_train, N_ROWS_1, 5, i)
X_smote_10, y_smote_10 = oversample_smote(X_train, y_train, N_ROWS_1, 10, i)
X_singlex, y_singlex = oversample_crossover(
X_train, y_train, N_ROWS_1, mode="single", knn=False, random_state=i)
X_singlex_knn, y_singlex_knn = oversample_crossover(
X_train, y_train, N_ROWS_1, mode="single", knn=True, random_state=i)
X_twox, y_twox = oversample_crossover(
X_train, y_train, N_ROWS_1, mode="two", knn=False, random_state=i)
X_twox_knn, y_twox_knn = oversample_crossover(
X_train, y_train, N_ROWS_1, mode="two", knn=True, random_state=i)
X_uniformx, y_uniformx = oversample_crossover(
X_train, y_train, N_ROWS_1, mode="uniform", knn=False, random_state=i)
X_uniformx_knn, y_uniformx_knn = oversample_crossover(
X_train, y_train, N_ROWS_1, mode="uniform", knn=True, random_state=i)
model = clf.fit(X_train, y_train)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_original["ROC AUC"].append(roc_auc)
results_original["PR AUC"].append(pr_auc)
results_original["Max F1"].append(max_f1)
results_original["Balanced Accuracy"].append(balanced_accuracy)
results_original["Recall"].append(recall)
results_original["Precision"].append(precision)
results_original["F1"].append(f1)
model = clf.fit(X_random, y_random)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_random["ROC AUC"].append(roc_auc)
results_random["PR AUC"].append(pr_auc)
results_random["Max F1"].append(max_f1)
results_random["Balanced Accuracy"].append(balanced_accuracy)
results_random["Recall"].append(recall)
results_random["Precision"].append(precision)
results_random["F1"].append(f1)
model = clf.fit(X_smote_1, y_smote_1)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_smote_1["ROC AUC"].append(roc_auc)
results_smote_1["PR AUC"].append(pr_auc)
results_smote_1["Max F1"].append(max_f1)
results_smote_1["Balanced Accuracy"].append(balanced_accuracy)
results_smote_1["Recall"].append(recall)
results_smote_1["Precision"].append(precision)
results_smote_1["F1"].append(f1)
model = clf.fit(X_smote_3, y_smote_3)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_smote_3["ROC AUC"].append(roc_auc)
results_smote_3["PR AUC"].append(pr_auc)
results_smote_3["Max F1"].append(max_f1)
results_smote_3["Balanced Accuracy"].append(balanced_accuracy)
results_smote_3["Recall"].append(recall)
results_smote_3["Precision"].append(precision)
results_smote_3["F1"].append(f1)
model = clf.fit(X_smote_5, y_smote_5)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_smote_5["ROC AUC"].append(roc_auc)
results_smote_5["PR AUC"].append(pr_auc)
results_smote_5["Max F1"].append(max_f1)
results_smote_5["Balanced Accuracy"].append(balanced_accuracy)
results_smote_5["Recall"].append(recall)
results_smote_5["Precision"].append(precision)
results_smote_5["F1"].append(f1)
model = clf.fit(X_smote_10, y_smote_10)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_smote_10["ROC AUC"].append(roc_auc)
results_smote_10["PR AUC"].append(pr_auc)
results_smote_10["Max F1"].append(max_f1)
results_smote_10["Balanced Accuracy"].append(balanced_accuracy)
results_smote_10["Recall"].append(recall)
results_smote_10["Precision"].append(precision)
results_smote_10["F1"].append(f1)
model = clf.fit(X_singlex, y_singlex)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_singlex["ROC AUC"].append(roc_auc)
results_singlex["PR AUC"].append(pr_auc)
results_singlex["Max F1"].append(max_f1)
results_singlex["Balanced Accuracy"].append(balanced_accuracy)
results_singlex["Recall"].append(recall)
results_singlex["Precision"].append(precision)
results_singlex["F1"].append(f1)
model = clf.fit(X_singlex_knn, y_singlex_knn)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_singlex_knn["ROC AUC"].append(roc_auc)
results_singlex_knn["PR AUC"].append(pr_auc)
results_singlex_knn["Max F1"].append(max_f1)
results_singlex_knn["Balanced Accuracy"].append(balanced_accuracy)
results_singlex_knn["Recall"].append(recall)
results_singlex_knn["Precision"].append(precision)
results_singlex_knn["F1"].append(f1)
model = clf.fit(X_twox, y_twox)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_twox["ROC AUC"].append(roc_auc)
results_twox["PR AUC"].append(pr_auc)
results_twox["Max F1"].append(max_f1)
results_twox["Balanced Accuracy"].append(balanced_accuracy)
results_twox["Recall"].append(recall)
results_twox["Precision"].append(precision)
results_twox["F1"].append(f1)
model = clf.fit(X_twox_knn, y_twox_knn)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_twox_knn["ROC AUC"].append(roc_auc)
results_twox_knn["PR AUC"].append(pr_auc)
results_twox_knn["Max F1"].append(max_f1)
results_twox_knn["Balanced Accuracy"].append(balanced_accuracy)
results_twox_knn["Recall"].append(recall)
results_twox_knn["Precision"].append(precision)
results_twox_knn["F1"].append(f1)
model = clf.fit(X_uniformx, y_uniformx)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_uniformx["ROC AUC"].append(roc_auc)
results_uniformx["PR AUC"].append(pr_auc)
results_uniformx["Max F1"].append(max_f1)
results_uniformx["Balanced Accuracy"].append(balanced_accuracy)
results_uniformx["Recall"].append(recall)
results_uniformx["Precision"].append(precision)
results_uniformx["F1"].append(f1)
model = clf.fit(X_uniformx_knn, y_uniformx_knn)
roc_auc = roc_auc_score(y_test, model.predict_proba(X_test)[:, 1])
precision_, recall_, _ = precision_recall_curve(
y_test, model.predict_proba(X_test)[:, 1])
f1_ = (2*precision_*recall_) / (recall_+precision_)
pr_auc = auc(recall_, precision_)
max_f1 = np.max(f1_)
recall = recall_score(y_test, model.predict(X_test))
precision = precision_score(y_test, model.predict(X_test))
f1 = f1_score(y_test, model.predict(X_test))
balanced_accuracy = balanced_accuracy_score(y_test, model.predict(X_test))
results_uniformx_knn["ROC AUC"].append(roc_auc)
results_uniformx_knn["PR AUC"].append(pr_auc)
results_uniformx_knn["Max F1"].append(max_f1)
results_uniformx_knn["Balanced Accuracy"].append(balanced_accuracy)
results_uniformx_knn["Recall"].append(recall)
results_uniformx_knn["Precision"].append(precision)
results_uniformx_knn["F1"].append(f1)
metrics = ["ROC AUC", "PR AUC", "Max F1",
"Balanced Accuracy", "Recall",
"Precision", "F1"]
for metric in metrics:
plt.figure(figsize=(30, 10))
data = pd.DataFrame(data={
"Original": results_original[metric],
"Random": results_random[metric],
"SMOTE\nk=1": results_smote_1[metric],
"SMOTE\nk=3": results_smote_3[metric],
"SMOTE\nk=5": results_smote_5[metric],
"SMOTE\nk=10": results_smote_10[metric],
"Crossover\nSingle": results_singlex[metric],
"Crossover\nSingle KNN": results_singlex_knn[metric],
"Crossover\nTwo-points": results_twox[metric],
"Crossover\nTwo-points KNN": results_twox_knn[metric],
"Crossover\nUniform": results_uniformx[metric],
"Crossover\nUniform KNN": results_uniformx_knn[metric],
})
sns.boxplot(data=data)
plt.title(metric)
plt.show()

交叉过采样的所有变体以及带有SMOTE的所有值(最近邻参数#的所有值k)均胜过原始数据集和随机过采样。
表现最好的是SMOTE,k = 5和k = 10,以及单点交叉(有和没有KNN)。
以上结果是由较高的查全率驱动的,并且表明过采样数据的新颖性,因为随机森林分类器可以识别特征空间中可能对应于目标1的新区域。
但是,ROC AUC指标并不是在不平衡数据集中使用的最佳指标。我们接下来看的精度-召回曲线可以说更合适。

在上面可以清楚地看出,在不同的k个参数上,交叉过采样的所有变体都胜过SMOTE。
没有KNN的单点和两点交叉操作是过滤器中表现最好的。
我寻找的另一个度量标准是选择最佳概率阈值后可获得的最大F1分数。那就是下面的最大F1图。

同样,这些见解与从PR AUC图表获得的见解相同。交叉变体的表现尤其出色,尤其是没有KNN的单点和两点交叉。

平衡准确度等于召回率为1s和召回率为0s的未加权平均值。并且这两个值有相等的权重。
准确性和其他指标之间保持平衡的缺点是,假设使用0.5的概率阈值,他们会考虑模型的预测性能。通常,使用不同的阈值,模型可能会具有明显更好的性能。
但是,平衡的精度表明交叉采样率过高,这是明显的获胜者,并且在均匀交叉方面略有优势,并且没有KNN。

召回率的比较也再次证实了我们以前对交叉采样率过高的表现的见解。在这种情况下,参数为10的SMOTE也是性能最高的,但是在下面的精度比较中,我们可以看到,即使使用具有更多邻居的SMOTE可以添加一些新颖的数据来增加召回率,但精度下降是 与使用交叉机制相比,情况更为严重。
这说明了在更平衡的指标(例如PR AUC,平衡的精度和Max F1)上实现了更好的性能交叉采样。

交叉和SMOTE过采样实现的更高召回率带来了精度的提高。当我们开始以1为目标标记合成的过采样数据时,即使我们不确定应分配的标签是100%,精度也会下降。
通常,在大多数数据集中,此类过采样技术会降低精度。
如前所述,一个关键的见解是,通过交叉过采样获得的数据的精度要比具有高k的SMOTE更好。
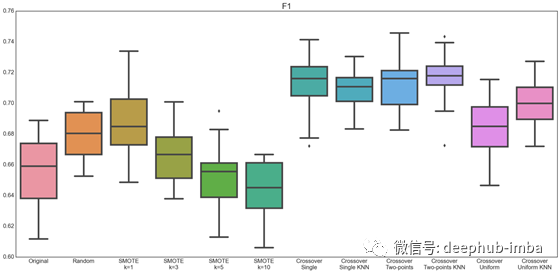
最后F1分数表明,在实现召回率和精度的完美结合方面,具有单边和两点交叉优势的交叉过采样是最佳技术。我更喜欢使用考虑不同概率阈值的Max F1分数。
总结
我们可以设计出许多过采样技术。本文的目的是展示非常简单的技术如何通过允许特征空间中的非线性组合实现良好的性能。
这在上面的数据集中成立,但是我看到的数据集中,与此类技术相关的精度损失导致性能指标降低,因此每个数据集都是不同的,因此应以不同的方式处理。
最后一点是,我发现在将交叉过采样与SMOTE结合使用时,使用整体技术对数据进行过采样效果很好,因此尝试使用不同的技术生成综合数据也有助于创建更好的集合。
作者:Bassel Karami
deephub翻译组