LEfSe (Linear discriminant analysis Effect Size) 是一种用于发现和解释高维度数据生物标识(基因、通路和分类单元等)的分析工具,可以进行两个或多个分组的比较,它强调统计意义和生物相关性,能够在组与组之间寻找具有统计学差异的生物标识(Biomarker)。
1. 在线安装
Galaxy 中可以使用 admin 账号在 Tool Shed 中直接搜索 lefse,并根据提示一步一步进行安装。本文章的所有截图与操作均来自于 zGalaxy,一个基于 Galaxy release_17.09,部署在阿里云 ECS 经过深度定制的中文版生物信息分析测试平台。
安装完成的网页界面:
2. 模块依赖
LEfSe 的执行需要解决 R、python 依赖:
- R libraries: splines, stats4, survival, mvtnorm, modeltools, coin, MASS
- python libraries: rpy2 (v. 2.1 or higher), numpy, matplotlib (v. 1.0 or higher), argparse
$ cd <GALAXY_ROOT>$ . .venv/bin/activate(.venv)galaxy@ecs-steven 16:30:55 /data/galaxy-dist/galaxy$ pip install matplotlib==1.5.0(.venv)galaxy@ecs-steven 16:30:55 /data/galaxy-dist/galaxy$ pip install rpy2==2.8.6
matplotlib >= 2.0.0 会导致 LEfSe 的
B)LDAEffectSize(LEfSe)运行出现 warnning,虽然生成的结果是没问题的。
/data/galaxy-dist/galaxy/.venv/lib/python2.7/site-packages/matplotlib/artist.py:896: MatplotlibDeprecationWarning: The set_axis_bgcolor function was deprecated in version 2.0. Use set_facecolor instead.return func(v)
3. 测试与使用
3.1 A) Format Data for LEfSe
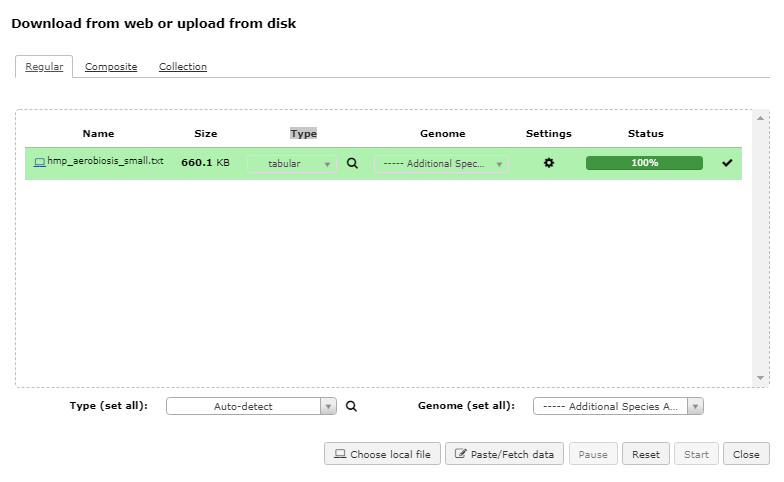
第一步,点击 Galaxy 的 "获取数据" → "数据上传" → "Choose local file" → 选择本地文件(hmpaerobiosissmall.txt) → 设置 Type: tabular → **"start"**。
wget http://huttenhower.sph.harvard.edu/webfm_send/129 -O hmp_aerobiosis_small.txt
第二步,点击 Galaxy 中 LEfSe 分析下的 **"A) Format Data for LEfSe"**,选择第一步输入的数据,设置参数如下。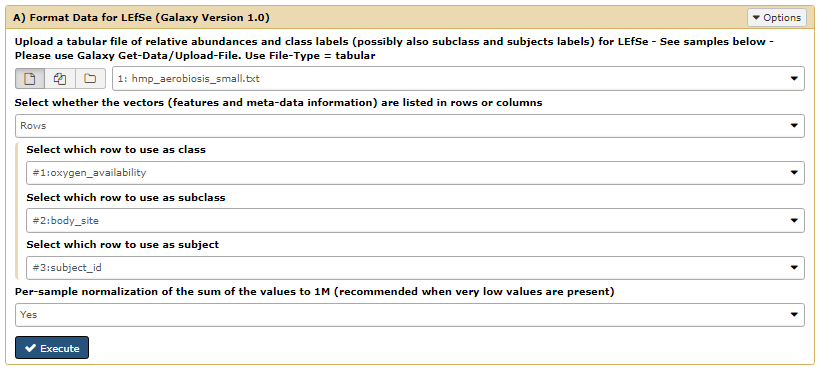
第三步,点击 **"Execute"**,提交任务执行。任务完成,在右侧历史栏可以看到生成的结果 **"2: A) Format Data for LEfSe on data 1"**。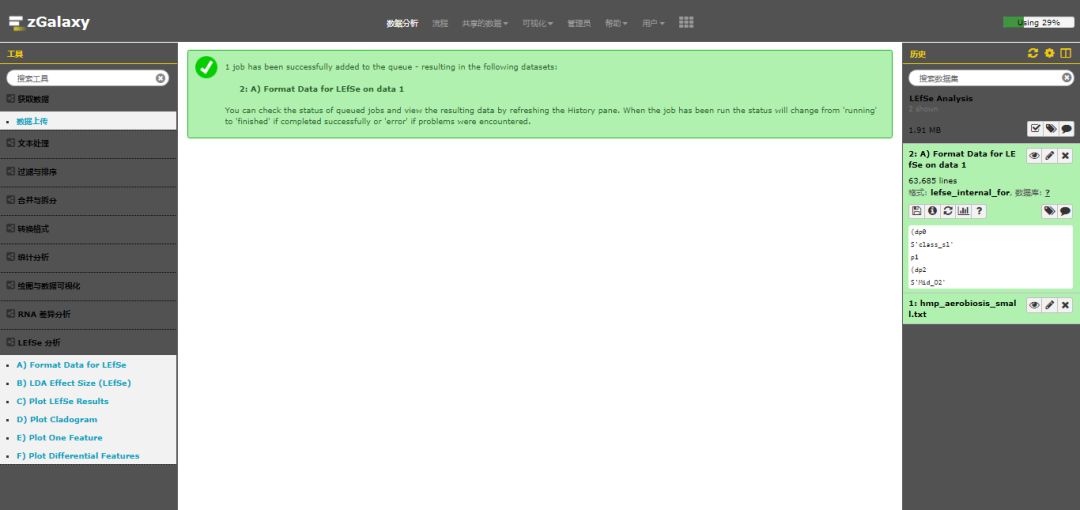
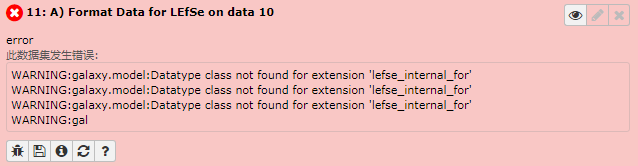
如果 Galaxy 历史栏的结果中出现格式报错:
这是因为在 Galaxy 中执行 A) Format Data for LEfSe 分析产生的结果默认为
lefse_internal_for格式,但 Galaxy 本身无法识别该格式。这就需要我们在 config/datatypesconf.xml 中增加 lefseinternal_for 数据格式的登记信息,然后重启 Galaxy 。
<?xml version="1.0"?><datatypes><registration>...<datatypeextension="lefse"type="galaxy.datatypes.data:Lefse"display_in_upload="true"/><datatypeextension="lefse_res"type="galaxy.datatypes.tabular:LefseRes"display_in_upload="true"/></registration></datatypes>参考:https://toolshed.g2.bx.psu.edu/repository/viewchangeset?ctxstr=db64b6287cd6&id=cabebb0465f009e4
3.2. B) LDA Effect Size (LEfSe)
第一步,点击 Galaxy 中 LEfSe 分析下的 **"B) LDA Effect Size (LEfSe)"**,选择 A 的结果数据,设置参数如下。
第二步,点击 **"Execute"**,提交任务执行。任务完成,在右侧历史栏可以看到生成的结果 **"3: B) LDA Effect Size (LEfSe) on data 2"**。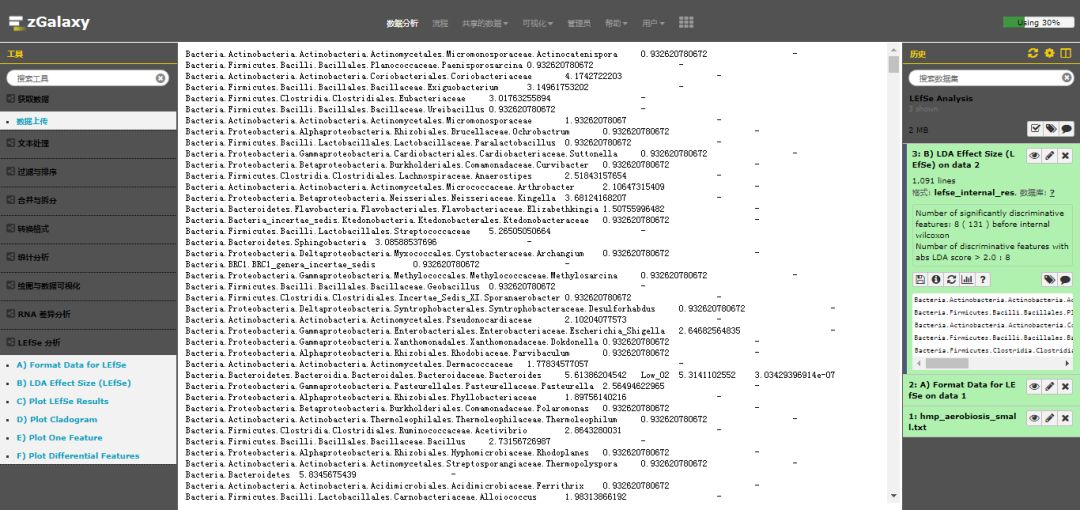
3.3. C) Plot LEfSe Results
第一步,点击 Galaxy 中 LEfSe 分析下的 **"C) Plot LEfSe Results"**,选择 B 的结果数据,设置参数如下。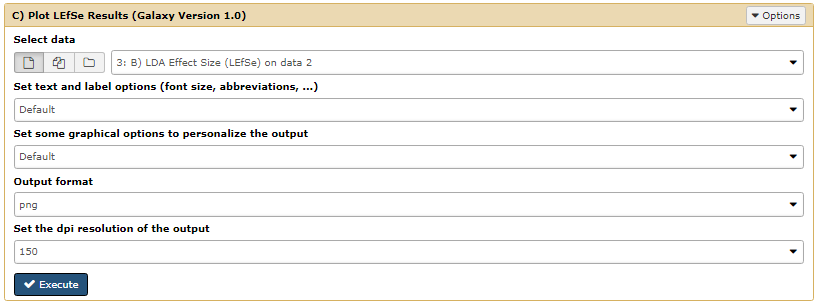
第二步,点击 **"Execute"**,提交任务执行。任务完成,在右侧历史栏可以看到生成的结果 **"4: C) Plot LEfSe Results on data 3"**。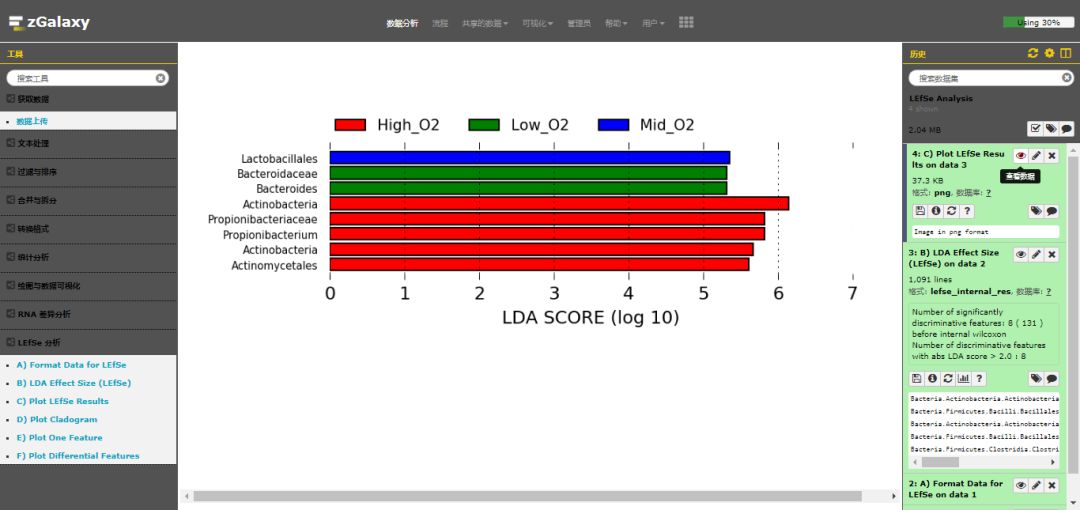
3.4. D) Plot Cladogram
第一步,点击 Galaxy 中 LEfSe 分析下的 **"D) Plot Cladogram"**,选择 B 的结果数据,设置参数如下。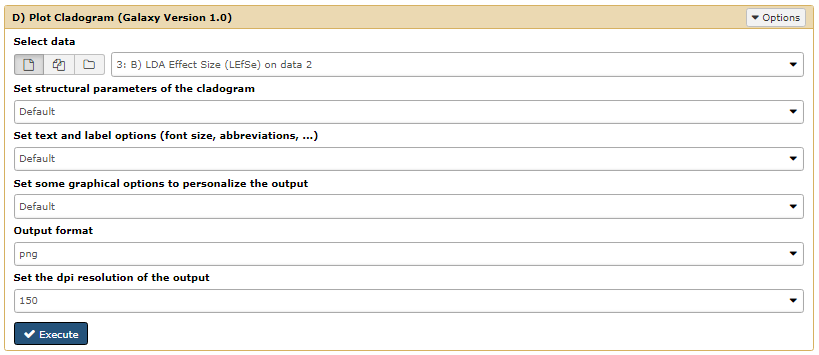
第二步,点击 **"Execute"**,提交任务执行。任务完成,在右侧历史栏可以看到生成的结果 **"5: D) Plot Cladogram on data 3"**。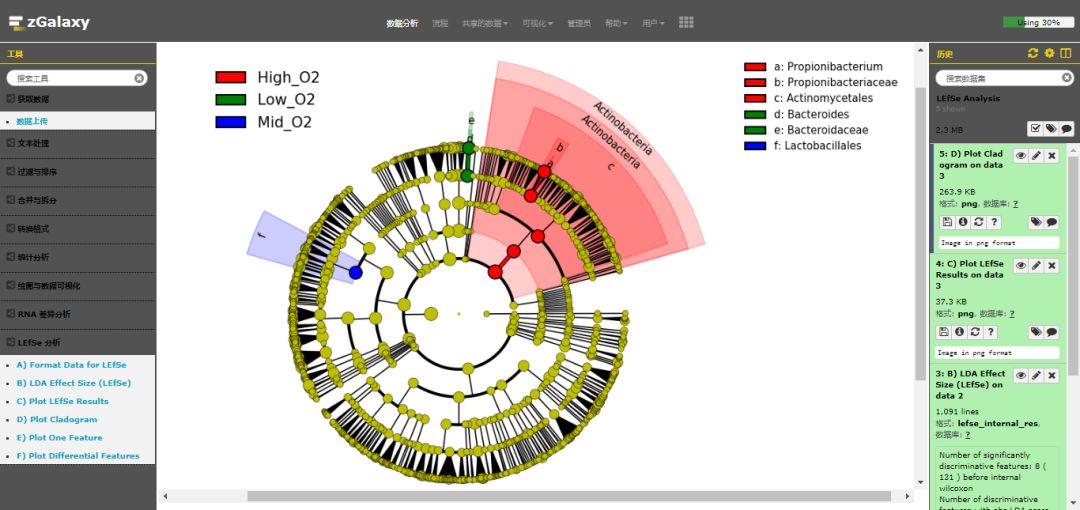
3.5. E) Plot One Feature
第一步,点击 Galaxy 中 LEfSe 分析下的 **"E) Plot One Feature"**,选择 A 和 B 的结果数据,设置参数如下。
第二步,点击 **"Execute"**,提交任务执行。任务完成,在右侧历史栏可以看到生成的结果 **"6: E) Plot One Feature on data 3 and data 2"**。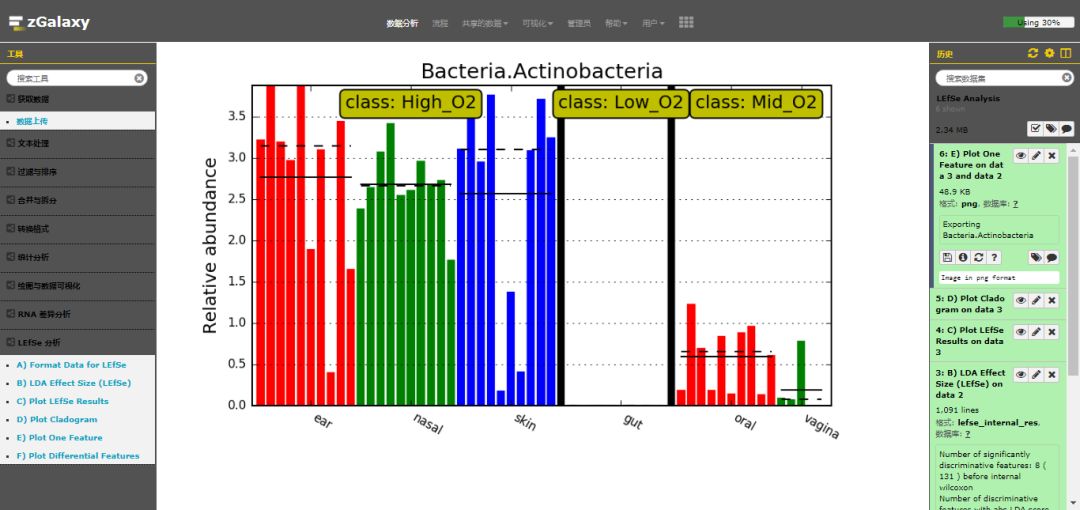
3.6. F) Plot Differential Features
第一步,点击 Galaxy 中 LEfSe 分析下的 **"F) Plot Differential Features"**,选择 A 和 B 的结果数据,设置参数如下。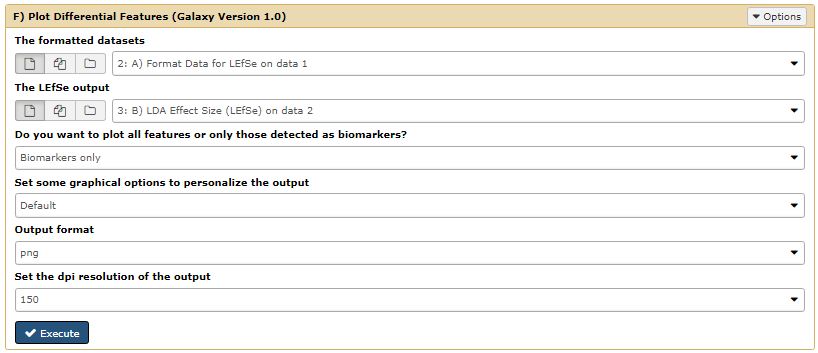
第二步,点击 **"Execute"**,提交任务执行。任务完成,在右侧历史栏可以看到生成的结果 **"7: F) Plot Differential Features on data 3 and data 2"*,点击眼睛图片可以下载本次分析的打包文件(.zip)。
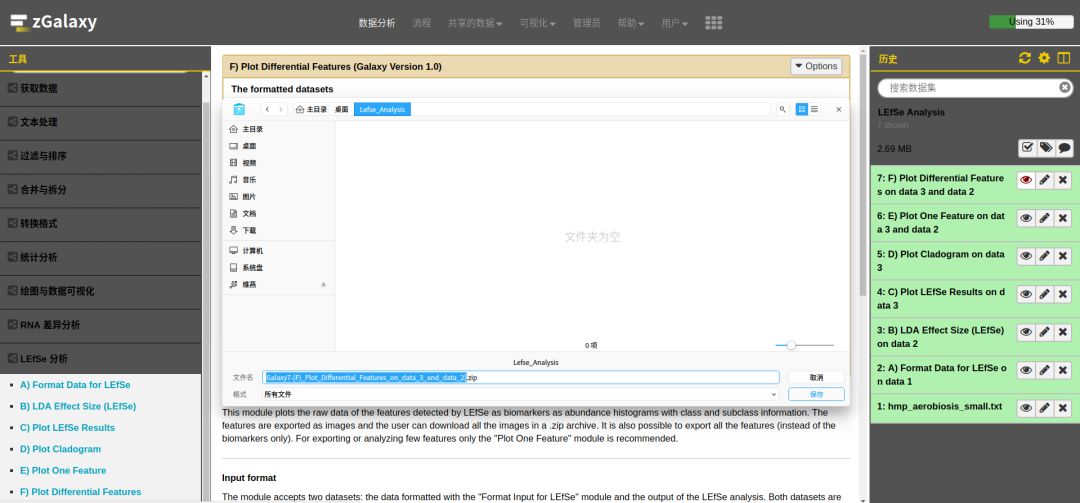
·end·
—如果喜欢,快分享给你的朋友们吧—
我们一起愉快的玩耍吧
本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
版权归原作者 BioIT爱好者 所有, 如有侵权,请联系我们删除。