1 写在前面
nnUnet的作者是Linux平台下实现的,Windows下实现需要一些别的步骤,翻阅了很多博文,踩了很多坑,终于用官方数据跑通了一遍。
2 代码下载
2.1 Windows下:
GitHub - marcus-wirtz-snkeos/nnUNet-Windows
mirrors / MRCWirtz / nnUNet-1 · GitCode
这两个都是一样的,感谢这些大佬把代码变成了Windows可用的!!!
2.2 Linux下:
GitHub - MIC-DKFZ/nnUNet
这个代码就是nnUnet作者的代码
3 虚拟环境
3.1 代码放置
将下载好的代码解压到放在某个盘,我放在了D盘的A_A文件夹里。
用pycharm打开项目nn-Unet-Windows-master,我把项目改了名字为nnUnet-win,之后项目的操作都是这个名字。也就是说,我的这个项目的路径就是D:\A_A\nnUNet-win。
项目里面应该包含这些:
接下来所有步骤都是在pycharm的终端进行的。
3.2 创建虚拟环境
打开终端Terminal
创建虚拟环境:
conda create -n nnUnet_win python=3.7
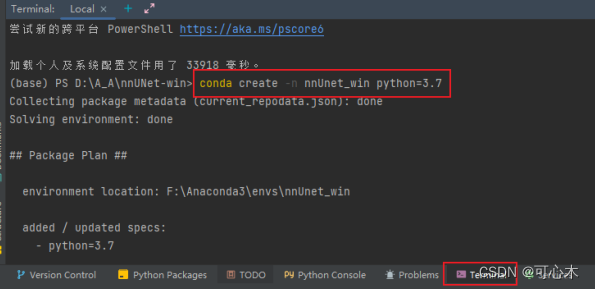
激活环境:
conda activate nnUnet_win
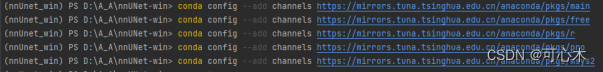
添加镜像源:
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/r
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/pro
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/msys2
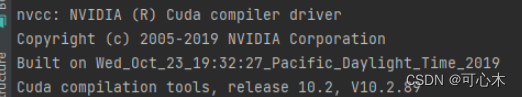
查看cuda,输入:
nvcc -V
显示为cuda版本为10.2
安装torch:
可以查看cuda与torch版本对应:Previous PyTorch Versions | PyTorch
需要安装cuda=10.2对应的torch版本,我安装的是1.7的,终端输入:
conda install pytorch==1.7.1 torchvision==0.8.2 torchaudio==0.7.2 cudatoolkit=10.2 -c pytorch
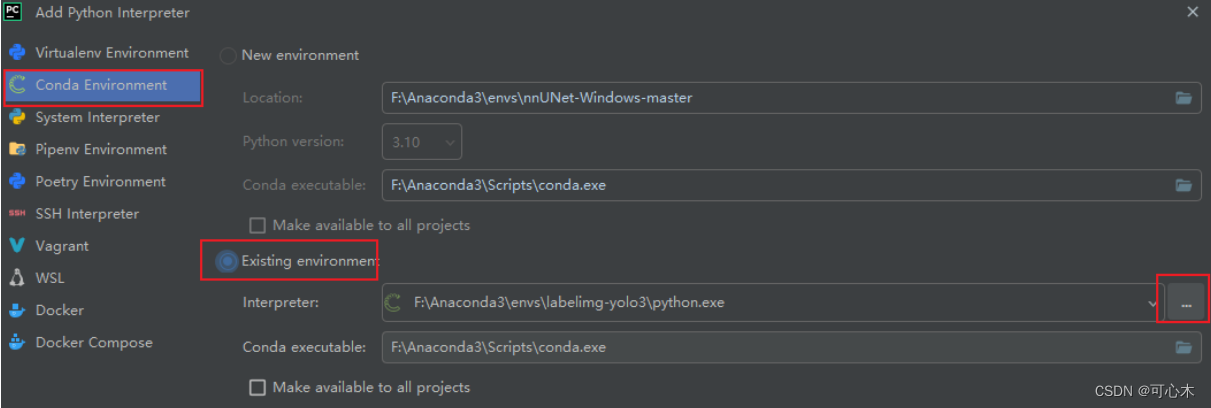
配置pycharm的虚拟环境:
依次点开file-->settings --> Project:nnUnet-Windows-master --> Python Interpreter

点击+号,把你创建的虚拟环境设置在这里。选择Conda Environment--> Existing environment,找到自己的Anaconda所在文件夹,选择envs-->nnUnet-win-->python.exe,然后点击ok。

4 安装nnUnet的相关配置
4.1 requirements
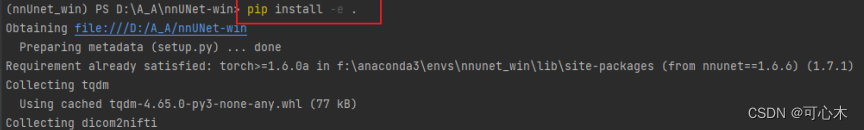
pip install -e .
若安不好,就加一个源,如清华源:
pip install -e . -i https://pypi.tuna.tsinghua.edu.cn/simple/
这一步是安装setup.py文件里的requirements。
4.2 安装隐藏层
非必须,安不上也没关系,隐藏层使nnU-net能够给出其生成的网络拓扑图
pip install --upgrade git+https://github.com/nanohanno/hiddenlayer.git@bugfix/get_trace_graph#egg=hiddenlayer
显示这样就装好了:
5 数据集准备
5.1 创建文件
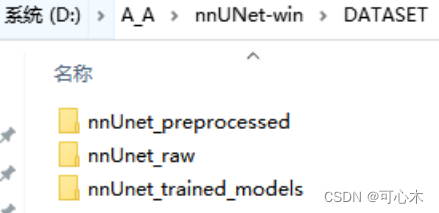
第一步:在nnUnet-win里新建文件夹DATASET,会用它来存放数据
第二步:在DATASET文件夹中创建三个文件夹,它们分别是nnUNet_raw,nnUNet_preprocessed,nnUNet_trained_models
第三步:在nnUNet_raw文件夹中创建nnUNet_cropped_data文件夹(待会用来存放原始数据)和nnUNet_raw_data文件夹(待会用来存放crop以后的数据)。
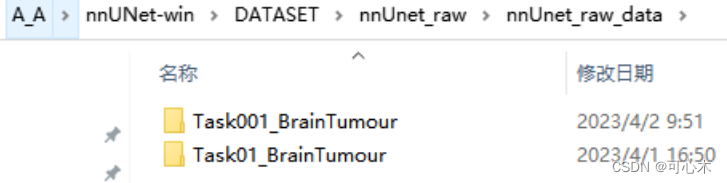
第四步:在nnUNet_raw_data文件夹中创建一个名为Task01_BrainTumour的文件夹
解释:这个Task01_BrainTumour是nnUNet项目参加的一个医学图像十项全能竞赛的任务名,本文也是实践的这个任务,类似的还有Task02_Heart(分割心脏的)。
这里的Task001_BrainTumour是我后面运行程序生成的,这里不用管他。
5.2 数据集下载
官网下载task01数据集
进入网站:http://medicaldecathlon.com/.(<–网上学科议建<–)
下载对应的数据集Task01_BrainTumour
下载好后,解压后是一个文件夹Task01_BrainTumour:
打开它,是这样的:
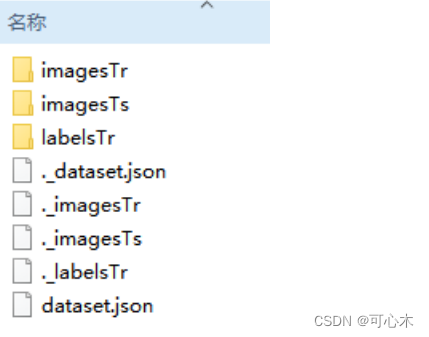
将所有东西复制到D:\A_A\nnUNet-win\DATASET\nnUnet_raw\nnUnet_raw_data\Task01_BrainTumour里面。
其实,这里重要的东西就是四个文件夹:imagesTr、imageTs、labelsTr、dataset.json。
- json文件是对三个文件夹内容的字典呈现(关乎你的训练)
- imagesTr是你的训练数据集,打开后很多的有序的nii.gz的训练文件
- labelsTr里对应imagesTr的标签文件,同样为nii.gz
- imageTs训练阶段先不管,其实这个文件夹出现在任何位置都可以。(解释:nnUNet使用的是5折交叉验证,并没有验证集)。
5.3 路径添加
文件路径添加
Windows下,还需要为这些添加路径。
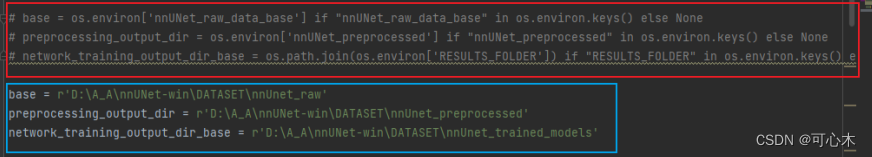
第一步:进入nnunet-->paths.py,到29行,把29-31行注释掉,根据自己的路径,把相关路径修改并添加。像图片里的那样,注释红框,添加蓝框
可以直接复制:
base = r'D:\A_A\nnUNet-win\DATASET\nnUnet_raw'
preprocessing_output_dir = r'D:\A_A\nnUNet-win\DATASET\nnUnet_preprocessed'
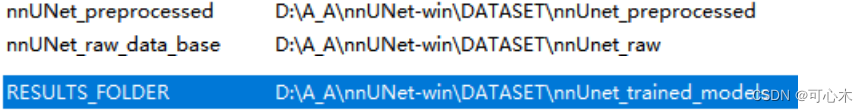
network_training_output_dir_base = r'D:\A_A\nnUNet-win\DATASET\nnUnet_trained_models'
第二步:在系统变量添加这三个路径:
添加好后是这样
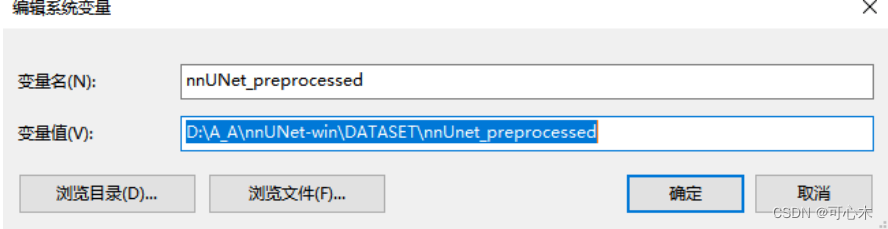
具体过程:右键“此电脑”--> 属性-->高级系统设置 -->环境变量 -->系统变量 --> 新建
然后把左边的添加为变量名, 后边的添加为变量值。添加好后,点击确定,再继续添加另两个的路径。
6 数据转换和识别
nnUNet要求将原始数据转换成特定的格式,以便了解如何读取和解释数据。
终端输入:
nnUNet_convert_decathlon_task -i /A_A/nnUNet-win/DATASET/nnUNet_raw/nnUNet_raw_data/Task01_BrainTumour
这一步完成后,会在D:\A_A\nnUNet-win\DATASET\nnUnet_raw\nnUnet_raw_data的文件夹里发现Task001_BrainTumour文件夹,打开后,里面就是这四个文件夹。
其格式与Task01_BrainTumour一模一样。
在pycharm里可以看到,它是每四个:0000、0001、0002、0003模态组成一个图像。
7 数据预处理
输入指令的格式应该如下:
nnUNet_plan_and_preprocess -t XXX --verify_dataset_integrity
因为我们的Task_id是1,所以这里XXX的就是1。因此,我这里输入的是:
nnUNet_plan_and_preprocess -t 1 --verify_dataset_integrity
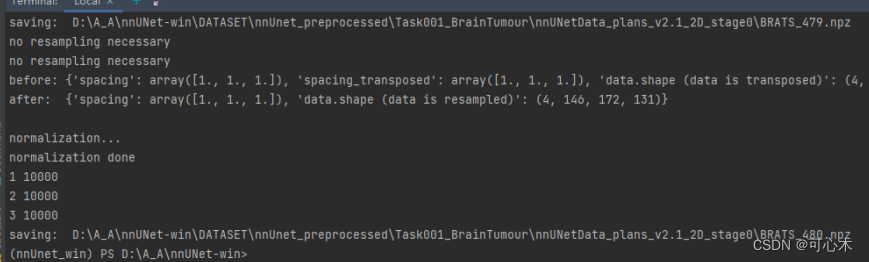
这个过程会消耗很多的时间,速度慢的原因在于对要进行插值等各种操作。
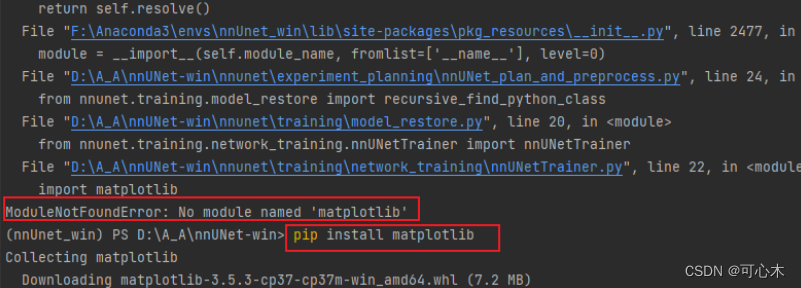
提示没有某个模块,pip install即可。
再重新运行预处理指令,需要耗费一点时间。可以看到是这样的:
最后像这样就运行好了
这一步会进行数据格式的转换,裁剪crop,重采样resample以及标准化normalization。
运行结束后,在D:\A_A\nnUNet-win\DATASET\nnUnet_raw\nnUnet_cropped_data里有Task001_BrainTumour,打开后有一些文件
这里注意:预处理的数据文件夹仅包含训练案例,测试图像没有经过预处理。
8 开始训练
如果你的显卡内存没有8g以上,就不要参考方法一,直接进入方法二下载预训练好的模型。
8.1 方法一
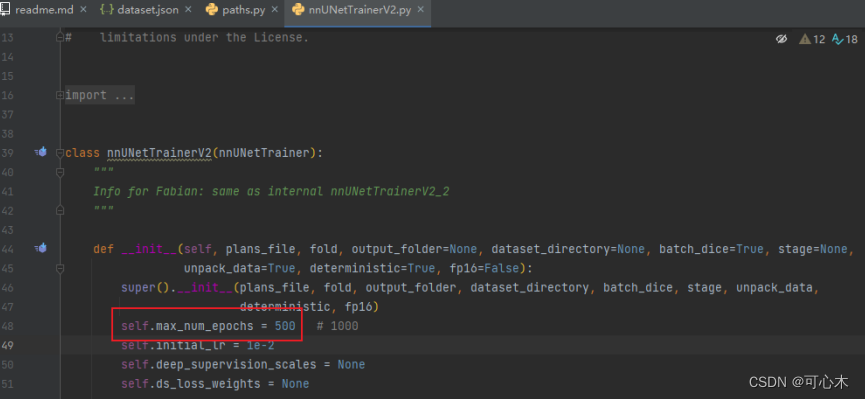
进入nnunet/training/network_training/nnUNetTrainerV2.py
修改第48行的max_epoch,1000运行时间长,可以改小一些,例如100,500等。
然后,在终端运行:
nnUNet_train 3d_fullres nnUNetTrainerV2 xx all
这里xx为数据编号,比如1。 all代表5-fold验证,可填写0-4,0代表五折交叉验证中的第0折,3d_fullres代表选用对全分辨率图像进行操作的3D U-Net,nnUNetTrainerV2是我们选用的训练器。
我这里运行的是这个命令:
nnUNet_train 3d_fullres nnUNetTrainerV2 1 4
也可以在末尾加“–npz”:
nnUNet_train 3d_fullres nnUNetTrainerV2 1 0 --npz
“–npz”使模型在最终验证期间保存softmax输出。它仅适用于计划在之后运行“nnUNet_find_best_configuration”的训练。
这就跑起来了:
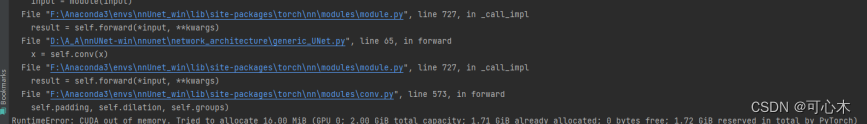
一会儿就报错了,因为我的内存不够。
如果你成功的运行出来,其结果应该保存在D:\A_A\nnUNet-win\DATASET\nnUnet_trained_models\nnUNet里面。
8.2 方法二:
进入这个网址:pretrained models for 3D semantic image segmentation with nnU-Net | Zenodo
下载预训练模型,建议《<--网上学科<--》

下载好后,解压打开是这样的:
把它们全部复制到D:\A_A\nnUNet-win\DATASET\nnUnet_trained_models\nnUNet里面去。
9 推理
进入D:\A_A\nnUNet-win\DATASET\nnUnet_raw\nnUnet_raw_data\Task001_BrainTumour
创建文件夹inferTs和labelTs。
labelsTs中存放测试集的标签,inferTs是待推理测试集的推理结果。
由于原本的imagesTs中有1000多个nii文件,会花费巨额的时间,建议你先找个地方把其他文件拷贝出来,只留4个nii.gz文件进行推理,例如留下485的所有模态
在终端中执行下面命令行:
nnUNet_predict -i /A_A/nnUNet-win/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BrainTumour/imagesTs/ -o /A_A/nnUNet-win/DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BrainTumour/inferTs -t 1 -m 3d_fullres -f 4
nnUNet_predict:执行预测的命令;
-i: 输入(你的待推理测试集);
-o: 输出(测试集的推理结果);
-t: 你的任务对应的数字ID;
-m: 对应的训练时使用的网络架构;
-f: 数字4代表使用五折交叉验证训练出的模型;
推理完全部需要消耗相当长的时间,建议使用用4个测试文件进行推理。一开始我只留了一个推理文件,结果报错,说task001需要至少4个。

若嫌弃太长,可以cd到路径下,再运行:
cd DATASET/nnUNet_raw/nnUNet_raw_data/Task001_BrainTumour/
nnUNet_predict -i imagesTs/ -o inferTs -t 1 -m 3d_fullres -f all
如果你想调试的话,在inference文件夹中找到predict_simple.py文件,然后输入参数就可以调试了。
最后推理结果在inferTs里,解压后,用itk打开可以查看结果。
但是只推理一个文件,结果不是很明显,可以多推理几个看看效果。把一个编号的4个模态:0000、0001、0002、0003都复制到imagesTs里。我后面又运行了9个文件(485-493),速度就比较慢了。大概用了几分钟就出结果了。
之后对全部数据进行推理,几小时出结果。
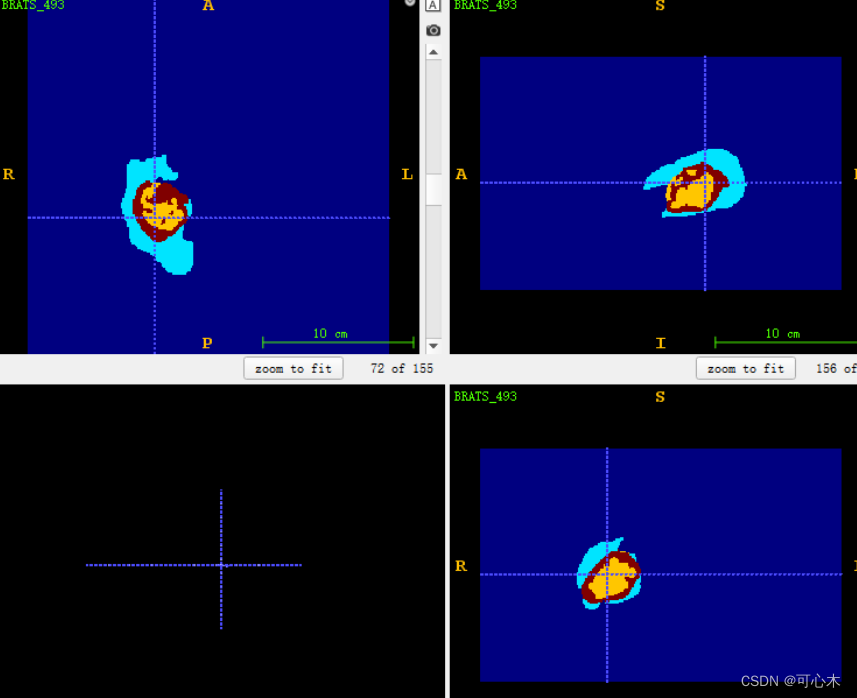
打开了493的推理结果:
参考的博文
nnUnet 在windows 10 上使用_nnunet windows_skyous的博客-CSDN博客
(四:2020.07.28)nnUNet最舒服的训练教程(让我的奶奶也会用nnUNet(上))(21.04.20更新)_nnuet_花卷汤圆的博客-CSDN博客
(五:2020.07.31)nnUNet最简单的推理教程(让我的奶奶也会用nnUNet(下))_nnunet推理_花卷汤圆的博客-CSDN博客
在windows下安装nnUnet,并制作数据集以及运行(让隔壁奶奶也能学会的教程)_花生米生花@的博客-CSDN博客nnUNet保姆级使用教程!从环境配置到训练与推理(新手必看)_nnunet推理_江江ahh的博客-CSDN博客在ubuntu/windows中安装nnUNet并训练自己的数据_nnunet安装_为了待会去码头整点薯条的博客-CSDN博客
版权归原作者 可心木 所有, 如有侵权,请联系我们删除。