前言
博主共参与了数十场数学建模,其中对于未给出标签的数据进行分析时一般第一个想到的就是聚类算法。聚类算法分析又称群分析,它是研究(样品或指标)分类问题的一种统计分析方法,同时也是数据挖掘的一个重要算法。聚类(Cluster)分析是由若干模式(Pattern)组成的,通常,模式是一个度量(Measurement)的向量,或者是多维空间中的一个点。聚类分析以相似性为基础,在一个聚类中的模式之间比不在同一聚类中的模式之间具有更多的相似性。
K-means均值聚类算法作为最经典也是最基础的无标签分类学习算法,根据不断的迭代优化衍生出许多十分好用的算法,例如K-mean++、K-MEDOIDS等。因此学习K-means的底层原理和计算方法是十分有必要。
本篇博客的愿景是希望我或者读者通过阅读这篇博客能够学会方法并能实际运用,而且能够记录到你的思想之中。希望读者看完能够提出错误或者看法,博主会长期维护博客做及时更新。

一、聚类分析
我们知道我们是使用聚类算法的目的就是从大量数据中将他们具有相关性的特征输入,然后通过算法返回标签类型。也就是说该算法的目的就是将具有相同特性的数据归纳为一类。当然我们的算法是贪心的,尽可能将所有相同类型的数据归为一类,本质还在站在分类的角度上,只不过没有标签需要我们进行运算得出。
那么既然是找到具有相同性质的数据,那么回到原始的方法,例如KNN算法,我们只需要去根据两个数据点的距离去判断他们是否属于一类,其实聚类的思想也类似,只不过我们选择圆心的点不再是判断数据类型的点,而是划分为一类标签的最大范围半径的点,类似画一个最大的圆:
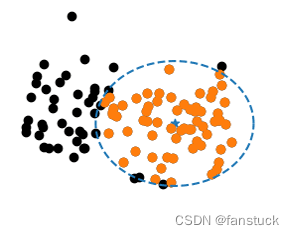
二、K-means原理
既然前面我们谈到了聚类分析也就是根据彼此的相关性来划分聚类,那么他们的相关性又以什么来衡量呢?这点和KNN算法类似,这设计到了距离度量算法。
1.距离度量算法
在不同情况维度下,我们计算两点之间的距离也不同,不过我们在初中高中学的两点之间的距离计算公式,为欧式距离:
欧几里得距离(欧氏距离)
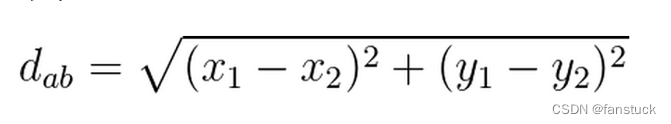
衡量多维空间中的两点间距离,也是最常用的距离度量方法。
曼哈顿距离
对于曼哈顿距离大家可能会比较陌生,这是种使用在几何度量空间的几何学用语,用以标明两个点在标准坐标系上的绝对轴距总和。
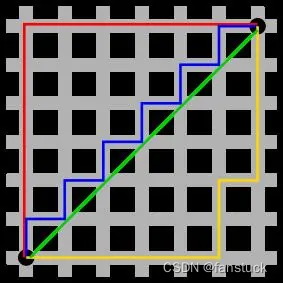
图中红线代表曼哈顿距离,绿色代表欧氏距离,也就是直线距离,而蓝色和黄色代表等价的曼哈顿距离。曼哈顿距离——两点在南北方向上的距离加上在东西方向上的距离,即:
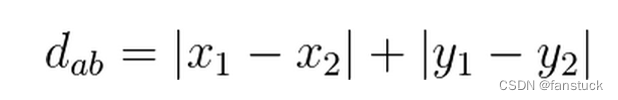
对于一个具有正南正北、正东正西方向规则布局的城镇街道,从一点到达另一点的距离正是在南北方向上旅行的距离加上在东西方向上旅行的距离,因此,曼哈顿距离又称为出租车距离。曼哈顿距离不是距离不变量,当坐标轴变动时,点间的距离就会不同。
切比雪夫距离
相信大家对于距离的了解,了解多的知道除了欧式距离以外还有个曼哈顿距离,但是除去曼哈顿距离之外还有个切比雪夫距离。在数学中,切比雪夫距离或是L∞度量,是向量空间中的一种度量,二个点之间的距离定义是其各坐标数值差绝对值的最大值。

国际象棋棋盘上二个位置间的切比雪夫距离是指王要从一个位子移至另一个位子需要走的步数。由于王可以往斜前或斜后方向移动一格,因此可以较有效率的到达目的的格子。图是棋盘上所有位置距f6位置的切比雪夫距离。一维空间中,所有的Lp度量都是一样的,即为二座标差的绝对值。
平面上两点的切比雪夫距离为:
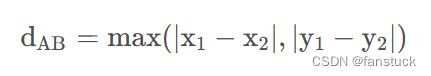
n维空间上的切比雪夫距离:
n 维空间则有两点
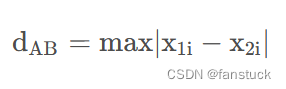
2.K-means算法思想
K-means聚类算法思想可以看它设计诞生的伪代码看出:

我们发现这是一种迭代求解的聚类分析算法,其步骤是,预将数据分为K组,则随机选取K个对象作为初始的聚类中心,然后计算每个对象与各个种子聚类中心之间的距离,把每个对象分配给距离它最近的聚类中心。聚类中心以及分配给它们的对象就代表一个聚类。每分配一个样本,聚类的聚类中心会根据聚类中现有的对象被重新计算。这个过程将不断重复直到满足某个终止条件。终止条件可以是没有(或最小数目)对象被重新分配给不同的聚类,没有(或最小数目)聚类中心再发生变化,误差平方和局部最小。
三.K-means算法实现
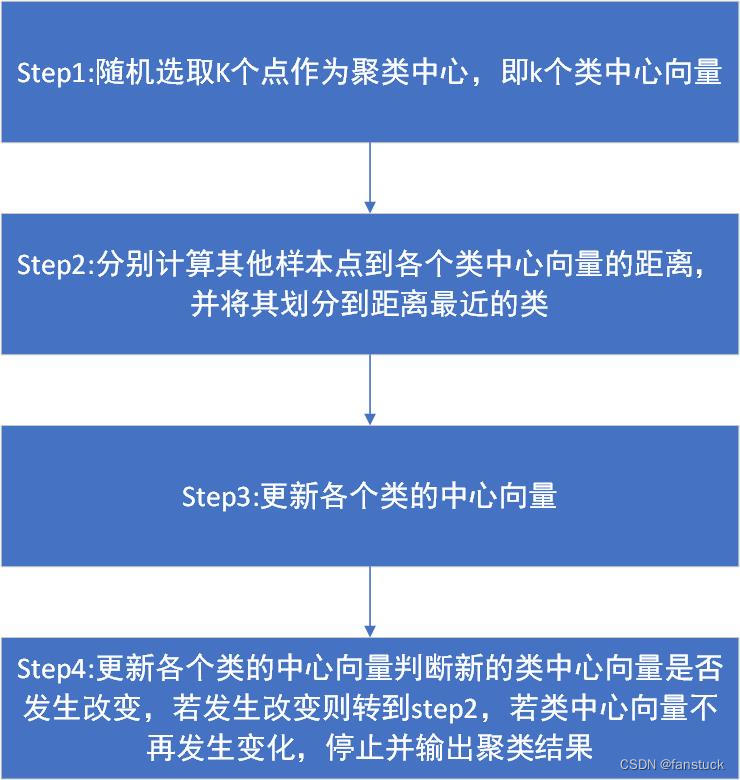
我们将算法步骤细化来分析:
step1:选取K值
k 的选择一般是按照实际需求进行决定,或在实现算法时直接给定 k 值。这是基于项目你想要聚类的个数来决定的,但是也有不确定的情况,我们可能需要去一个最优的K值来将数据很好的归类达到最大化区分类别,这时候就需要思考从数据角度出发,应该进行怎么样的计算能够得到最优的K。
1.手肘法
手肘法是最常用的确定K-means算法K值的方法,所用到的衡量标准是SSE(sum of the squared errors,误差平方和) 。
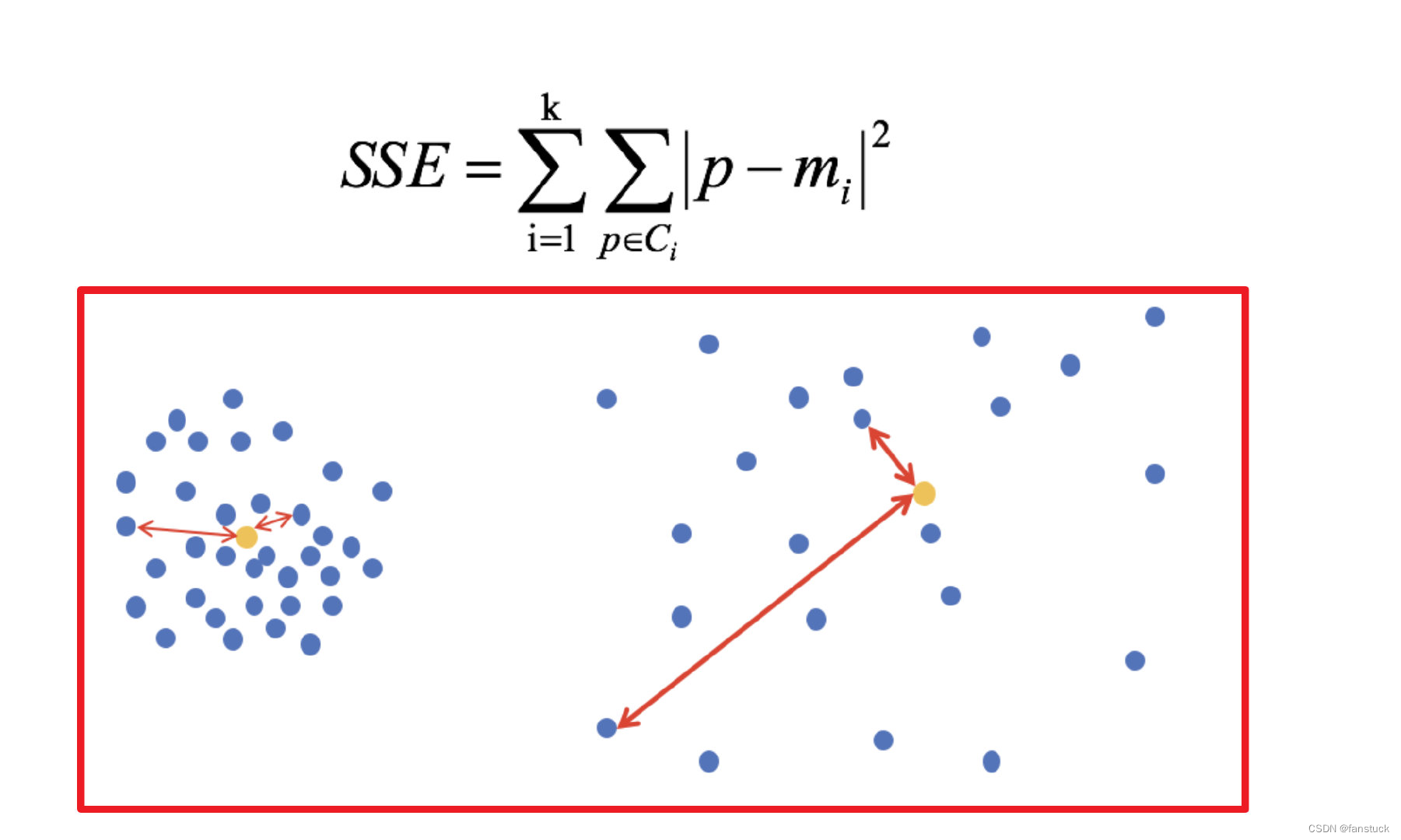
SEE各个计算在K-means里含义如下图:
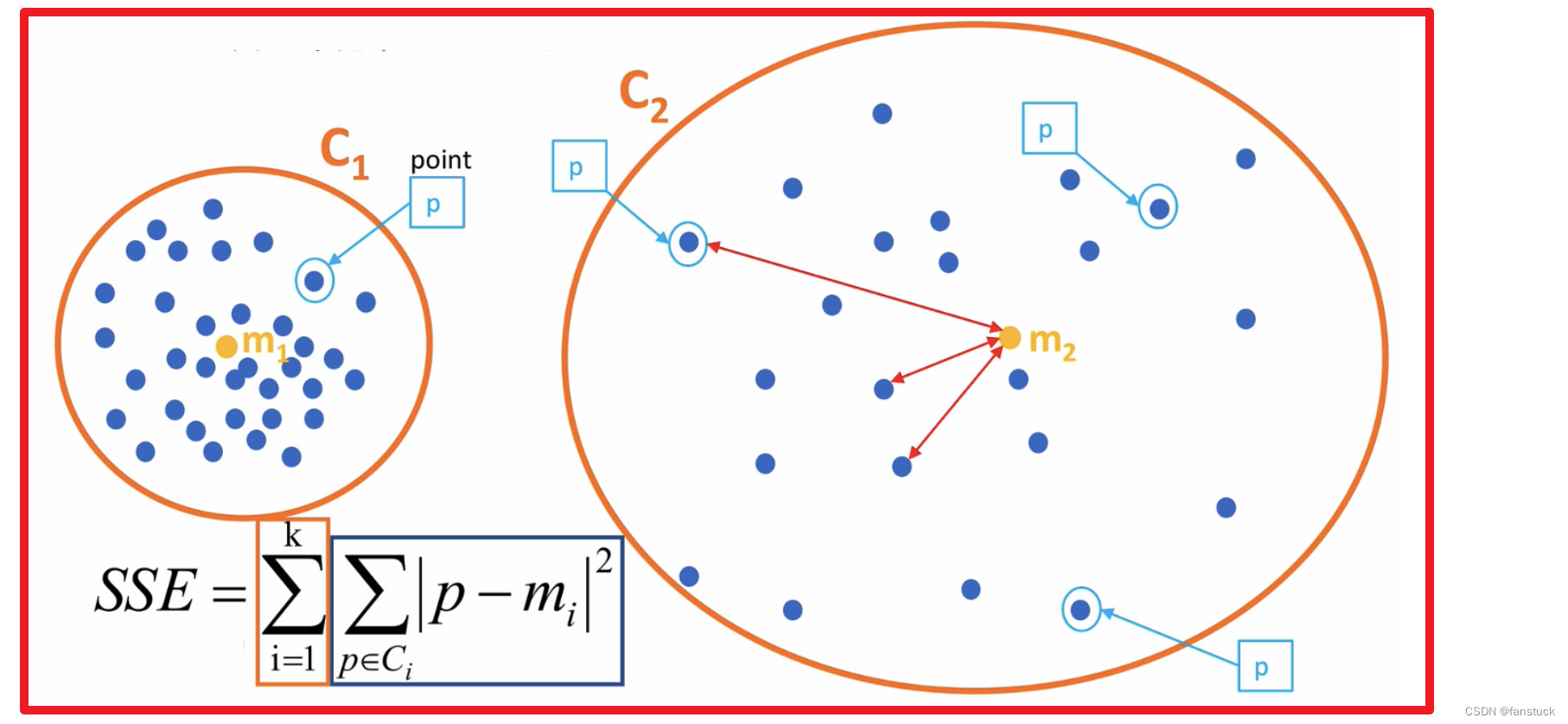
误差平方和又称残差平方和,根据n个观察值拟合适当的模型后,余下未能拟合部份()称为残差,其中y平均表示n个观察值的平均值,所有n个残差平方之和称误差平方和。
残差我在最小二乘法已经做了详细解释,想要了解的可以去看我这篇文章:
一文速学-最小二乘法曲线拟合算法详解+项目代码
在回归分析中通常用SSE表示,其大小用来表明函数拟合的好坏。将残差平方和除以自由度n-p-1(其中p为自变量个数)可以作为误差方差σ2的无偏估计,通常用来检验拟合的模型是否显著也用来寻找K值。
主要思想:当k小于真实聚类数时,随着k的增大,会大幅提高类间聚合程度,SSE会大幅下降,当k达到真实聚类数时,随着k的增加,类间的聚合程度不会大幅提高,SSE的下降幅度也不会很大,所以k/SSE的折线图看起来像一个手肘,我们选取肘部的k值进行运算。
这里我们以欧几里德距离来计算两点之间的相关性:
python代码:
import matplotlib.pyplot as plt
import pandas as pd
from sklearn.metrics import silhouette_score
from sklearn.cluster import KMeans
data=pd.read_csv(r'C:\Users\10799\get_info\sklearn_try\series_gpstime_level.csv')
distortions=[]#簇内误差平方和 SSE
for i in range(2,10):
Kmeans_model=KMeans(n_clusters=i)
predict_=Kmeans_model.fit_predict(data)
distortions.append(Kmeans_model.inertia_)
print("簇内误差平方和:",distortions)
#SSE 手肘法
plt.plot(range(2,10),distortions,marker='x')
plt.xlabel('Number of clusters')
plt.ylabel('Distortion')
plt.title('distortions')
plt.show()
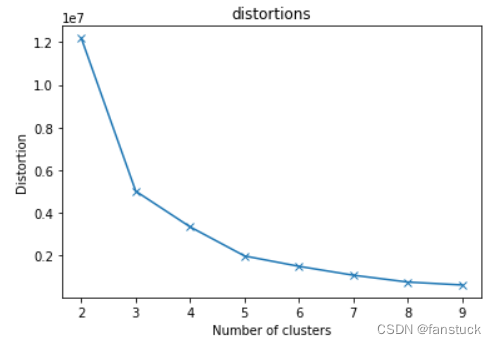
根据拟合图片我们知道选K为5时能够得到最效率的K值。
2.轮廓系数法
轮廓系数这一指标无需知道数据集的真实标签。取值范围[-1, 1],值越大,聚类效果越好。旨在将某个对象与自己的簇的相似程度和与其他簇的相似程度作比较。轮廓系数最高的簇的数量表示簇的数量的最佳选择。
轮廓系数综合考虑了内聚度和分离度两种因素。
方法: 轮廓系数公式:
1)计算样本i到同簇其他样本的平均距离a(i)。a(i) 越小,说明样本i越应该被聚类到该簇。将a(i)称为样本i的簇内不相似度。
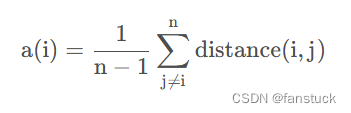
最近簇定义如下:
2)计算样本i到其他某簇Cj的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, ..., bik},bi越大,说明样本i越不属于其他簇。
- 轮廓系数范围在[-1,1]之间。该值越大,越合理。
- si接近1,则说明样本i聚类合理;
- si接近-1,则说明样本i更应该分类到另外的簇;
- 若si 近似为0,则说明样本i在两个簇的边界上。
- 有样本的s i 的均值称为聚类结果的轮廓系数,是该聚类是否合理、有效的度量。
3)根据样本i的簇内不相似度a(i)和簇间不相似度b(i) ,定义样本i的轮廓系数.
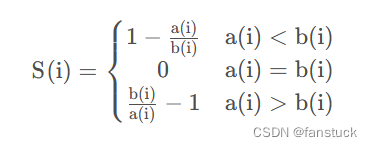
求出所有点的轮廓系数再求平均值就得出了平均轮廓系数,平均轮廓系数的取值在[-1,1],显然,由轮廓系数公式可以观察出,凝聚度越小,分离度越大,分类效果越好,平均轮廓系数也越大,所以,取平均轮廓系数最大的点的k值时,分类效果越好.
python代码:
scores=[] #存放轮廓系数
for i in range(2,10):
Kmeans_model=KMeans(n_clusters=i)
predict_=Kmeans_model.fit_predict(data)
scores.append( silhouette_score(data,predict_))
print("轮廓系数:",scores)
#轮廓系数法
plt.plot(range(2,10),scores,marker='x')
plt.xlabel('Number of clusters')
plt.ylabel('scores')
plt.title('scores')
plt.show()
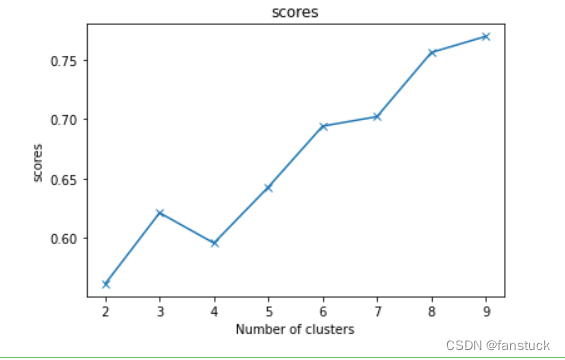
step2:计算初始化K点
初始质心随机选择即可,每一个质心为一个类。对剩余的每个样本点,计算它们到各个质心的欧式距离,并将其归入到相互间距离最小的质心所在的簇。
def euclDistance(x1, x2):
return np.sqrt(sum((x2 - x1) ** 2))
def initCentroids(data, k):
numSamples, dim = data.shape
# k个质心,列数跟样本的列数一样
centroids = np.zeros((k, dim))
# 随机选出k个质心
for i in range(k):
# 随机选取一个样本的索引
index = int(np.random.uniform(0, numSamples))
# 作为初始化的质心
centroids[i, :] = data[index, :]
return centroids
step3:迭代计算重新划分
- 计算各个新簇的质心。
- 在所有样本点都划分完毕后,根据划分情况重新计算各个簇的质心所在位置,然后迭代计算各个样本点到各簇质心的距离,对所有样本点重新进行划分
- 重复2. 和 3.,直到质心不在发生变化时或者到达最大迭代次数时
# 传入数据集和k值
def kmeans(data, k):
# 计算样本个数
numSamples = data.shape[0]
# 样本的属性,第一列保存该样本属于哪个簇,第二列保存该样本跟它所属簇的误差
clusterData = np.array(np.zeros((numSamples, 2)))
# 决定质心是否要改变的质量
clusterChanged = True
# 初始化质心
centroids = initCentroids(data, k)
while clusterChanged:
clusterChanged = False
# 循环每一个样本
for i in range(numSamples):
# 最小距离
minDist = 100000.0
# 定义样本所属的簇
minIndex = 0
# 循环计算每一个质心与该样本的距离
for j in range(k):
# 循环每一个质心和样本,计算距离
distance = euclDistance(centroids[j, :], data[i, :])
# 如果计算的距离小于最小距离,则更新最小距离
if distance < minDist:
minDist = distance
# 更新最小距离
clusterData[i, 1] = minDist
# 更新样本所属的簇
minIndex = j
# 如果样本的所属的簇发生了变化
if clusterData[i, 0] != minIndex:
# 质心要重新计算
clusterChanged = True
# 更新样本的簇
clusterData[i, 0] = minIndex
# 更新质心
for j in range(k):
# 获取第j个簇所有的样本所在的索引
cluster_index = np.nonzero(clusterData[:, 0] == j)
# 第j个簇所有的样本点
pointsInCluster = data[cluster_index]
# 计算质心
centroids[j, :] = np.mean(pointsInCluster, axis=0)
return centroids, clusterData
step4:可视化展现
def showCluster(data, k, centroids, clusterData):
numSamples, dim = data.shape
if dim != 2:
print('dimension of your data is not 2!')
return 1
# 用不同颜色形状来表示各个类别
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'dr', '<r', 'pr']
if k > len(mark):
print('your k is too large!')
return 1
# 画样本点
for i in range(numSamples):
markIndex = int(clusterData[i, 0])
plt.plot(data[i, 0], data[i, 1], mark[markIndex])
# 用不同颜色形状来表示各个类别
mark = ['*r', '*b', '*g', '*k', '^b', '+b', 'sb', 'db', '<b', 'pb']
# 画质心点
for i in range(k):
plt.plot(centroids[i, 0], centroids[i, 1], mark[i], markersize=20)
plt.show()
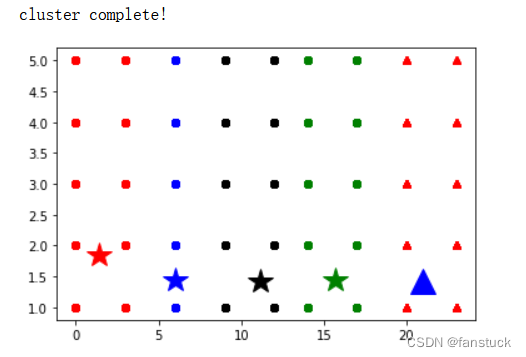
最后结果根据 手肘法我们选取k为5:
k = 5
centroids, clusterData = kmeans(data, k)
if np.isnan(centroids).any():
print('Error')
else:
print('cluster complete!')
# 显示结果
showCluster(data, k, centroids, clusterData)
四、K-means优缺点
优点:
- k‐均值算法原理简单,容易实现,且运行效率比较高
- k‐均值算法聚类结果容易解释,适用于高维数据的聚类
缺点:
- k‐均值算法采用贪心策略,导致容易局部收敛,在大规模数据集上求解较慢
- k‐均值算法对离群点和噪声点非常敏感,少量的离群点和噪声点可能对算法求平均值产生极大影响,从而影响聚类结果
点关注,防走丢,如有纰漏之处,请留言指教,非常感谢
以上就是本期全部内容。我是fanstuck ,有问题大家随时留言讨论 ,我们下期见。
参阅:
曼哈顿距离
数据科学中常见的9种距离度量方法,内含欧氏距离、切比雪夫距离等
K均值聚类算法
K-means算法(知识点梳理)
误差平方和
K-means算法代码实现(python)
手撕K-means聚类算法
版权归原作者 fanstuck 所有, 如有侵权,请联系我们删除。