入门小菜鸟,希望像做笔记记录自己学的东西,也希望能帮助到同样入门的人,更希望大佬们帮忙纠错啦~侵权立删。
可参照以下博客一起看(涉及一些概念解析)深度学习之常用模型评估指标(一)—— 分类问题和目标检测_tt丫的博客-CSDN博客_深度网络模型特异度 yolov5训练后会产生runs文件夹,其中的train文件夹中的exp文件夹里存放的即是训练后模型的各种信息,里面的weights文件夹即放置模型权重参数,其余的文件即为各种性能指标信息。
一、confusion_matrix.png —— 混淆矩阵
混淆矩阵是对分类问题预测结果的总结。使用计数值汇总正确和不正确预测的数量,并按每个类进行细分,显示了分类模型进行预测时会对哪一部分产生混淆。通过这个矩阵可以方便地看出机器是否将两个不同的类混淆了(把一个类错认成了另一个)。
混淆矩阵不仅可以让我们直观的了解分类模型所犯的错误,更重要的是可以了解哪些错误类型正在发生,正是这种对结果的分解克服了仅使用分类准确率带来的局限性(总体到细分)。
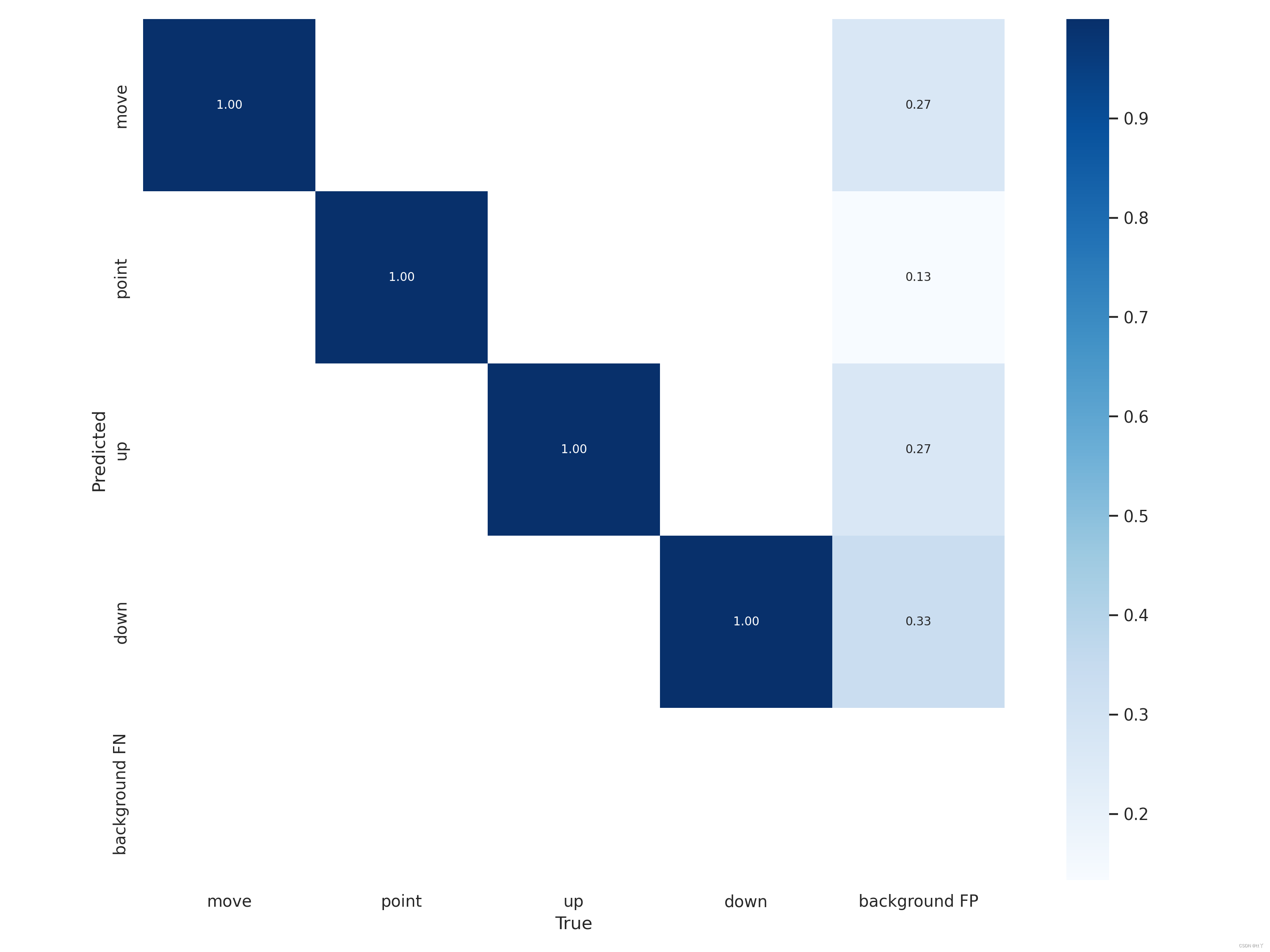
行是预测类别(y轴),列是真实类别(x轴);
矩阵中Aij的含义是:第j个类别被预测为第i个类别的概率;
从图中可分析到:每一类被正确分类的概率为100%(emmm可能过拟合了),但是背景被误分还是有一定的概率,尤其是“down”类。
二、F1_curve.png —— F1曲线
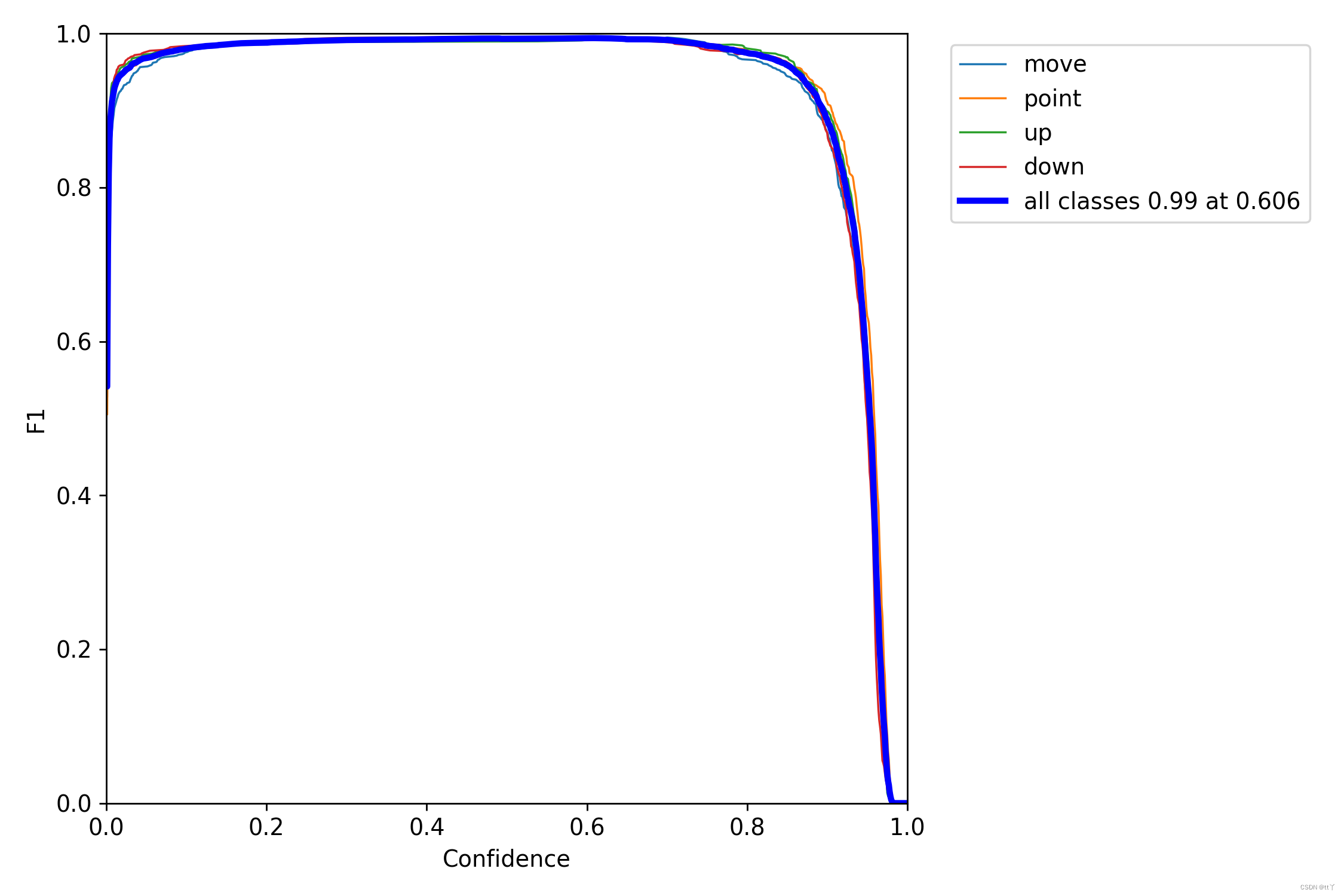
F1分数与置信度阈值(x轴)之间的关系。F1分数是分类的一个衡量标准,是精确率和召回率的调和平均数,介于0,1之间。越大越好。
一般来说,置信度阈值(该样本被判定为某一类的概率阈值)较低的时候,很多置信度低的样本被认为是真,召回率高,精确率低;置信度阈值较高的时候,置信度高的样本才能被认为是真,类别检测的越准确,即精准率较大(只有confidence很大,才被判断是某一类别),所以前后两头的F1分数比较少。
可以看到F1曲线很“宽敞”且顶部接近1,说明在训练数据集上表现得很好(既能很好地查全,也能很好地查准)的置信度阈值区间很大。
三、labels.jpg —— 标签
第一个图是训练集得数据量,每个类别有多少个;
第二个图是框的尺寸和数量;
第三个图是中心点相对于整幅图的位置;
第四个图是图中目标相对于整幅图的高宽比例;
四、labels_correlogram.jpg —— 体现中心点横纵坐标以及框的高宽间的关系
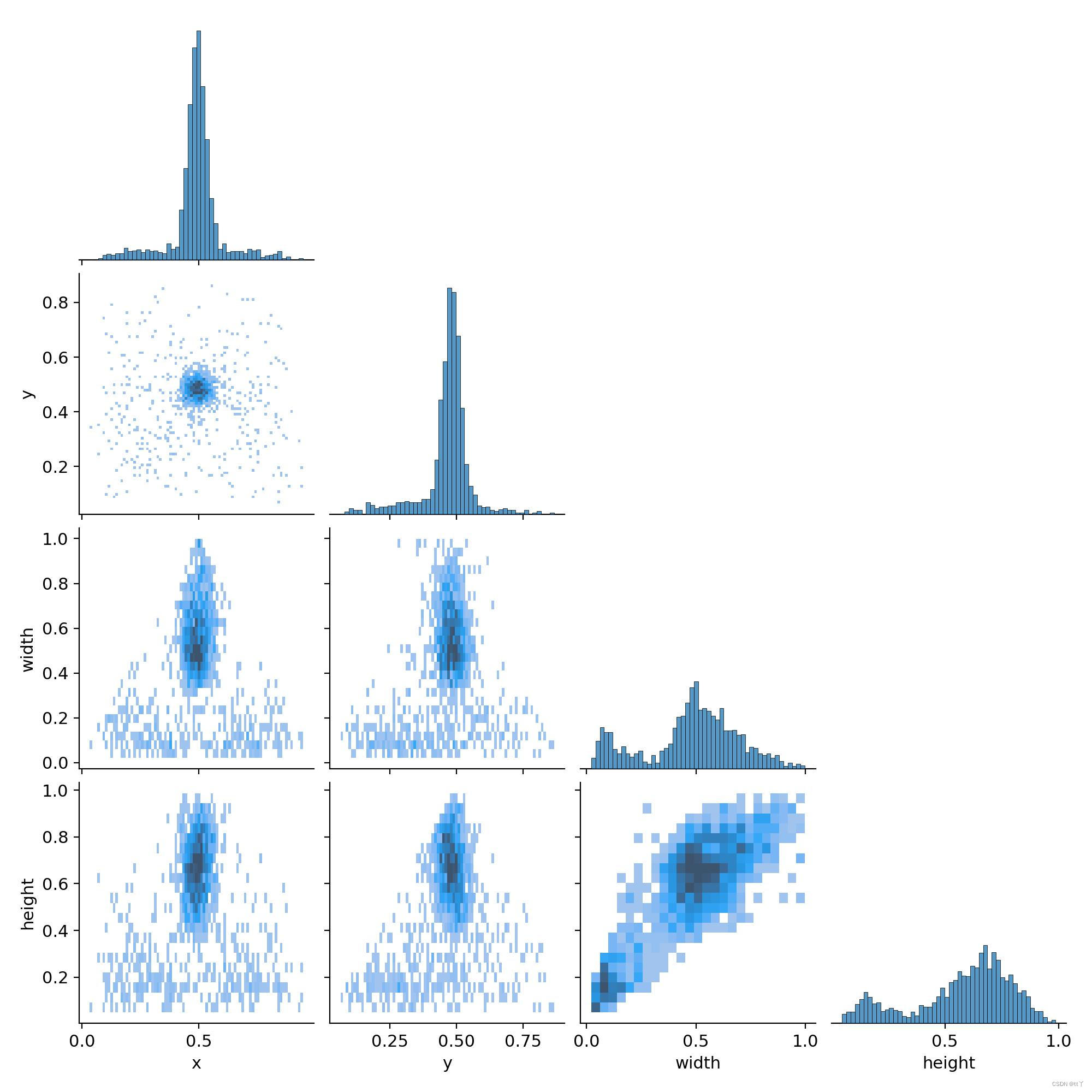
表示中心点坐标x和y,以及框的高宽间的关系。
每一行的最后一幅图代表的是x,y,宽和高的分布情况:
最上面的图(0,0)表明中心点横坐标x的分布情况,可以看到大部分集中在整幅图的中心位置;
(1,1)图表明中心点纵坐标y的分布情况,可以看到大部分集中在整幅图的中心位置;
(2,2)图表明框的宽的分布情况,可以看到大部分框的宽的大小大概是整幅图的宽的一半;
(3,3)图表明框的宽的分布情况,可以看到大部分框的高的大小超过整幅图的高的一半
而其他的图即是寻找这4个变量间的关系
五、P_curve.png —— 单一类准确率
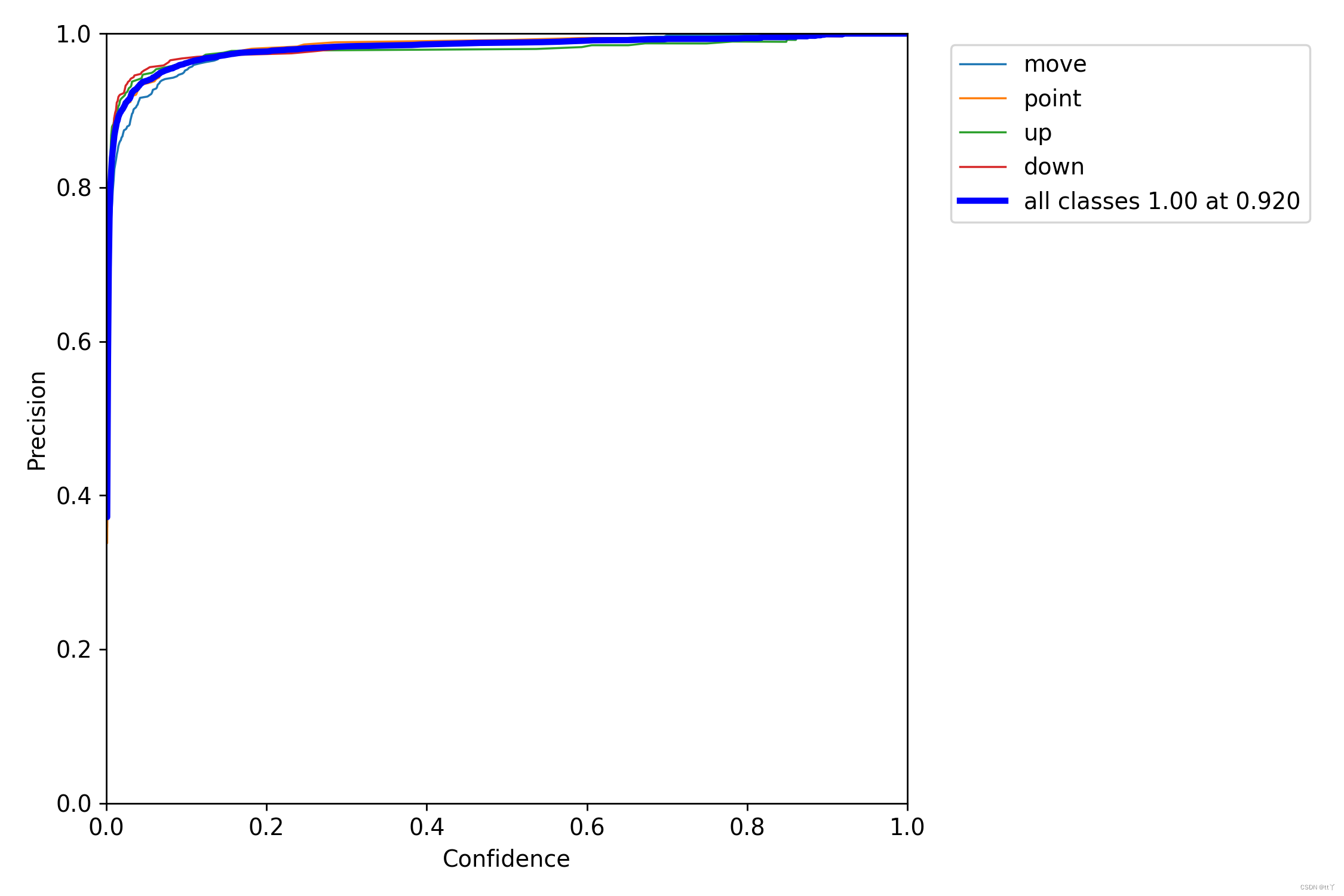
即置信度阈值 - 准确率曲线图
当判定概率超过置信度阈值时,各个类别识别的准确率。当置信度越大时,类别检测越准确,但是这样就有可能漏掉一些判定概率较低的真实样本。
六、R_curve.png —— 单一类召回率
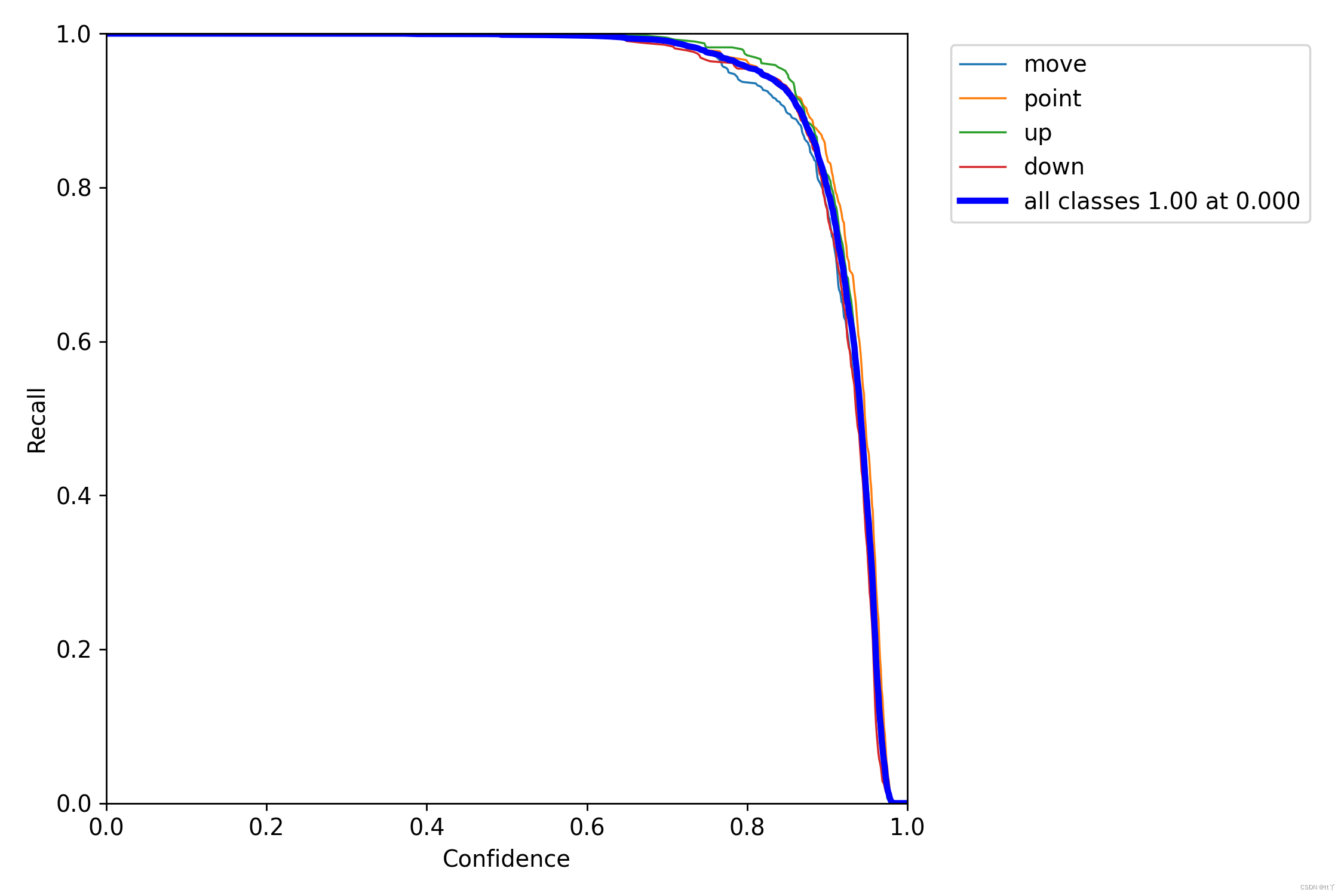
即置信度阈值 - 召回率曲线图
当置信度越小的时候,类别检测的越全面(不容易被漏掉,但容易误判)。
七、PR_curve.png —— 精确率和召回率的关系图
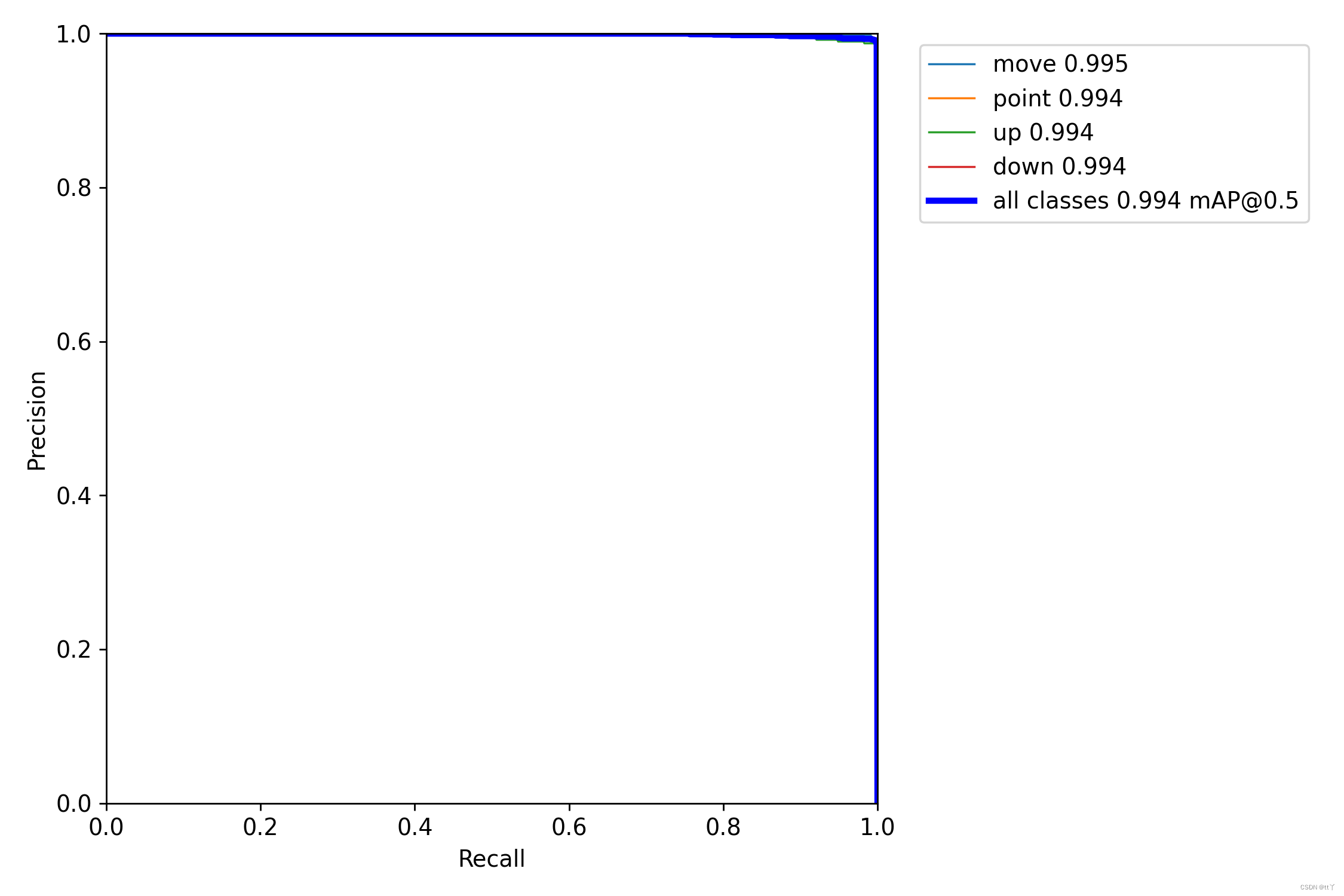
PR曲线体现精确率和召回率的关系。
mAP 是 Mean Average Precision 的缩写,即 均值平均精度。可以看到:精度越高,召回率越低。
我们希望:在准确率很高的前提下,尽可能的检测到全部的类别。因此希望我们的曲线接近(1,1),即希望mAP曲线的面积尽可能接近1。
八、result.png —— 结果loss functions
🌳定位损失box_loss:
预测框与标定框之间的误差(CIoU),越小定位得越准;🌳置信度损失obj_loss:
计算网络的置信度,越小判定为目标的能力越准;🌳分类损失cls_loss:
计算锚框与对应的标定分类是否正确,越小分类得越准;🌳mAP@0.5:0.95(mAP@[0.5:0.95])
表示在不同IoU阈值(从0.5到0.95,步长0.05)(0.5、0.55、0.6、0.65、0.7、0.75、0.8、0.85、0.9、0.95)上的平均mAP;🌳mAP@0.5:
表示阈值大于0.5的平均mAP
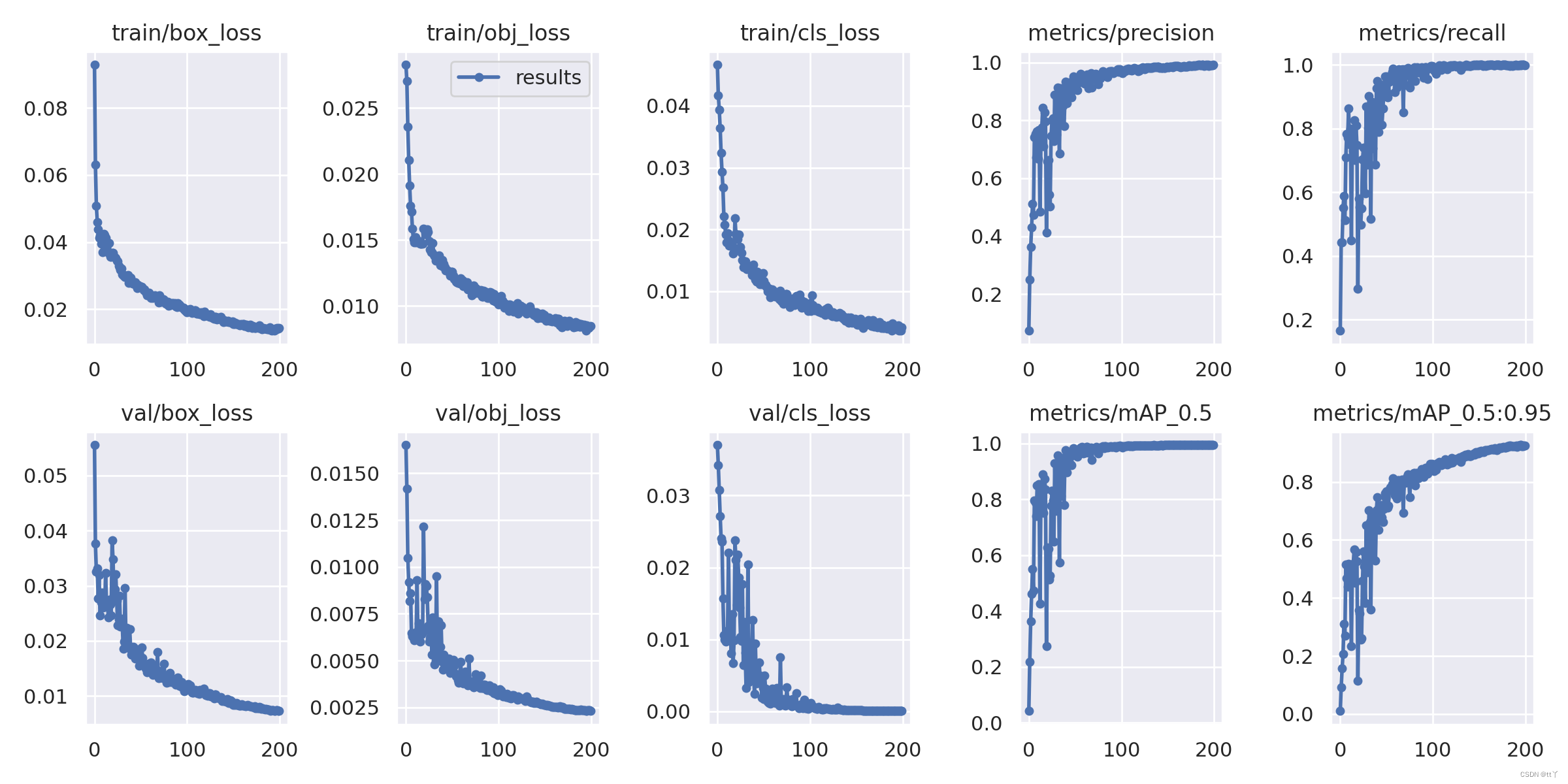
横坐标代表的是训练轮数(epoch)
一般训练结果主要观察“mAP曲线的面积大小”(PR曲线)与“精确率和召回率波动情况”(上图的右边);
PR曲线面积越接近1越好;
精确率和召回率波动不是很大则训练效果较好;如果训练比较好的话图上呈现的是稳步上升。
欢迎大家在评论区批评指正,谢谢啦~
版权归原作者 tt丫 所有, 如有侵权,请联系我们删除。