项目场景:
从github上下载CLAM代码,上传Camelyon-16中的部分WSI图像,将代码跑通。
CLAM项目地址:
GitHub - mahmoodlab/CLAM: Data-efficient and weakly supervised computational pathology on whole slide images - Nature Biomedical EngineeringData-efficient and weakly supervised computational pathology on whole slide images - Nature Biomedical Engineering - GitHub - mahmoodlab/CLAM: Data-efficient and weakly supervised computational pathology on whole slide images - Nature Biomedical Engineeringhttps://github.com/mahmoodlab/CLAM
流程:
环境配置
按如下代码进行配置,参考链接:https://github.com/mahmoodlab/CLAM/blob/master/docs/INSTALLATION.md
// (1)安装openslide-tools
sudo apt-get install openslide-tools
// (2)使用项目已经写好的库依赖创建环境
conda env create -n clam -f docs/clam.yaml
// (3)由于topk库必须使用github安装,因而先退出CLAM文件夹克隆topk库,并进行安装
git clone https://github.com/oval-group/smooth-topk.git
cd smooth-topk
python setup.py install
** 但是由于我的CUDA版本为11.4,clam.yaml中安装的pytorch版本过低会导致与CUDA版本不匹配的问题。**前一篇博文已解决此问题,参考链接
关于pytorch与CUDA版本匹配问题_DragonJ__的博客-CSDN博客
下载数据集:
CLAM所用需要使用WSI图像,本人所用数据集为Camelyon16,下载地址
Data - Grand Challenge (grand-challenge.org)
在下载后数据集按如何方式组织在项目主文件夹下建立一个Original_Image文件夹,并把所下载数据集放到该文件夹下,如下:
将WSI图像切为patch
数据集调整好后,为方便模型的训练需要对WSI进行切片,并存储相应的坐标,运行如下代码:
// Original_Image文件夹下存储原始WSI图像
// Split Image下存储切片后的图像坐标相关信息
python create_patches_fp.py --source Original_Image --save_dir Split_Image --patch_size 256 --seg --patch --stitch
切割完成后会在主目录下得到Split_Image文件夹,并按如下方式进行组织:

特征提取
CLAM首先使用预训练的ResNet50提取图像特征,执行如下代码:
// 其中process_list.csv为切割patch自动生成的process_list_autogen.csv去掉文件名的后缀得到的
// Extracted_feature 为提取后的特征的存储路径
CUDA_VISIBLE_DEVICES=0,1 python extract_features_fp.py --data_h5_dir Split_Image/ --data_slide_dir Original_Image/ --csv_path Split_Image/process_list.csv --feat_dir Extracted_feature/ --batch_size 512 --slide_ext .tif
在执行特征提取后,特征会提取到Extracted_Feature文件夹下,以.pt文件格式存储,Extracted_Feature文件夹下目录如下:

数据集划分
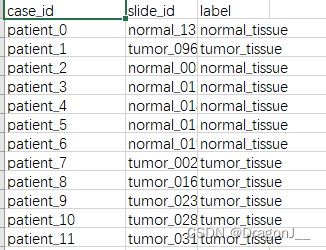
在训练开始前需要将数据集划分为训练集、验证集、测试集。这里是按照csv文件进行划分,共需要两个调整。首先在dataset_csv文件夹下创建一个test.csv文件,包含所有的样例,内部内容如下:
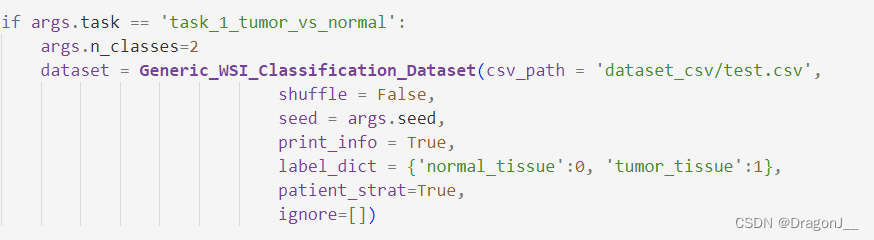
其次更改create_splits_seq代码将csv文件路径更改到test.csv,更改部分如下:
最后执行如下代码:
// 训练集占75%,共划分10次,类似10折交叉验证,每次划分均随机且不同。
python create_splits_seq.py --task task_1_tumor_vs_normal --seed 1 --label_frac 0.75 --k 10
划分后会在splits/task_1_tumor_vs_normal文件夹下产生10次划分的结果如下:
模型训练:
模型训练需要更改main.py中csv文件路径,如下:

随后输入如下代码即可训练:
CUDA_VISIBLE_DEVICES=0 python main.py --drop_out --early_stopping --lr 2e-4 --k 10 --label_frac 0.75 --exp_code task_1_tumor_vs_normal_CLAM_75 --weighted_sample --bag_loss ce --inst_loss svm --task task_1_tumor_vs_normal --model_type clam_sb --log_data --data_root_dir Extracted_feature
版权归原作者 DragonJ__ 所有, 如有侵权,请联系我们删除。