医学成像数据与其他我们日常图像的最大区别之一是它们很多都是3D的,比如在处理DICOM系列数据时尤其如此。DICOM图像由很多的2D切片组成了一个扫描或身体的特定部分。
那么如何为这类数据构建深度学习解决方案呢?本文中将介绍6种神经网络架构,可以使用它们来训练3D医疗数据上的深度学习模型。
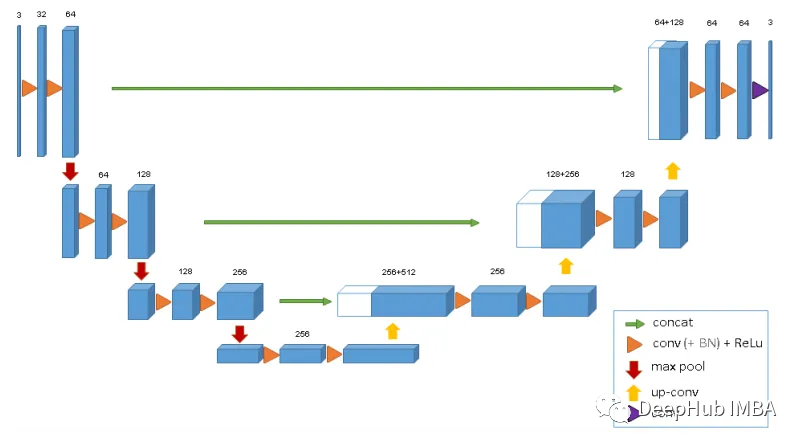
3 d U-Net
U-Net体系结构是一种强大的医学图像分割模型。3D U-Net将经典的U-Net模型扩展到3D分割。它由编码(下采样)路径和解码(上采样)路径组成。
编码路径捕获输入图像中的上下文,而解码路径允许精确定位。3D U-Net在处理体积图像的3D特性方面非常有效。
V-Net
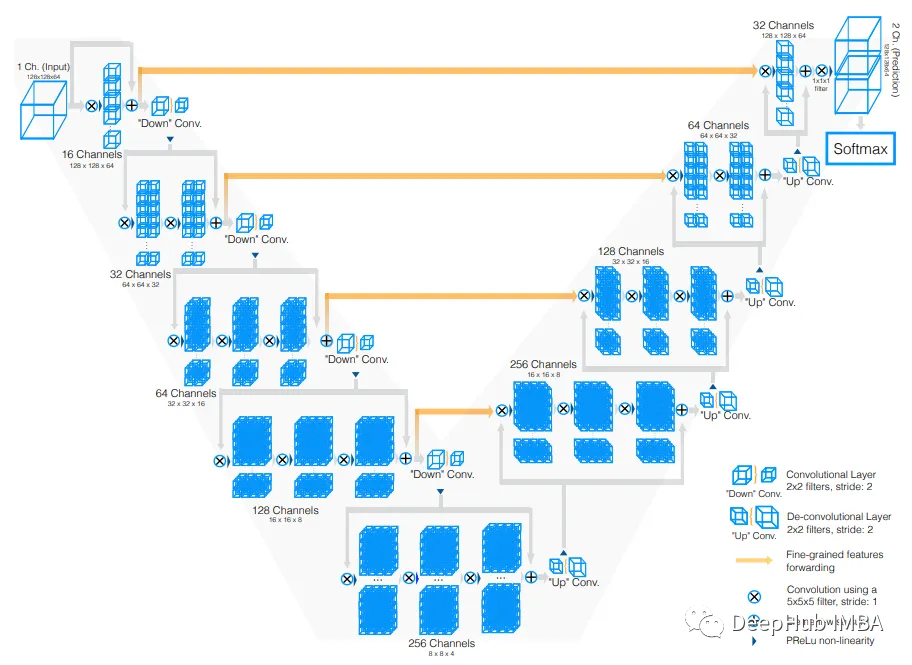
V-Net架构是另一种用于体积图像分割的3D卷积神经网络。与U-Net类似,V-Net有一个编码器-解码器架构,但它使用全分辨率3D卷积,所以它比U-Net计算成本更高。
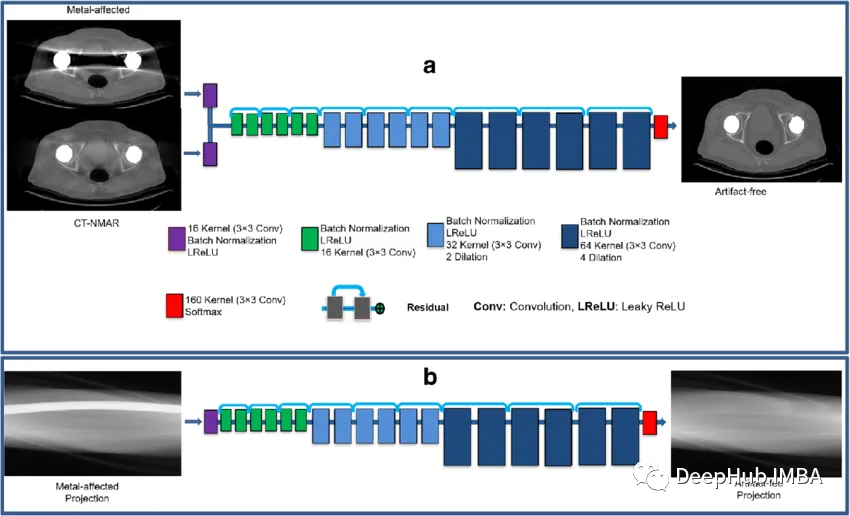
HighResNet
它使用一系列带有残差连接的3D卷积层。该模型是端到端训练的,可以一次处理整个3D图像。
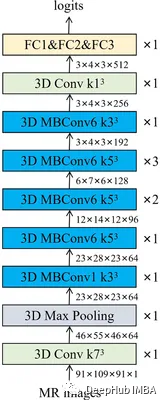
EfficientNet3D
这是对EfficientNet架构的3D改进,它不像U-Net或V-Net那样常用于3D分割,但如果计算资源有限,它是可以考虑的,因为它在计算成本和性能之间的良好权衡。
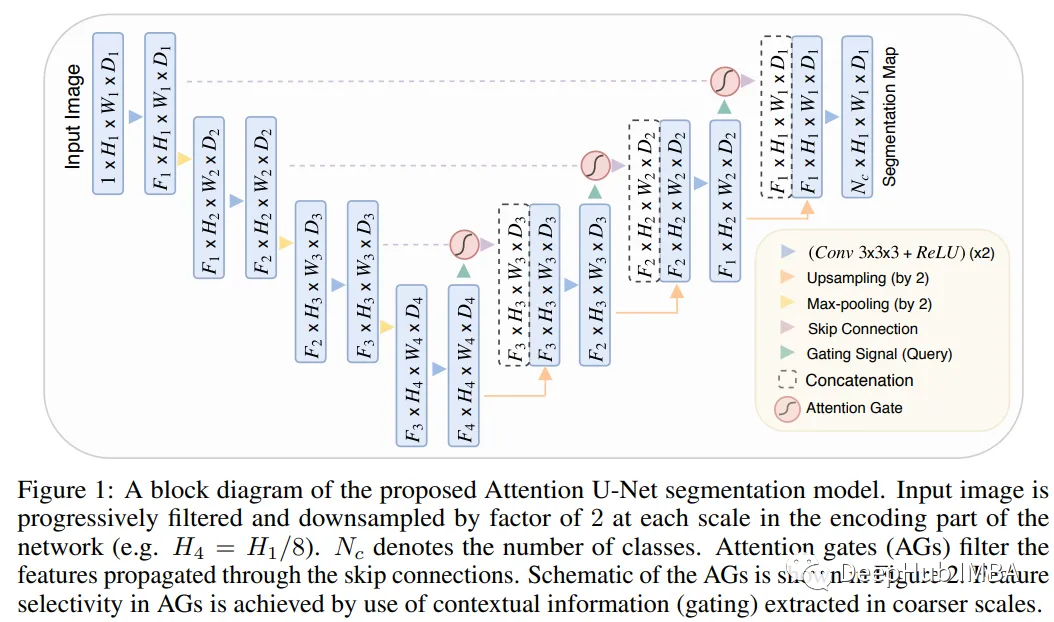
Attention U-Net
这是U-Net的一种变体,它包含了一个注意力机制,允许网络将注意力集中在与手头任务更相关的图像的某些部分。
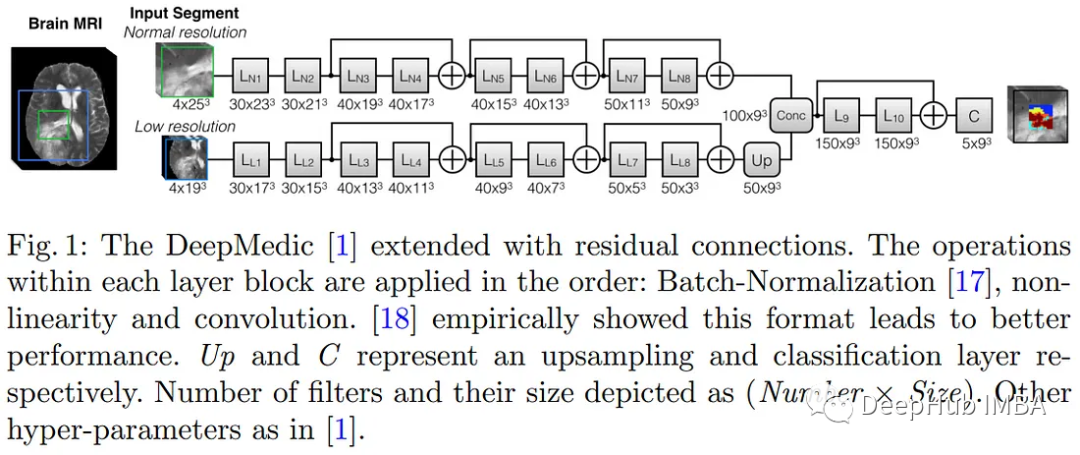
DeepMedic
这是一个使用双路径的3D CNN,一个是正常分辨率,另一个是下采样输入,这样可以结合局部和更大的上下文信息。
总结
本文中,我们介绍了医学成像行业在处理3D MRI和CT扫描时使用的一些深度学习模型。这些神经网络被设计用来接收3D数据作为输入,以学习DICOM系列身体特定部位的复杂性。
以下是本文所有模型设计的论文,如果你感兴趣,可以直接查看论文了解更详细的信息
[1] 3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
https://arxiv.org/pdf/1606.06650.pdf
https://github.com/wolny/pytorch-3dunet
[2] V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation
https://arxiv.org/pdf/1606.04797.pdf
https://github.com/Flaick/VNet
[3] Deep learning-based metal artifact reduction in PET/CT imaging
https://pubmed.ncbi.nlm.nih.gov/33569626/
https://github.com/ServiceNow/HighRes-net
[4] A modified 3D EfficientNet for the classification of Alzheimer’s disease using structural magnetic resonance images
https://github.com/shijianjian/EfficientNet-PyTorch-3D
[5] Attention U-Net: Learning Where to Look for the Pancreas
https://arxiv.org/pdf/1804.03999.pdf
https://github.com/LeeJunHyun/Image_Segmentation
[6] DeepMedic for Brain Tumor Segmentation
https://www.microsoft.com/en-us/research/wp-content/uploads/2017/02/kamnitsas2016brats.pdf
https://github.com/deepmedic/deepmedic
作者:Nour Islam Mokhtari